практикум №6
GO, PANTHER, STRING
Базы данных функций белков.
Анализируемые белки
В предложенном для анализа списке генов человека, кодирующих ферменты, содержится 7 идентификаторов. Небольшой размер списка позволил подробно изучить информацию о каждом ферменте «вручную/глазами» на NCBI Gene (табл. 1). Общей характеристикой этих ферментов является участие в метаболизме фосфолипидов.
| ID гена | Название | Локализация кодируемого белка | Функция кодируемого белка |
| GDPD1 | Глицерофосфодиэфирная фосфодиэстераза, содержащая домен 1 | Цитоплазма | Гидролиз деацилированных глицерофосфолипидов до глицерофосфата и спирта. |
| GDE1 | Глицерофосфодиэстераза 1 | Мембрана | Предполагается, что участвует в метаболизме N-ацилэтаноламинов, метаболизме этаноламинов и метаболизме фосфолипидов. |
| GDPD3 | Глицерофосфодиэстераза, содержащая домен 3 | Эндоплазматический ретикулум | Обеспечивает активность фосфодиэстеразы. Участвует в метаболизме N-ацилэтиламина. |
| PNPLA6 | Пататиноподобный фосфолипазный доменсодержащий белок 6 | Эндоплазматический ретикулум | Деацетилирует внутриклеточный фосфатидилхолин с образованием глицерофосфохолина. |
| GDPD5 | Глицерофосфодиэфирная фосфодиэстераза, содержащая домен 5 | Эндоплазматический ретикулум, мембрана | Предполагается, что обеспечивает активность глицерофосфодиэстеразы. |
| PNPLA7 | Пататиноподобный фосфолипазный доменсодержащий белок 7 | Эндоплазматический ретикулум | Отвечает за превращение лизофосфатидилхолина в глицерофосфохолин в печеночном фосфатидилхолин-катаболическом пути. [1] |
| ENPP6 | Эктонуклеотидная пирофосфатаза/фосфодиэстераза 6 | Мембрана | Обеспечивает активность глицерофосфохолин-холинфосфодиэстеразы. |
База данных GO и анализ обогощения терминами
Первой БД для анализа списка генов выбрана Gene Ontology (GO), представляющая собой граф биологических терминов (узлов графа , GO терминов), связанных с другими терминами различными отношениями (ребрами графа). Продукт гена и термин GO связывает аннотация. Стандартная аннотация включает несколько аспектов, а именно:
- нормальную молекулярную функцию
(мутации и роль в развитии заболеваний не входят в сферу применения GO), - биологический процесс,
- клеточный компонент
(клеточное местоположение, где реализуется молекулярная функция)
генного продукта. Один из сервисов GO - это PANTHER, представляющий собой базу данных семейств филогенетических деревьев генов, обеспечивающих основу для аннотирования свойств белков. В частности, он позволяет провести анализ обогащения терминами (GO Enrichment Analysis -> Overrepresentation Test) для нахождения общих биологических функций заданных генов.
Параметры анализа: точный тест Фишера, поправка Бонферрони на множественное тестирование. В качестве набора данных аннотаций использовались аспекты GO. Все таблицы находок сортировались по p-value с поправкой Бонферрони.
Для аспекта биологический процесс выдача содержит 14815 терминов, из которых 18 являются значимыми, то есть имеют поправленный p-value < 0.05 (рис. 1). Получено, что все генные продукты участвуют в липидном обмене, в частности в метаболизме фосфо- и/или глицеролипидов. Из них 3 фермента участвуют в метаболизме аминов и/или алкоголя, 3 других - в метаболизме фосфатидилхолина (один из ферментов - GDPD3, указан в обеих списках).
Для аспекта молекулярная функция выдача содержит 5071 термин, из которых 9 являются значимыми (рис. 2). По выдаче видно, что ферменты являются фосфолипазами и фосфогидролазами, которые разрушают сложноэфирные связи. Из них 3 фермента (GDPD5, ENPP6 и GDE1) объединяет глицерофосфодиэстеразная активность.
Для аспекта клеточный компонент выдача содержит 1997 терминов, ни один из которых не является значимым. Это говорит о том, что продукты генов из анализируемого списка не сконцентрированы в каком-либо месте (компартменте или органелле) в клетке.
База данных STRING
Второй БД для анализа списка генов выбрана STRING, предназначенная для поиска информации об известных и предсказанных белок-белковых (физических и функциональных) взаимодействиях. Я знакома с этой базой данных, поэтому выбрала ее (хотя она и прогрузилась только с vpn). Анализ белковых взаимодействий представляется в том числе красивыми графами - это первое, что видит пользователь после ввода данных.
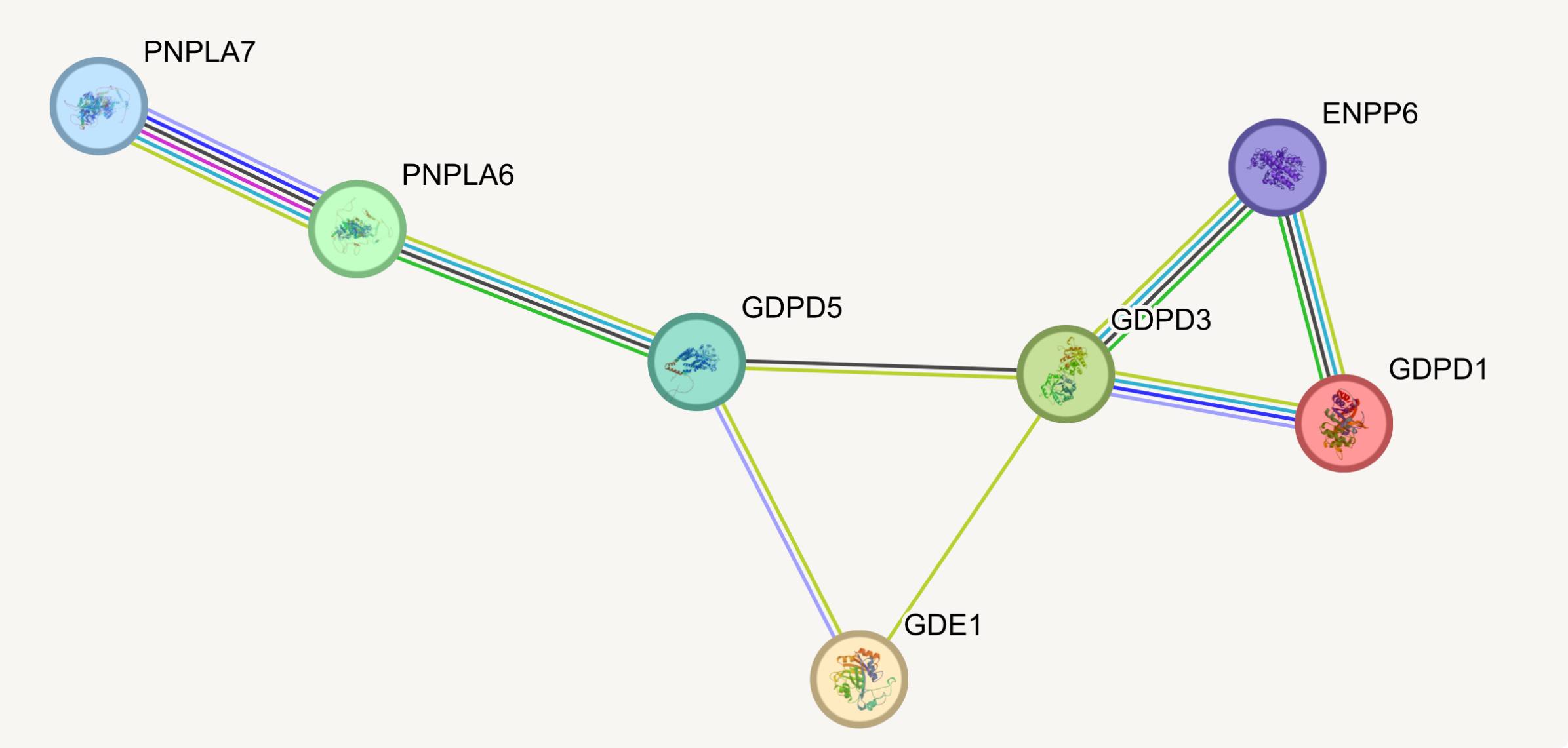

Для всех рассматриваемых ферментов известна или предсказана пространственная структура (рис. 3). Все белки обладают от одной до нескольких взаимосвязей. В циклы объединены попарно взаимодействующие ферменты, которые, например, совместно упоминаются в научных публикациях (салатовые ребра, textmining), что косвенно свидетельствует об их функциональной связи. Только для одной пары ферментов - PNPLA7 И PNPLA6, взаимодействие было определено эксперементально (розовое ребро, experimentally determined). Также серсвис приводит таблицу с некоторым саммари по каждому ферменту (названием, каталитической активностью, субстратной специфичностью и участием в метаболических путях).
Несмотря на то, что использование кластеризации характернее для больших наборов генов (когда получается «комок» ребер), мне интересно посмотреть, выделяются ли циклы графа в подгруппы. С использованием алгоритма MCL (Markov Cluster Algorithm, inflation parameter 3) для кластеризации, был получен рис. 6. Белки разделились на два кластера: два цикла с общим узлом (красный кластер) и пара узлов, между которыми больше всего интеракций (зеленый кластер).
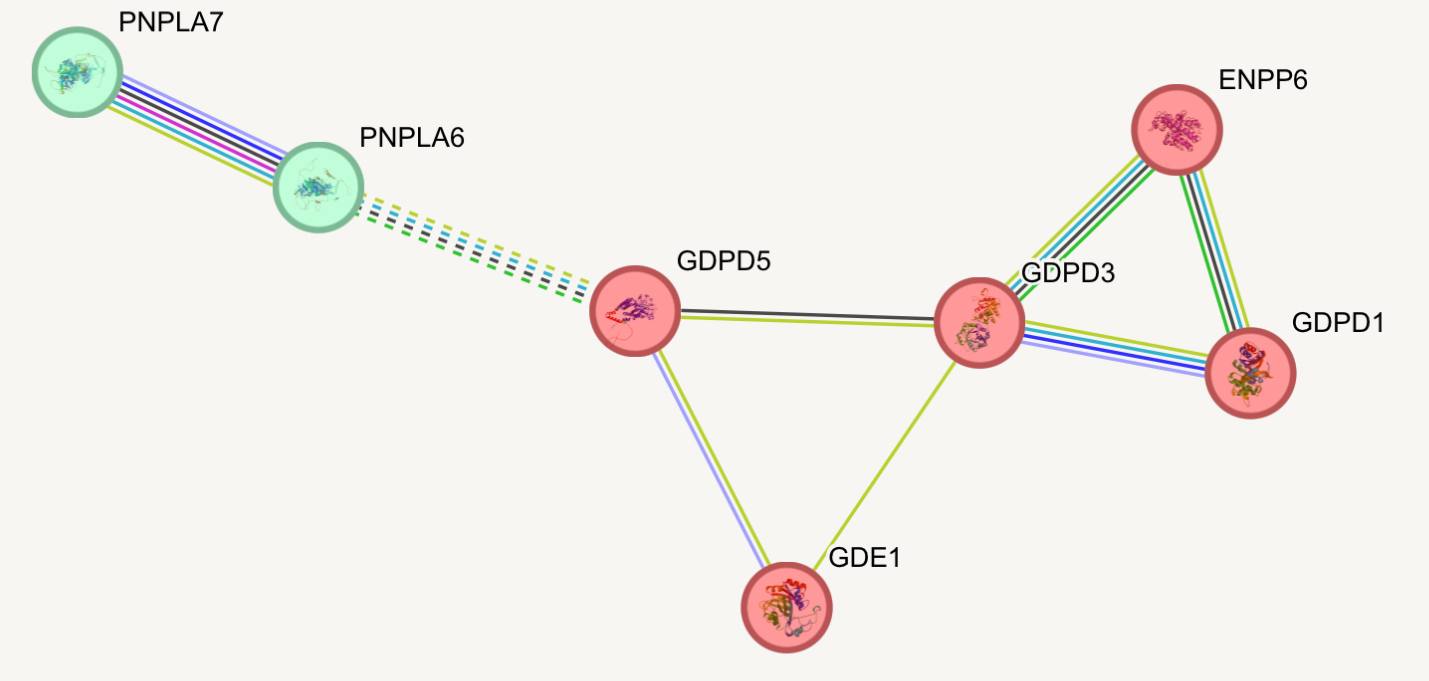
Кластеризация позволяет выделить функциональные модули, то есть белки внутри одного кластера с высокой вероятностью участвуют в одном и том же биологическом процессе.
- Красный кластер содержит 5 ферментов, участвующих в глицерофосфолипидном катаболизме;
- Зеленый кластер содержит 2 фермента, которые помимо участия в глицерофосфолипидном катаболизме объединяются по активности как фосфатидил фоспалипазы В и по домену связывания cNMP.
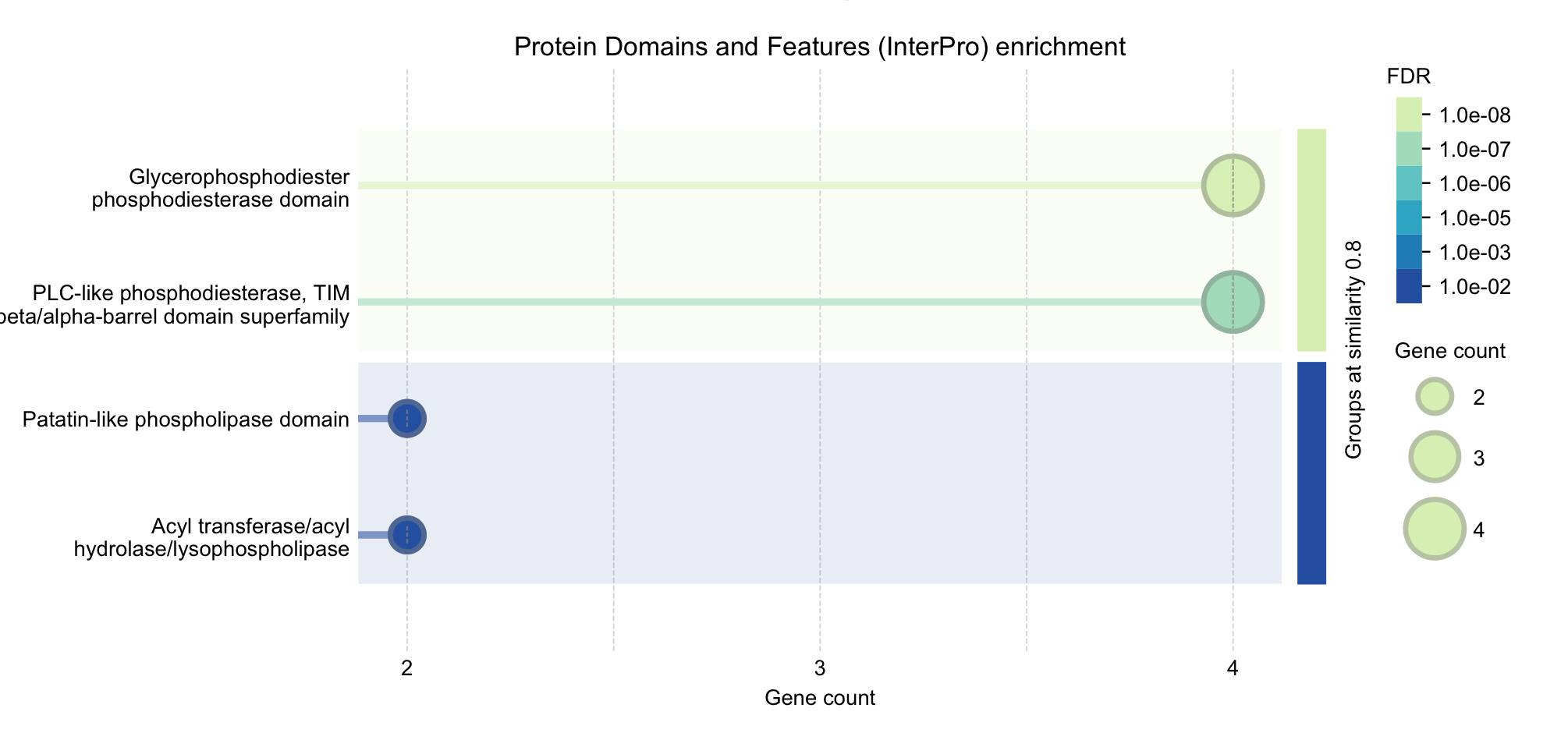
Также сервис приводит функциональное обогощение в сети, основанное на GO, публикациях в PubMed, метаболических путях (KEGG и Reactome) и белковых доменах. Обогащение по белковым доменам и фичам из InterPro (рис. 7) показывает (если покликать), что четыре фермента - GDPD5, GDPD3, GDPD1 и GDE1 содержат первые два домена. Другие два домена входят в состав белков PNPLA7 и PNPLA6. Таким образом, из рассмотрения выпал ENPP6. Распределение по доменам соответсвует распределению по кластерам.
Выводы
Итого, что же стало понятно о данном списке генов по результатам анализа?
- Продукты данных генов - ферменты, участвующие в метаболизме липидов и имеющие специфичность к фосфолипидам, а также участвующие в метаболизме этаноламинов;
- Эти белки обладают гидролазной активностью, разрушают сложноэфирные связи - деградируют липиды;
- Ферменты делятся на два кластера по доменам: 4 фермента содержат домен глицерофосфодиэстеразы и структурный домен фосфолипаз С; 2 фермента содержат пататин-подобный домен фосполипазы и структурный домен с 3-слойной топологией α/β/α. Домены приведены в том же порядке, что и на рис.7. Один из белков (ENPP6) не учитывался в анализе доменов.
Источники
- Harada, S.; Taketomi, Y.; Aiba, T.; Kawaguchi, M.; Hirabayashi, T.; Uranbileg, B.; Kurano, M.; Yatomi, Y.; Murakami, M. The Lysophospholipase PNPLA7 Controls Hepatic Choline and Methionine Metabolism. Biomolecules 2023, 13, 471. https://doi.org/10.3390/biom13030471