практикум №7
DeepTMHMM, OPM
Трансмембранные белки.
Трансмембранные белки (ТМБ) представляют собой особый класс мембранных белков, которые пронизывают клеточные мембраны и играют важную роль во многих биологических процессах. Предсказание их трёхмерной структуры остается сложной задачей структурной биоинформатики из-за уникальных физико-химических свойств и трудностей в экспериментальном определении структур. Современным инструментом для предсказания топологии и классификации ТМБ по последовательности является DeepTMHMM, основанный на глубоком обучении [1]. Для рассчета положения белка относительно мембраны (глубины погружения, угла наклона) используется БД Orientations of Proteins in Membranes (OPM), принимающая пространственные структуры белков из PDB и использующая физико-химические расчеты [2].
Целью практикума является сравнение предсказаний трансмембранных участков, сделанных вышеупомянутыми программами, в представителях двух основных структурных классов ТМБ: альфа-спирального и бета-листового белков.
Вместо «бета-листовых» в данном контексте правильнее говорить о «бета-цилиндрических» или «бета-бочонковых» белках (beta-barrel transmembrane protein).
Сравнение предсказаний в бета-листовом белке
Для анализа белка, в трансмембранной части которого находятся β-листы, в БД OPM был выбран гемолитический лектин CEL-III (hemolytic lectin CEL-III), выделенный из морского огурца и представляющий собой гомогептамер. Интерес к данной макромолекуле вызван тем, что это единственный белок в семействе эукариотических порообразующих токсинов в суперсемействе порообразующих бета-листковых токсинов (табл. 1) со структурой, вызывающей целый ряд ассоциаций (рис. 1).
| PDB ID | 3W9T |
| UniProt ID | CEL3_PSEEC |
| Тип | Трансмембранные белки |
| Класс | Бета-бочковые трансмембранные белки |
| Суперсемейство | Токсины, образующие бета-листы |
| Семейство | Эукариотические порообразующие токсины |
| Вид | Pseudocnus echinatus/Cucumaria echinata (Sea cucumber) |
| Локализация | Секретируемый токсин |
Данный лектин состоит из двух углеводсвязывающих доменов (1, 2) и одного домена олигомеризаци (3). После связывания с углеводными цепями на поверхности клетки через домены 1 и 2 домен 3 самоассоциируется, образуя трансмембранные поры, что приводит к лизису или гибели клетки, подобно другим порообразующим токсинам различных организмов [3].
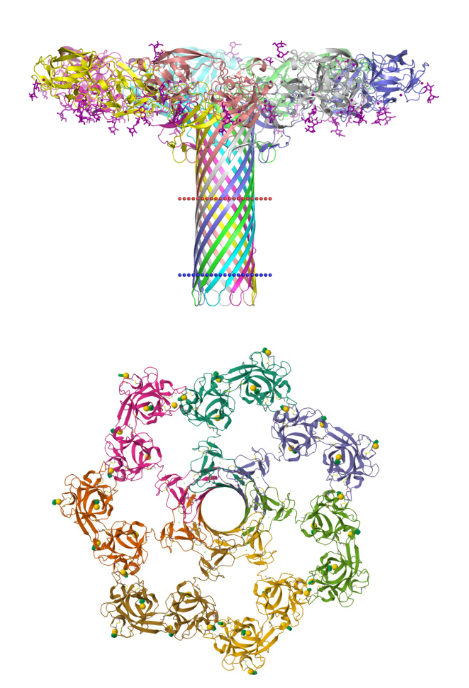
Сверху белок в мембранном окружении (ОРМ): красный пунктир - внешний липидный слой мембраны., синий пунктир - внутренний липид. сл. м. Снизу - вид в пору (PDB).
Координаты трансмембранных участков белка в OPM: 1(318- 330), 2(340- 353).
Предсказанные DeepTMHMM координаты трансмембранных участков: -
Последовательность цепи белка для запуска программы взята с PDB. Результат продемонстрирован на рис. 2 и в текстовом виде.
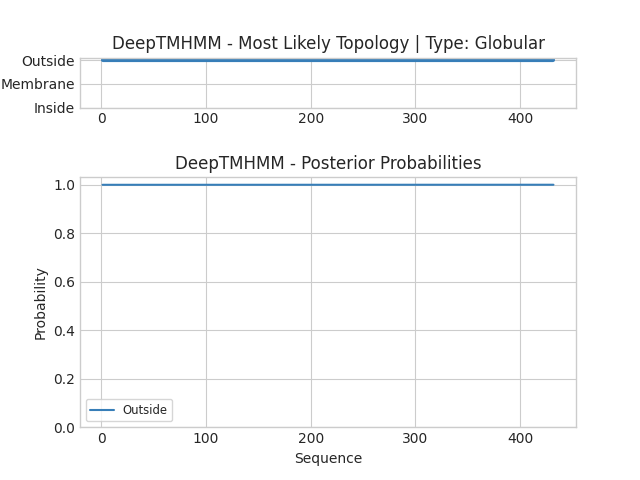
Трансмембранных участков предсказано не было. Я предполагаю, что это связано с биологией белка (см. выше). Подобные белки секретируются в растворимых формах, то есть имеют определенное количество заряженных аминокислот. Поскольку модель работает с вероятностями, то чем больше будет заряженных аминокислот, тем с большей вероятностью весь белок предскажется глобулярным. Действительно, в статье [3] преполагается, что гидрофобные участки белка станновятся доступными только после связывания токсина с мишенью, что, в свою очередь, приводит к гептамеризации и формированию поры. То есть кодирующая первичную аминокислотную последовательность и декодирующая топологию с помощью модели пространства состояний программа не может предсказать(/знать/учесть) конформационные перестройки белка в третичной форме.
Сравнение предсказаний в альфа-спиральном белке
Для анализа белка, в трансмембранной части которого находятся α-спирали, мне был выдан симпортер двухвалентных анионов/Na+ холерного вибриона и его гуманизированного варианта (divalent anion/Na+ symporter from Vibrio cholerae and a humanized variant). Название белка копирует название статьи [4], в которой была получена его структура, а точнее комплекс с ионами Na+ и субстратом (сукцинатом). Мембранные переносчики, принадлежащие к семейству симпортеров двухвалентных анионов/Na+ (DASS), перемещают промежуточные продукты цикла Кребса или сульфат через клеточную мембрану, как правило, используя уже существующий градиент Na+. Белок из семейства DASS холерного вибриона, то есть объект интереса, называется VcINDY - Na+-зависимый дикарбоксилатный транспортер из Vibrio cholerae. В БД UniProt он называется транспортер сеймейства NadC (Transporter, NadC family).
| PDB ID | 5uld |
| UniProt ID | Q9KNE0_VIBCH |
| Тип | Трансмембранные белки |
| Класс | Альфа-спиральный политоп |
| Суперсемейство | Переносчик ионов |
| Семейство | Двухвалентный анион: семейство Na+-переносчиков (DASS) |
| Вид | Vibrio cholerae |
| Локализация | Внутренняя мембрана грамотрицательных бактерий |
Данный транспортер представляет собой гомодимер, субъединицы состоят из двух идентичных цепей.

Последовательность цепи белка для запуска программы взята с PDB. Результат продемонстрирован на рис. 4 и в текстовом виде. В табл. 3 приведено сравнение предсказаний.
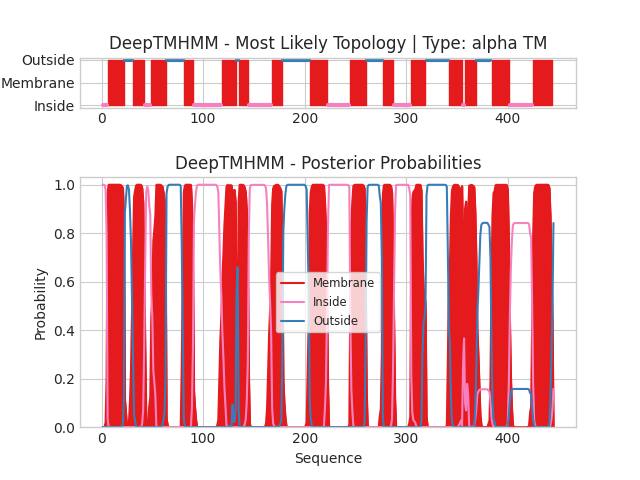
| № | Предсказание DeepTMHMM | Расчет OPM |
| 1 | 6-22 | 20-38 |
| 2 | 31-42 | 47-61 |
| 3 | 49-63 | 66-80 |
| 4 | 81-90 | 95-110 |
| 5 | 119-132 | / |
| 6 | 135-144 | / |
| 7 | 168-178 | 185-195 |
| 8 | 205-222 | 214-236 |
| 9 | 245-260 | 262-277 |
| 10 | 277-287 | 292-306 |
| 11 | 305-319 | 320-336 |
| 12 | 342-355 | 358-375 |
| 13 | 358-369 | 378-391 |
| 14 | 384-401 | 401-413 |
| 15 | 425-444 | 440-461 |
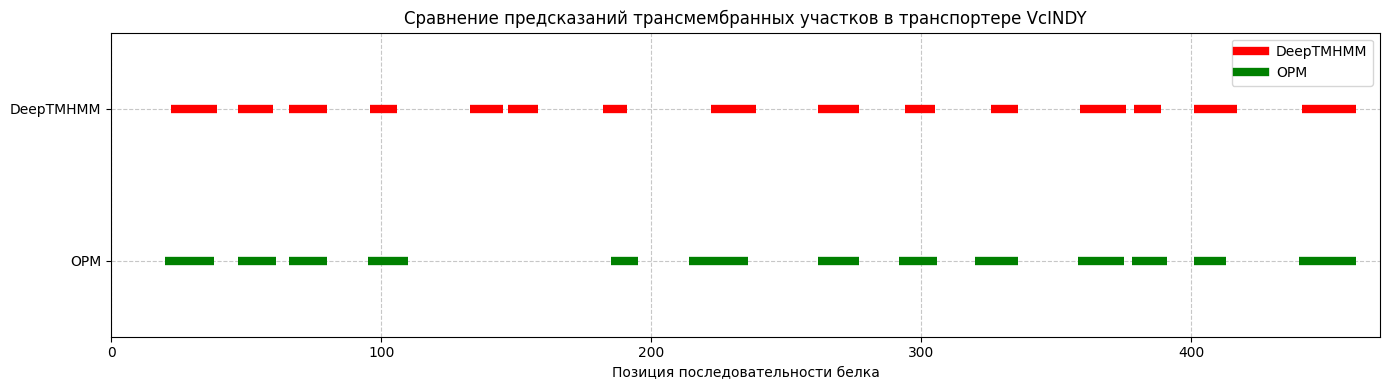
Чтобы было удобнее анализировать данные табл. 3, построим по ней график, на котором предсказанные и рассчитанные трансмембранные участки отложим в виде отрезков (рис. 5).
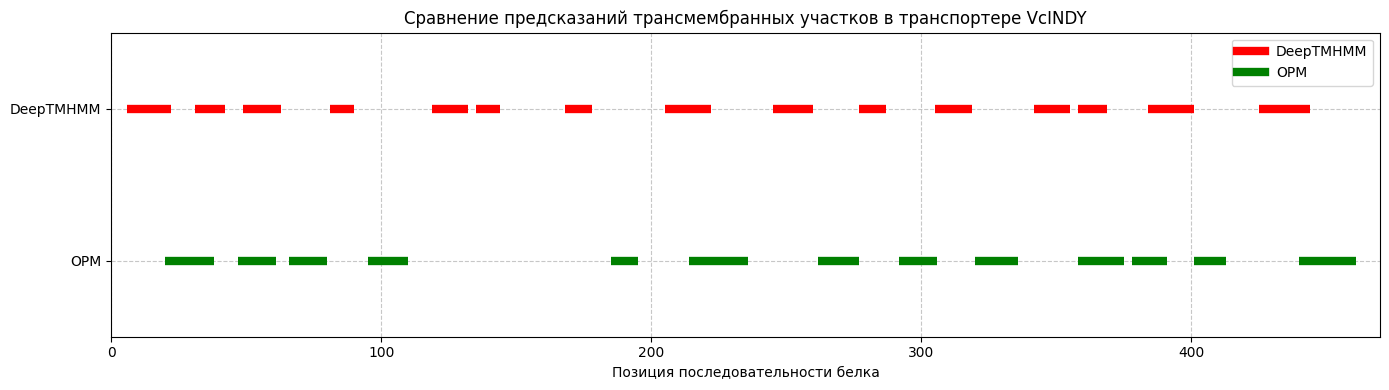
В целом, по графику видно, что предсказания похожи. Однако, в предсказании от DeepTMHMM на два ТМ участка больше (119-132, 135-144). ТМ участки практически не перекрываются, можно сказать, сдвинуты. Также сопоставимые участки различаются по длине.
В процессе выполнения практикума помимо упоминавшихся баз данных я также изучала PDBsum и обратила внимание, что для транспортера VcINDY указанные длины последовательности (в амк.) и структуры (также в амк.) не совпадают (рис. 6). Причем разница между этими длинами подозрительно похожа разницу тех позиций, на которые различаются предсказания выше. Оказывается, OPM для выполнения расчетов использует п-ть с UniProt, в то время как для предсказания в DeepTMHMM я подавала п-ть с PDB. Они, действительно, не совпадают по длине: с UniProt'а длинне. Таким образом, сравнение предсказаний, выполненное выше, вероятно, некорректно, поскольку использует хоть и немного, но различающиеся входные данные.
На рис. 7 и в текстовом файле приведены результаты запуска DeepTMHMM с поданной п-тью с UniProt'а. В табл. 4 приведено сравнение, а на рис. 8 - график предсказаний ТМ участков.
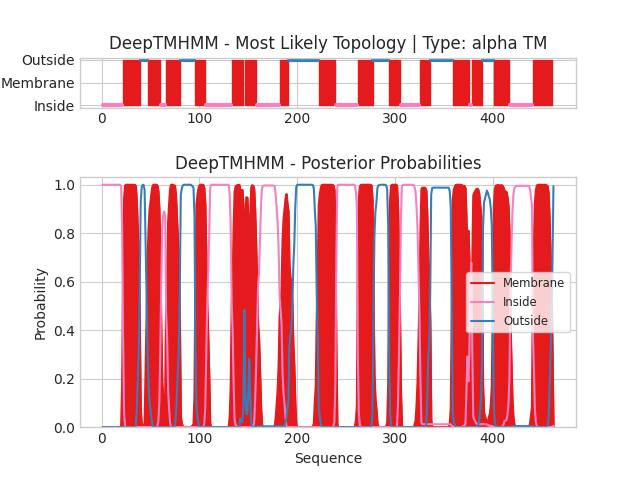
| № | Предсказание DeepTMHMM | Расчет OPM |
| 1 | 22-39 | 20-38 |
| 2 | 47-60 | 47-61 |
| 3 | 66-80 | 66-80 |
| 4 | 96-106 | 95-110 |
| 5 | 133-145 | / |
| 6 | 147-158 | / |
| 7 | 182-191 | 185-195 |
| 8 | 222-239 | 214-236 |
| 9 | 262-277 | 262-277 |
| 10 | 294-305 | 292-306 |
| 11 | 326-336 | 320-336 |
| 12 | 359-376 | 358-375 |
| 13 | 379-389 | 378-391 |
| 14 | 401-417 | 401-413 |
| 15 | 441-461 | 440-461 |
Теперь предсказания совпали более полно, есть перекрывания, границы отличаются незначительно. При этом в предсказании DeepTMHMM сохранилось два дополнительных участка (133-145, 147-158). Это может быть связано с тем, что структура белка получена в комплексе с лигандом, из-за чего происходит изменение конформации и пропадают два ТМ участка, либо иметь другую причину.
- Hallgren J. et al. DeepTMHMM predicts alpha and beta transmembrane proteins using deep neural networks //biorxiv. – 2022. – С. 2022.04. 08.487609.
- Lomize M. A. et al. OPM: orientations of proteins in membranes database //Bioinformatics. – 2006. – Т. 22. – №. 5. – С. 623-625.
- Unno, Hideaki et al. “Hemolytic lectin CEL-III heptamerizes via a large structural transition from α-helices to a β-barrel during the transmembrane pore formation process.” The Journal of biological chemistry vol. 289,18 (2014): 12805-12. doi:10.1074/jbc.M113.541896
- Nie, R., Stark, S., Symersky, J. et al. Structure and function of the divalent anion/Na+ symporter from Vibrio cholerae and a humanized variant. Nat Commun 8, 15009 (2017). https://doi.org/10.1038/ncomms15009