практикум №8
SECISearch3
Сигналы в геноме. Поиск известных сигналов.
Интересным для меня сигналом, закодированным в геноме, является последовательность вставки селеноцистеина SECIS (selenocysteine insertion sequence).
Все живые организмы обладают способностью преобразовывать кодоны UGA из сигнала о прекращении трансляции в кодон селеноцистеина (Sec). Белки, в которые успешно включается Sec, называются селенопротеинами и выполняют функции оксидоредуктаз [1].
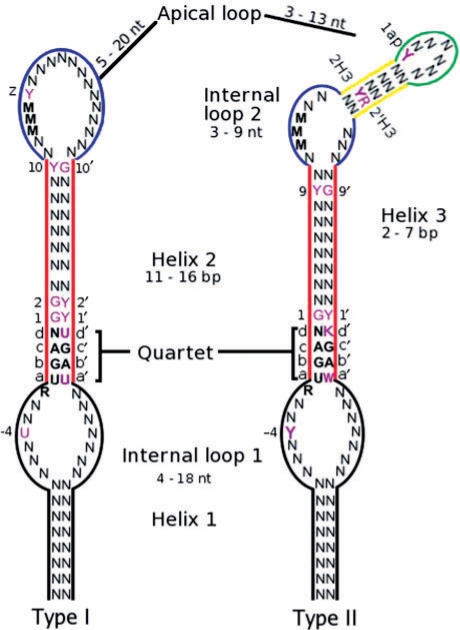
Система декодирования сигнала у эукариот включает как минимум два белка: фактор элонгации, специфичный для селеноцистеин-тРНК (eEFSec) и SECIS-связывающий белок 2 (SBP2), взаимодействующий с последовательностью SECIS в 3'-нетранслируемой области (3'-НТО) транскрипта мРНК селенопротеинов (рис. 3, 4) [2]. SECIS имеет минимальный функциональный размер 50–60 нуклеотидов, представляет собой структуру стебль-петля (шпильку) с тремя консервативными последовательностями: AUGА, (A)AA, GA (рис. 1, 2). Консервативные остатки (А)AA находятся на 5'-стороне апикальной петли, а основания AUGA и GA расположены близко друг к другу на противоположных сторонах спирального стебля [3]. Существует два типа эукариотических SECIS: тип I и тип II, которые отличаются наличием дополнительной спирали 3 в типе II. На рис. 2 приведены примеры сигнала SECIS эукариот, а именно консенсусные последовательности обоих типов [9].
SECIS, должно быть, является эффективным и важным сигналом для включения селеноцистеина в эукариотические клетки, поскольку поддерживает биосинтез селенопротеинов, играющих особую биологическую роль и обладающих важными каталитическими функциями. То есть, не будь сигнал эффективным, селенопротеины лишились бы функционального активного центра. Также приведу примеры селенопротеинов:
- Глутатионпероксидаза 1 (GPx1) участвует в детоксикации перекиси водорода и является одним из важнейших антиоксидантных ферментов организма человека [8];
- Йодтирониндейодиназа 1 (DIO1) активирует гормон щитовидной железы;
- Селенопротеин P (SelP) интересен тем, что содержит несколько кодонов UGA, кодирующих селеноцистеин, и всего один SECIS-элемент в 3'-НТО, который обеспечивает включение всех селеноцистеиновых остатков. Его точная функция ещё не выяснена, однако считается, что он обладает антиоксидантными свойствами [7].
Сервис для поиска SECIS
Для правильной аннотации селенопротеинов (чтобы избежать интерпретации UGA как сигнала остановки трансляции) разработаны специальные инструменты, например SECISearch3 [9]. SECISearch3 — это усовершенствованный конвейер для прогнозирования SECIS-элементов, развивший подходы оригинального SECISearch.
Описание работы сервиса
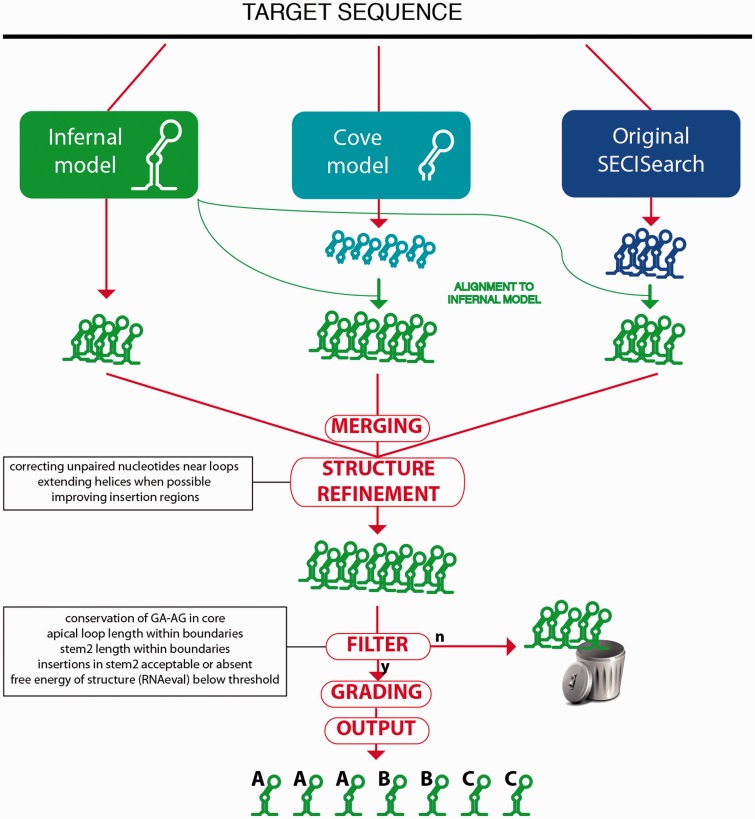
SECISearch использует PatScan для поиска по рукописным шаблонам последовательности, моделирующим спирали, петли и консервативное ядро SECIS. Результаты фильтруются по термодинамической стабильности (RNAfold) и структурным характеристикам. Ограничением метода является зависимость от шаблонов, требующих адаптации для новых видов. SECISearch3 объединяет несколько методов: Infernal (на основе ковариационных моделей) стал центральным методом прогнозирования и для оценки структур, предсказанных Covels и SECISearch. Конвейер обработки состоит из таких этапов, как объединение прогнозов от всех трёх методов, удаление избыточных кандидатов, структурное уточнение и оценка термодинамической стабильности (RNAeval), фильтрация по ключевым признакам. Ранжирование итоговых структур основано на важных структурных параметрах.
Запуск сервиса на примере
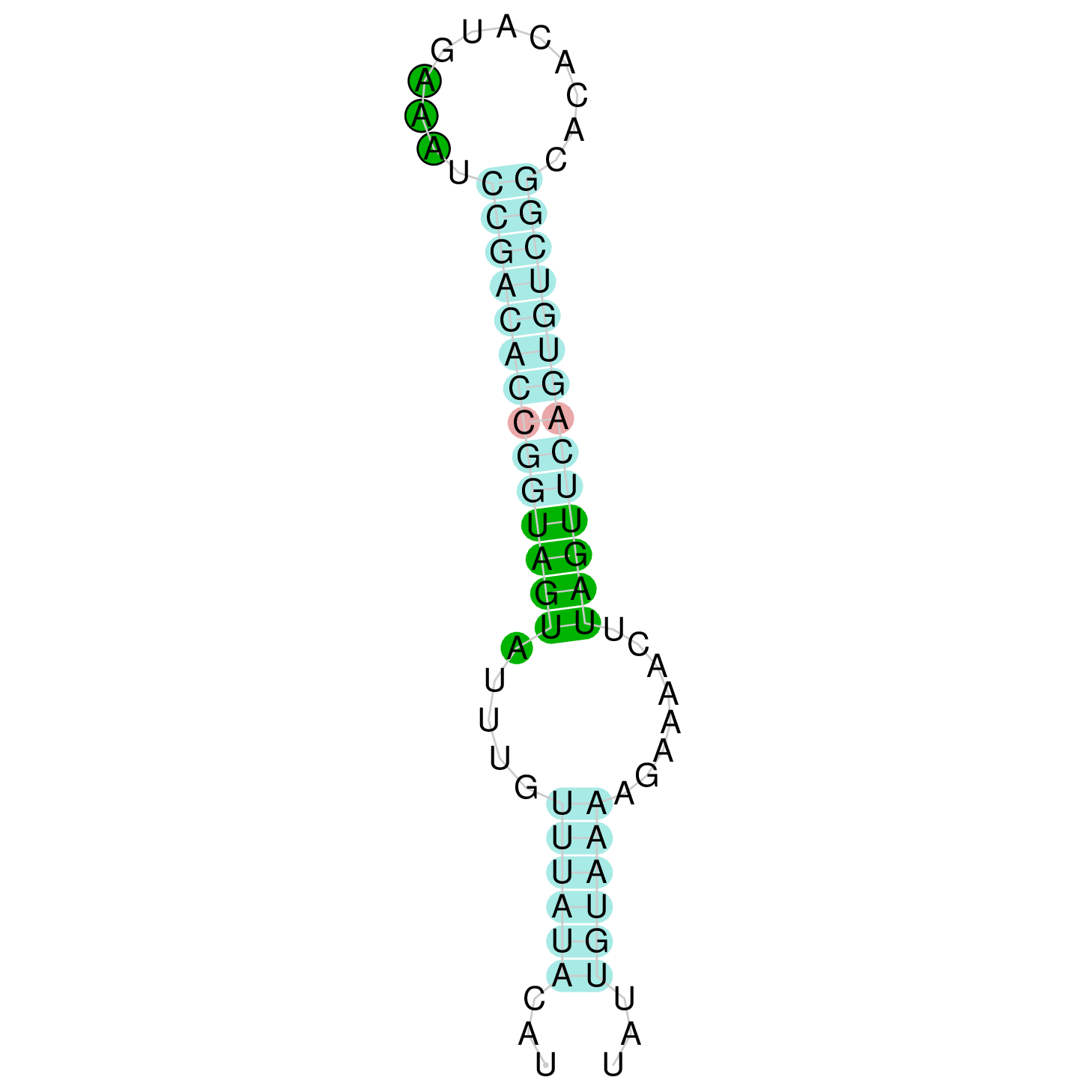
Примером для проверки работоспособности сервиса будет ген упоминавшегося раннее DIO1 (п-ти транскриптов с NCBI). Допустим, попробуем первый транскрипт (NM_000792.7). Сервисом предсказан один элемент SECIS в районе 1745-1815 нуклеотидов положительной цепи. Шпилька имеет оценку «А», принадлежит к типу I.
- Lobanov, A.V., Fomenko, D.E., Zhang, Y. et al. Evolutionary dynamics of eukaryotic selenoproteomes: large selenoproteomes may associate with aquatic life and small with terrestrial life. Genome Biol 8, R198 (2007). https://doi.org/10.1186/gb-2007-8-9-r198
- Xu H. et al. Identification of selenocysteine insertion sequence (SECIS) element in eukaryotic selenoproteins by RNA Draw program //Chinese Science Bulletin. – 2001. – Т. 46. – №. 14. – С. 1159-1161.
- Donovan, J., Copeland, P.R. Evolutionary history of selenocysteine incorporation from the perspective of SECIS binding proteins. BMC Evol Biol 9, 229 (2009). https://doi.org/10.1186/1471-2148-9-229
- Walczak R, Westhof E, Carbon P, Krol A. A novel RNA structural motif in the selenocysteine insertion element of eukaryotic selenoprotein mRNAs. RNA. 1996 Apr;2(4):367-79. PMID: 8634917; PMCID: PMC1369379.
- Fletcher JE, Copeland PR, Driscoll DM, Krol A. The selenocysteine incorporation machinery: interactions between the SECIS RNA and the SECIS-binding protein SBP2. RNA. 2001 Oct;7(10):1442-53. PMID: 11680849; PMCID: PMC1370188.
- Atkins, J., Gesteland, R. The twenty-first amino acid. Nature 407, 463–464 (2000). https://doi.org/10.1038/35035189
- Mostert, V. “Selenoprotein P: properties, functions, and regulation.” Archives of biochemistry and biophysics vol. 376,2 (2000): 433-8. doi:10.1006/abbi.2000.1735
- Entrez Gene: GPX1 glutathione peroxidase 1
- Mariotti M, Lobanov AV, Guigo R, Gladyshev VN. SECISearch3 and Seblastian: new tools for prediction of SECIS elements and selenoproteins. Nucleic Acids Res. 2013 Aug;41(15):e149. doi: 10.1093/nar/gkt550. Epub 2013 Jun 19. PMID: 23783574; PMCID: PMC3753652.