Практикум 4
Предсказание вторичной структуры тРНК
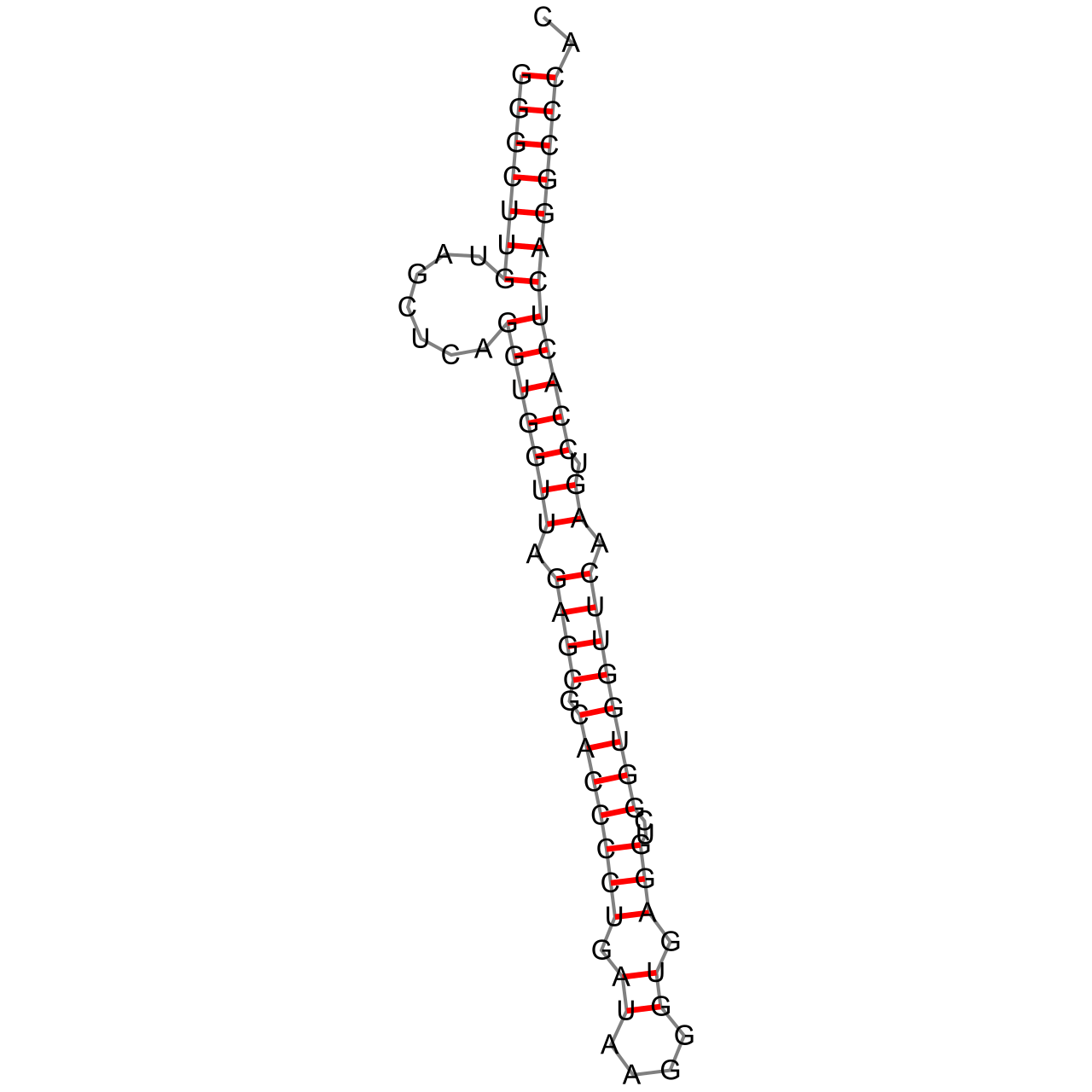
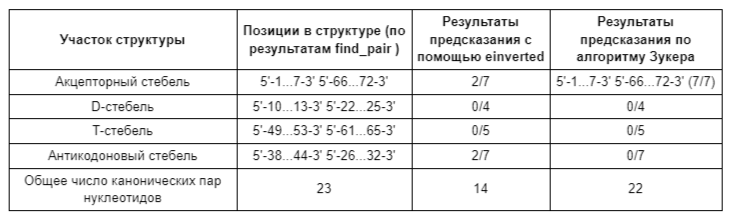
Была предсказана структура заданной из прошлого практикума тРНК с помощью программы einverted (параметры -gap 10, -threshold 8, -match 2, -mismatch 2) и по алгоритму Зукера (Рис. 1) с помощью модуля ViennaRna и языка Python, результаты были сравнены с выдачей программы find_pair (смотри прошлый практикум и Таб. 1):
Исследование комплекса ДНК-белок
Для проведения исследования и анализа была задана структура 1R4O.
Сценарии для выделения множеств атомов и визуализации
Были написаны два сценария: первый, который содержит команды для выделения заданных множеств атомов в PyMOL, второй является исполняемым, написан на языке Python, запускает визуалицию выделения атомов из заданных множеств в первом сценарии. Аналогичные сценарии были также написаны и для программы JMol: первый и второй.
Поиск нужных ДНК-белковых контактов
Поиск необходимых контактов осуществлялся командами PyMOL (их можно посмотреть здесь), на основе этого была создана следующая таблица (Таб. 2):
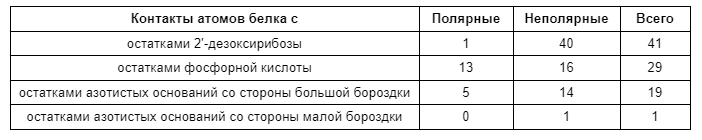
Из таблицы видно, что в данном комплексе преобладают в основном неполярные контакты между белком и ДНК (полярных связей крайне мало), также можно заметить, что атомы малой бороздки практически не играют никакой роли во взаимодействии данных макромолекул. В основном ключевая роль в контакте и связывании белка и нуклеиновой кислоты играет сахаро-фосфатный остов последней.
Схема ДНК-белковых контактов
С помощью программы nucplot была получена популярная схема ДНК-белковых контактов (Рис. 2):
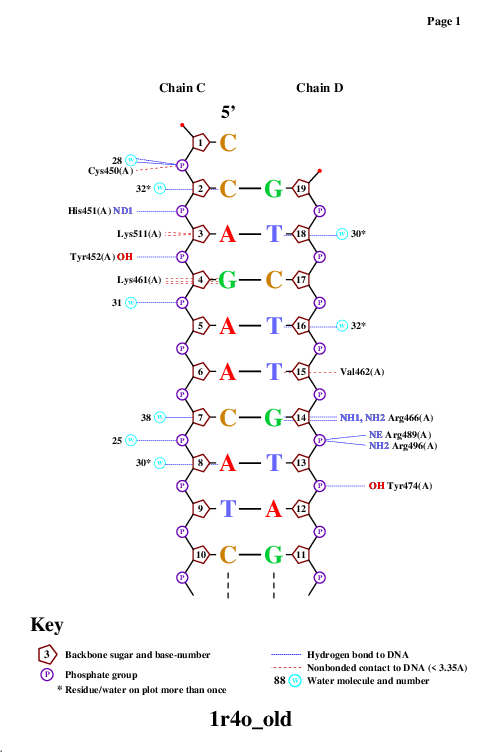
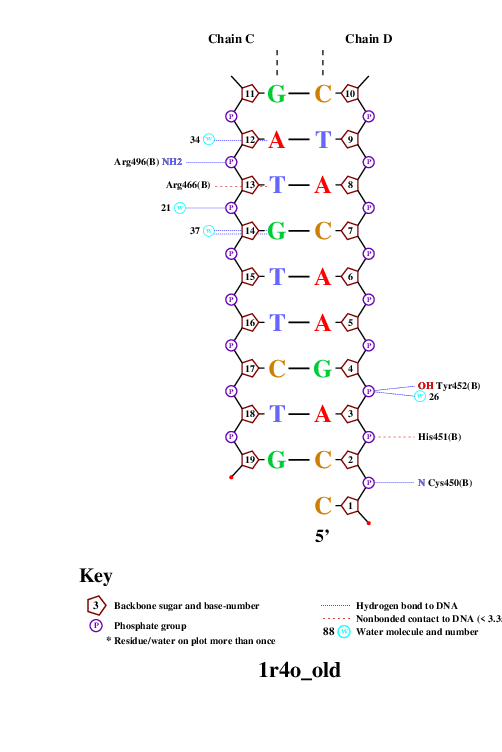
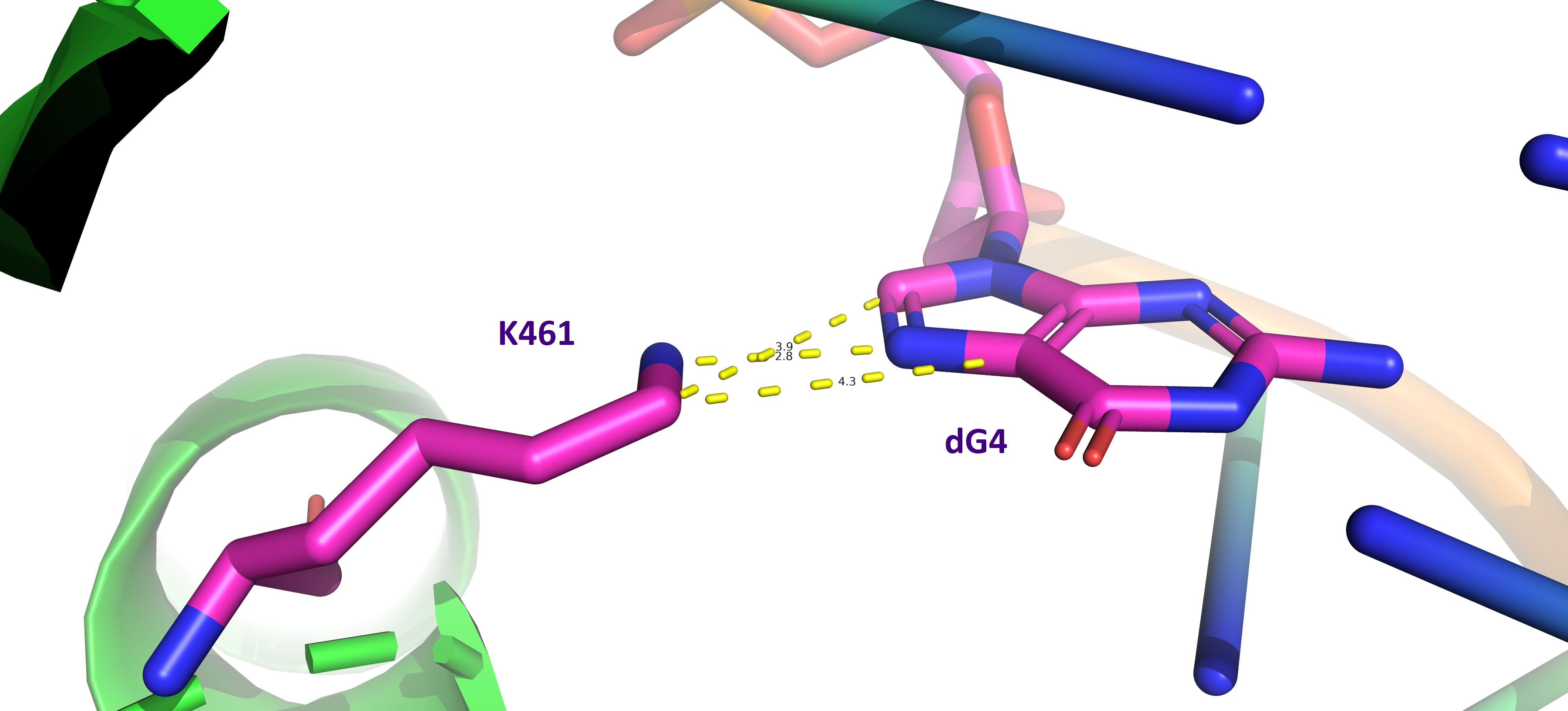
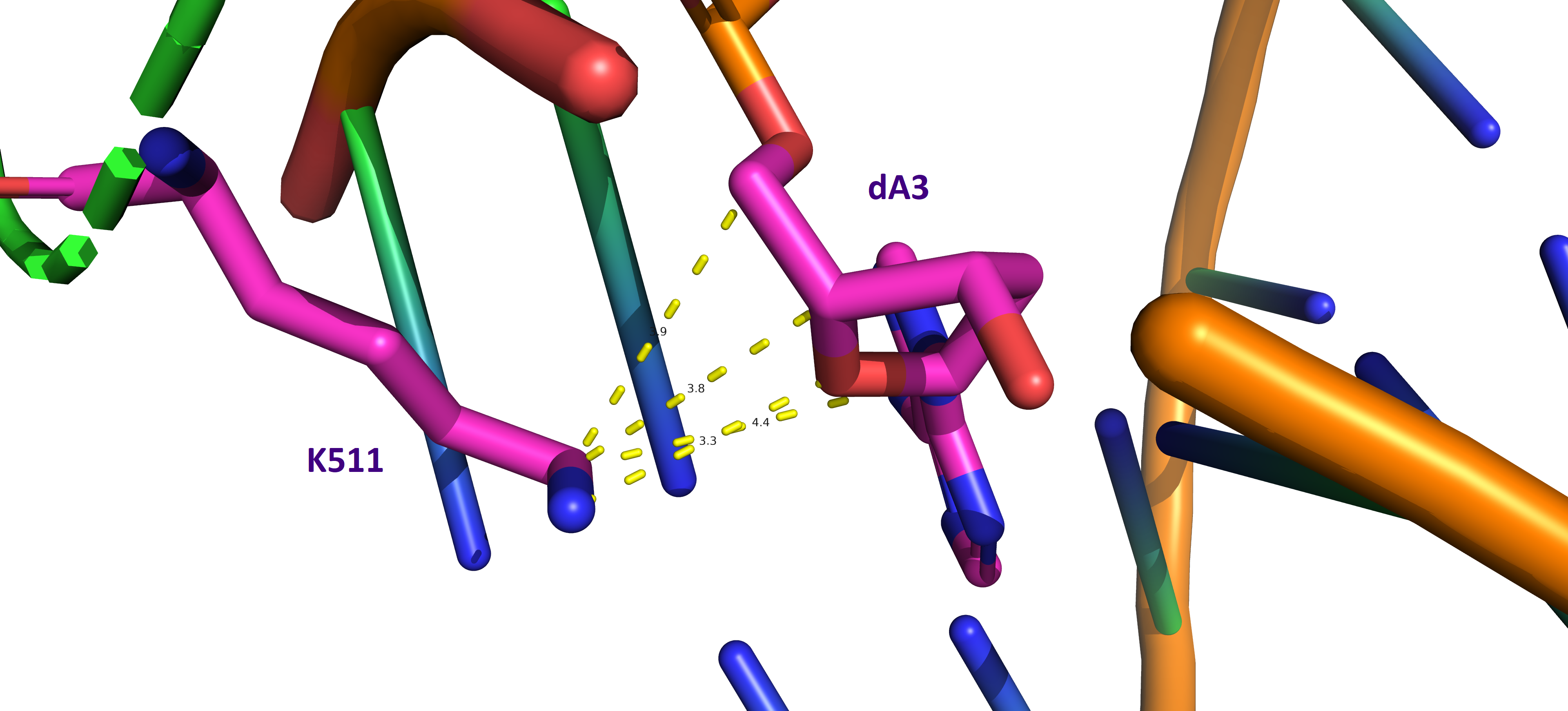
Наиболее важная аминокислота
Из схемы, иллюстрированной выше, видно, что наибольшее число контактов с заданной ДНК образует наибольшее число контактов остаток лизина Lys461 на A-цепи белковой части, этот же остаток был выбран мною вместе с остатком Lys511 на той же A-цепи как наиболее важные для распознавания ДНК, поскольку эти два лизина находятся довольно близко и оба взаимодействуют с соседними нуклеотидами ДНК, тем самым распознавая участок AG для начала взаимодействия (Рис. 3 и Рис. 4):