Практикум 4
1. Составление списка гомологичных белков, включающих паралоги
В данном задании было необходимо найти белки, гомологичные CLPX_ECOLI (АТФ-связывающая субъединица ClpX протеазы Clp Escherichia coli) среди последовательностей протеомов выбранных бактерий. Для этого после их индексирования программой makeblastdb по полученнму банку данных был проведен поиск программой blastp с последовательностью белка CLPX_ECOLI в качестве запроса. Порог по E-value был выставлен на 0.001. Использованные команды представлены ниже на примере протеома Corynebacterium efficiens (мнемоника COREF):
makeblastdb -in /P/y21/term4/Proteomes/COREF.fasta -dbtype prot -out db.fasta
blastp -task blastp -query CLPX_ECOLI.fasta -db db.fasta -out ./blastp/COREF.fasta -evalue 0.001
Для автоматизации процесса они были включены в bash-скрипт, проводящий поиск последовательно в каждом из протеомов. Выдача скрипта включает следующие файлы:
- Список находок с их весом в битах и E-value
- Список заголовков найденных последовательностей
- Fasta-файл с последовательностями находок.
2. Реконструкция и визуализация
После удаления из полученного fasta-файла описания последовательностей он был использован для построения филогенетического дерева найденных белков. Для этого он был загружен на сайт NGPhylogeny, где для него был запущен конвейер, состоящий из алгориттма множественного выравнивания MAFFT с параметрами по умолчанию и алгоритма реконструкции филогенетических деревьев FastME. Для последнего была отключена опция "Gamma distributed rates across sites", был выбран алгоритм построения начального дерева "BIONJ", а число bootstrap-реплик было выставлено на 100.
Формула полученного дерева в Newick-формате доступна по данной ссылке.
При условии правильной реконструкции филогенетического дерева оно несет информацию о парах ортологов и паралогов.
Примеры ортологов:
- CLPX_COREF - CLPX_CORDI
- Q47KU4_THEFY - A0LRB8_ACIC1
- Q8FMG2_COREF - Q6NF92_CORDI
Примеры паралогов:
- FTSH_MYCLE - CLPX_MYCLE
- A1TG29_MYCVP - A1TG43_MYCVP
- Q1AVT0_RUBXD - Q1AU05_RUBXD
Построенное дерево было переукоренено в среднюю точку. При детальном рассмотрении на нем явно заметны три ортологические группы - клады со схожим порядком ветвления, включающие белки большей части выбранных бактерий. Они были покрашены в разные цвета (Рис. 1) и "схлопнуты" (Рис. 2). Количества деревьев из bootstrap-реплик, включающих каждую из ветвей, отмечены числами.
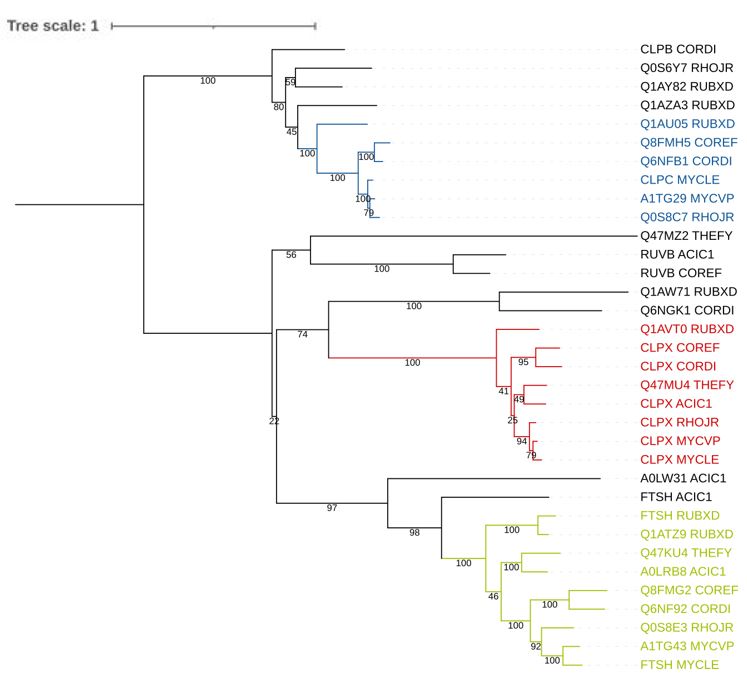
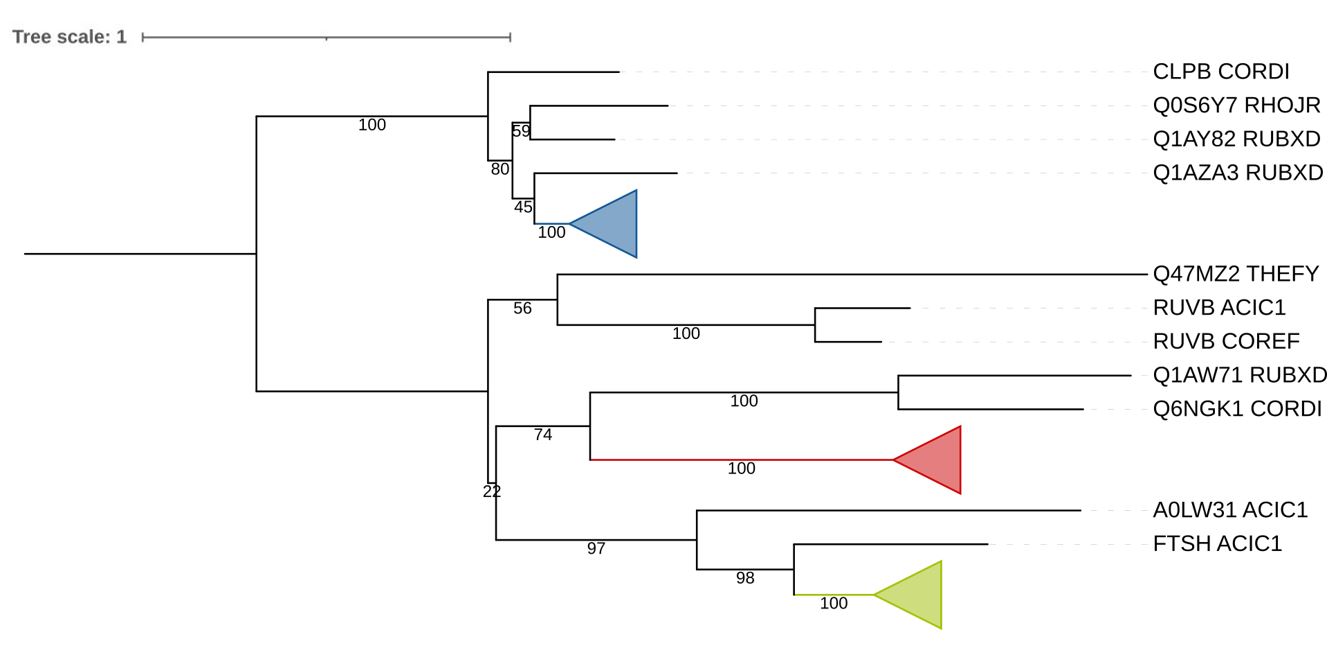
Клада, покрашенная в зеленый цвет, по топологии полностью соответствует филогенетическому дереву выбранных видов, что согласуется с высокими поддержками большинства ее ветвей. Два из девяти входящих в нее белков принадлежат RUBXD, тогда как остальные виды представлены только одной последовательностью. При этом все входящие в кладу белки являются цинковыми металлопротеиназами FtsH.
Красная клада также содержит белки всех выбранных организмов, однако отличается от зеленой порядком отхождения ветвей, соответствующих группам {THEFY, ACIC1} и {COREF, CORDI}. При этом поддержка разделяющей их неправильной ветви равна 25, что является самым низким значением для данной клады. Все восемь входящих в нее белков являются АТФ-связывающими субъединицами ClpX протеиназ семейства Clp.
Синяя клада не содержит белков бактерий THEFY и ACIC1, а в пределах группы {MYCLE, MYCVP, RHOJR} имеет неправильное ветвление. Уровень поддержки ветви, противопоставляющей листья MYCVP и RHOJR белку MYCLE, составляет 79, что достаточно много, но все же меньше значений для остальных ветвей клады. Из шести входящих в кладу белков четыре являются АТФ-связывающими субъединицами протеиназ семмейства Clp (для двух из них функция предсказана), а оставшиеся два подписаны в fasta-файле как белки, содержащие АТФазный домен ААА-2, наличие которого характерно для данного семейства (doi:10.1111/j.1365-2958.2007.05598.x). Отсутствие в синей кладе белков THEFY и ACIC1 может быть связано с делецией соответствующего гена, произошедшей у их общего предка.