Практикум 1
В данном практикуме я работал со структурой фермента S-аденозил-L-гомоцистеин гидролазы (PDB ID 5M65) в комплексе с лигандами: NAD+ и аденином. Этот белок участвует в регенерации S-аденозилметионина (SAM) и осуществляет расщепление его производного, образующегося при переносе метильной группы, на аденозин и гомоцистеин. Модель включает две цепи, образующие гомодимер [1].
Исследуемый комплекс не соответствует какой-либо из реальных стадий работы фермента, так как вместо своего субстрата, S-аденозилгомоцистеина (или продуктов его гидролиза), он содержит лишь небольшой фрагмент этой молекулы — аденин.
Задание 1
Для поиска взаимодействий белка с его лигандами сначала были выделены остатки, имеющие атомы на расстоянии не более 4 Å от NAD+ и аденина, а также от связанного рядом с ним иона Na+. Полярные контакты были предсказаны с помощью функции "A > find > polar contacts". Полученные взаимодействия были проверены вручную. Были приняты только водородные связи, для которых донорно-акцепторное расстояние лежит в диапазоне от 2.3 до 3.5 Å [2], а значение угла "D..H..A" не превышает 30° (атомы водорода в модели отсутствуют, поэтому углы были оценены "на глаз" с использованием знаний о геометрии химических связей). Поиск других взаимодействий проводился вручную, однако из них были обнаружены только контакты с ионом натрия, который в данном случае хелатируется атомами кислорода трех амидных групп в составе белка. Все найденные взаимодействия и участвующие в них остатки активного центра представлены на рисунке ниже (Рис. 1).
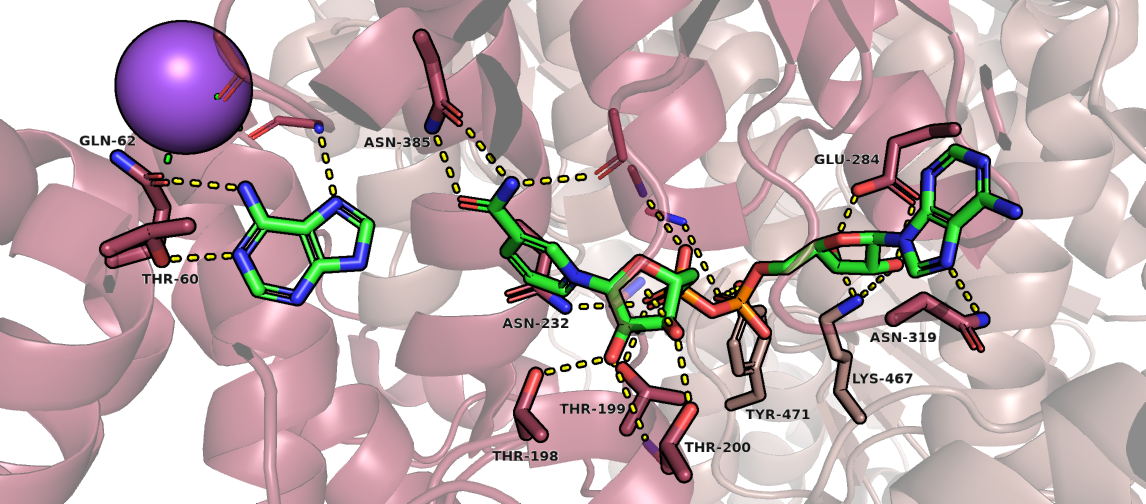
Несмотря на то что в комплексе естественный субстрат замещён на аденин, азотистое основание связано с белком несколькими водородными связями в плоскости кольца. В непосредственной близости от аденина три атома кислорода (два в составе пептидных связей и ещё один в боковой цепи глутамина) координируют катион натрия. В то же время остаток аденина, входящий в состав NAD+, связывается всего одной водородной связью с атомом азота в амидной группе аспарагина.
Аминокислоты белка образуют сразу три водородные связи с никотинамидом. В двух из них участвует амидная группа боковой цепи аспарагина, повёрнутая на 180° по отношению к амидной группе лиганда, тогда как в третьей в качестве акцептора выступает кислород пептидной связи в составе неструктурированной петли.
Стоит отметить, что несмотря на наличие в лигандах сразу трёх ароматических групп достоверные стекинг-взаимодействия обнаружить не удалось.
Каждая из двух гидроксильных групп остатка рибозы, связанного с никотинамидом, вступает в две водородные связи: с амидной группой в составе пептидной связи белка и с гидроксильной группой остатка треонина. Два атома кислорода в гидроксильных группах рибозы, принадлежащей к остатку аденозина, также связываются в активном центре фермента одинаковым образом. Каждый из них участвует в образовании двух водородных связей: в качестве акцептора с аминогруппой лизина 467 и в качестве донора — с кислородом карбоксигруппы глутамата 284.
Два фосфата в составе кофактора также удерживаются несколькими водородными связями. Хотя было бы логично предположить их участие в образовании солевых мостиков, на достаточно близком расстоянии от них (4 Å) [3] нет положительно заряженных групп.
Что интересно, судя по всему, в удерживании лигандов, расположенных в сайте связывания одной цепи, участвуют остатки обеих цепей гомодимера. В частности, остаток лизина 467 цепи B формирует две водородные связи с гидроксильными группами рибозы, а остаток тирозина 471 участвует в связывании одного из фосфатов.
Для выполнения этого задания были написаны два скрипта: для визуализации (взаимодействия были добавлены вручную) и для отрисовки изображения. Они доступны по ссылкам ниже. Для выделения некоторых остатков картинка была получена в виде двух слоев, которые были наложены друг на друга с помощью следующей команды:
composite -gravity center overlay.png base.png composite.png
Ссылка на проект pymol для задания 1
Скрипт для отрисовки изображения
Задание 2
В этом задании нужно было визуализировать в виде сетки (mesh) электронную плотность вокруг лиганда и окружающих остатков. Для этого помимо файла в pdb-файла для записи 5M65 был также скачан файл в формате 2fofc. Ниже представлено отображение электронной плотности при уровне подрезки 1 (Рис. 2).
Электронная плотность достаточно хорошо и примерно одинаково покрывает как лиганды, так и аминокислотные остатки белка. Возможно, вокруг лигандов ее ненамного меньше, что может быть связано с их большей степенью подвижностью по отношению к участкам полипептидной цели.
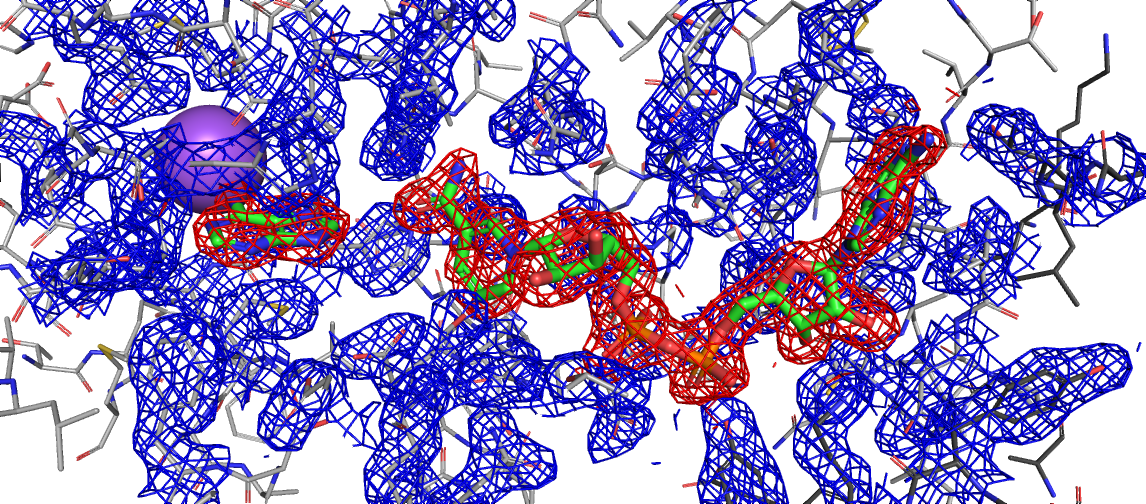
Электронная плотность достаточно хорошо и примерно одинаково покрывает как лиганды, так и аминокислотные остатки белка. Возможно, вокруг лигандов ее ненамного меньше, что может быть связано с их большей степенью подвижностью по отношению к участкам полипептидной цели.
Для визуализации электронной плотности использовался следующий скрипт:
Скрипт для визуализации (задание 2)
Источники
- Manszewski, T., Szpotkowski, K., & Jaskolski, M. (2017). Crystallographic and SAXS studies of S-adenosyl-l-homocysteine hydrolase from Bradyrhizobium elkanii. IUCrJ, 4(Pt 3), 271–282.
- George A. Jeffrey (1997). An introduction to hydrogen bonding. Oxford University Press.
- Kumar S., Nussinov R. (2002). Close-range electrostatic interactions in proteins. ChemBioChem, 3(7), 604–17.