Практикум 4. Комплексы ДНК-белок.
Предсказание вторичной структуры заданной тРНК
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Последовательность нуклеотидов тРНК с 3D-структурой 2cv0 была скачана с соответствующей страницы PDB. Поиск канонических пар в стеблях вначале осуществлялся с помощью команды "einverted" пакета "EMBOSS".
einverted 11.fasta -gap 15 -thr 10 -match 4 -mis -10 -outfile 1.inv -outseq 1.fasta
Относительно адекватные результаты дали параметры: штраф за гэп 15, порог очков не менее 10, 4 очка за совпадение, -10 за несовпадение. Но и они ничего не сказали про T- и D- стебли, только про акцепторный и антикодоновый. Результат выравнивания доступен по ссылке.
Предсказание вторичной структуры тРНК по алгоритму Зукера
Помимо метода инвертируемых пар использовался алгоритм Зукера, его запуск, перевод постскирпт файла в pdf и конечный результат предоставлены ниже.
export PATH=${PATH}:/home/preps/golovin/progs/bin
cat 11.fasta | RNAfold --noconv --MEA
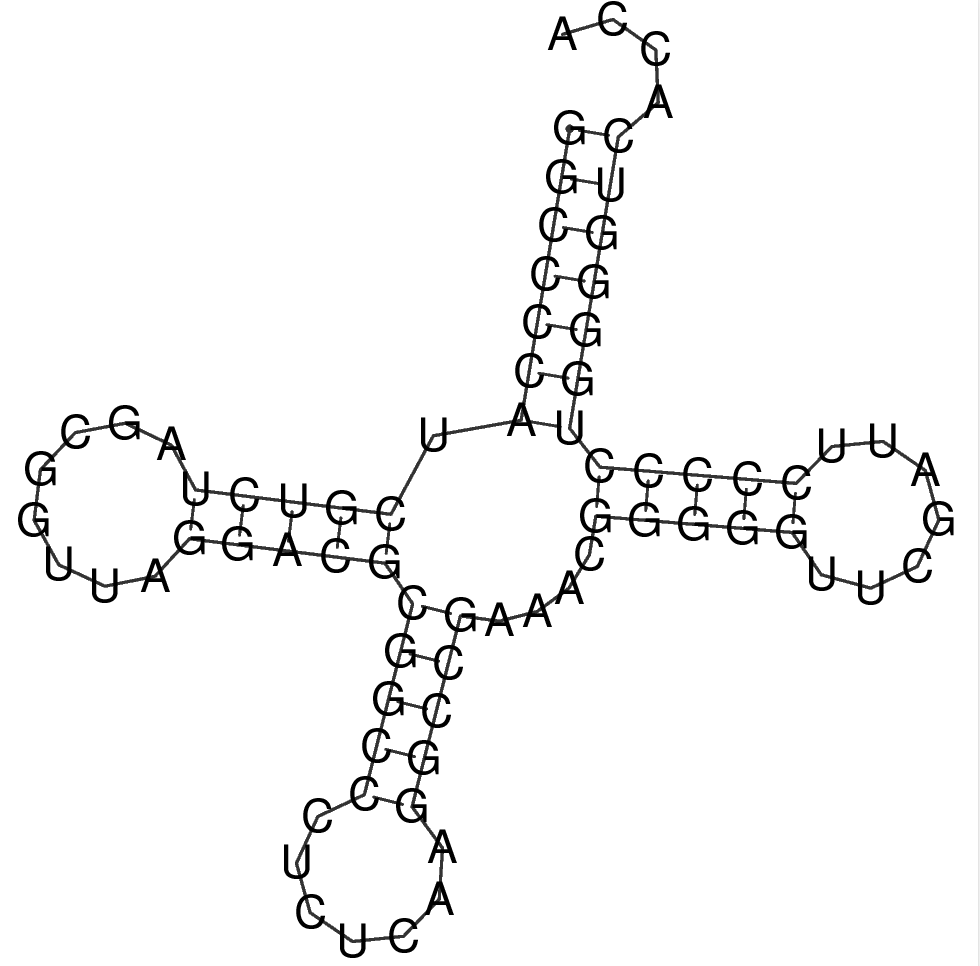
Результат работы можно наблюдать на рис.1.
Сравнение алгоритмов
| Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера | |
|---|---|---|---|
| Акцепторный стебель | 5'-503---570-3' 3'-507---566-5': 5 пар оснований | 5/5 | 5/5 |
| D-стебель | 5'-510---525-3' 3'-512--523-5': 3 пары оснований | 0/3 | 3/3 |
| T-стебель | 5'-549---565-3' 3'-553--561-5': 5 пар оснований | 0/5 | 5/5 |
| Антикодоновый стебель | 5'-539---531-3' 3'-543--527-5': 5 пар оснований | 0/5 | 5/5 |
| Общее число канонических пар нуклеотидов | 18 | 5 | 18 |
Поиск ДНК-белковых контактов в заданной структуре
2.1
Скрипт, определяющий множества атомов кислорода в сахаре, атомов фосфора в фосфатных мостиках и азота в азотистых основаниях: script1.txt
Скрипт с последующей визуализацией всех множеств в JMol: script2.txt
2.2
Полярные и неполярные взаимодействия между ДНК и белком. С помощью набора команд script3.txt было получено точное число атомов белка, образующих искомые связи:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 9 | 65 | 74 |
| остатками фосфорной кислоты | 36 | 30 | 66 |
| остатками азотистых оснований со стороны большой бороздки | 10 | 38 | 48 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 2 | 2 |
3
В результате использования программы nucplot была получена популярная схема ДНК-белковых контактов
4
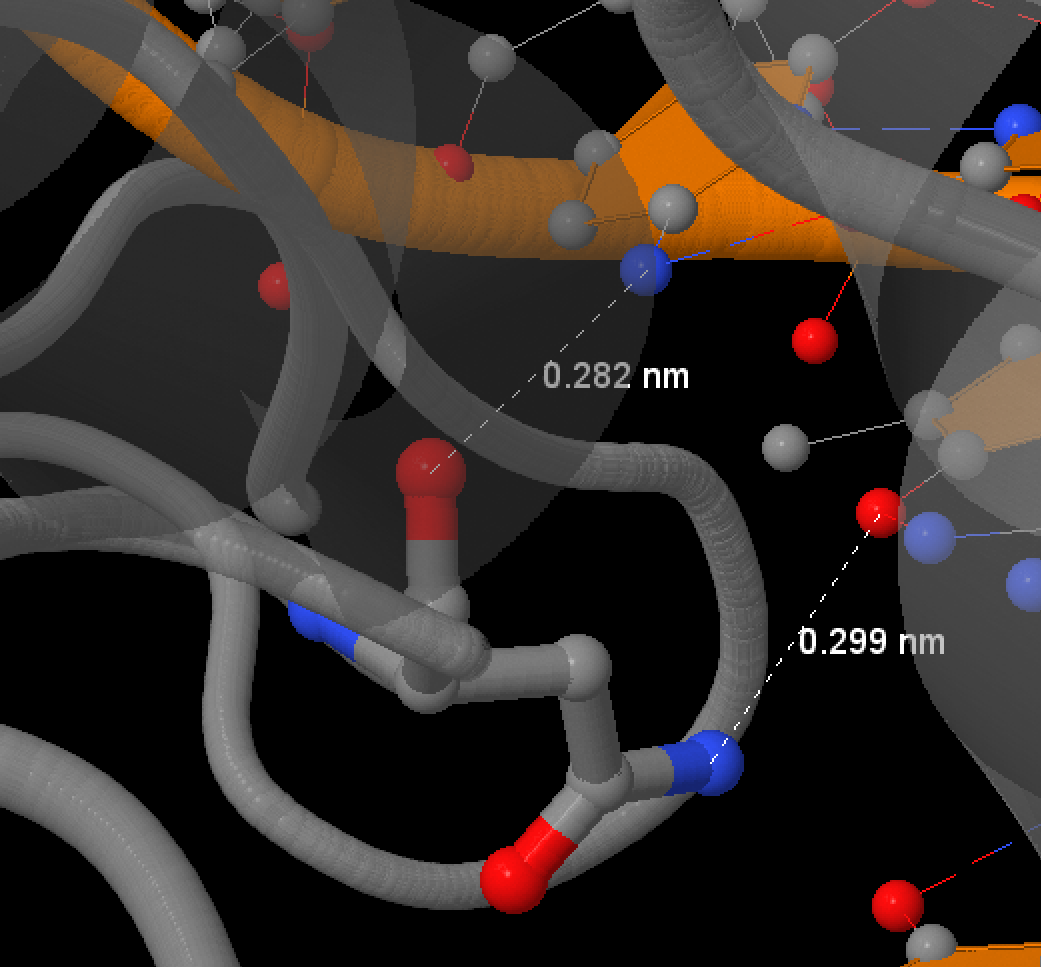
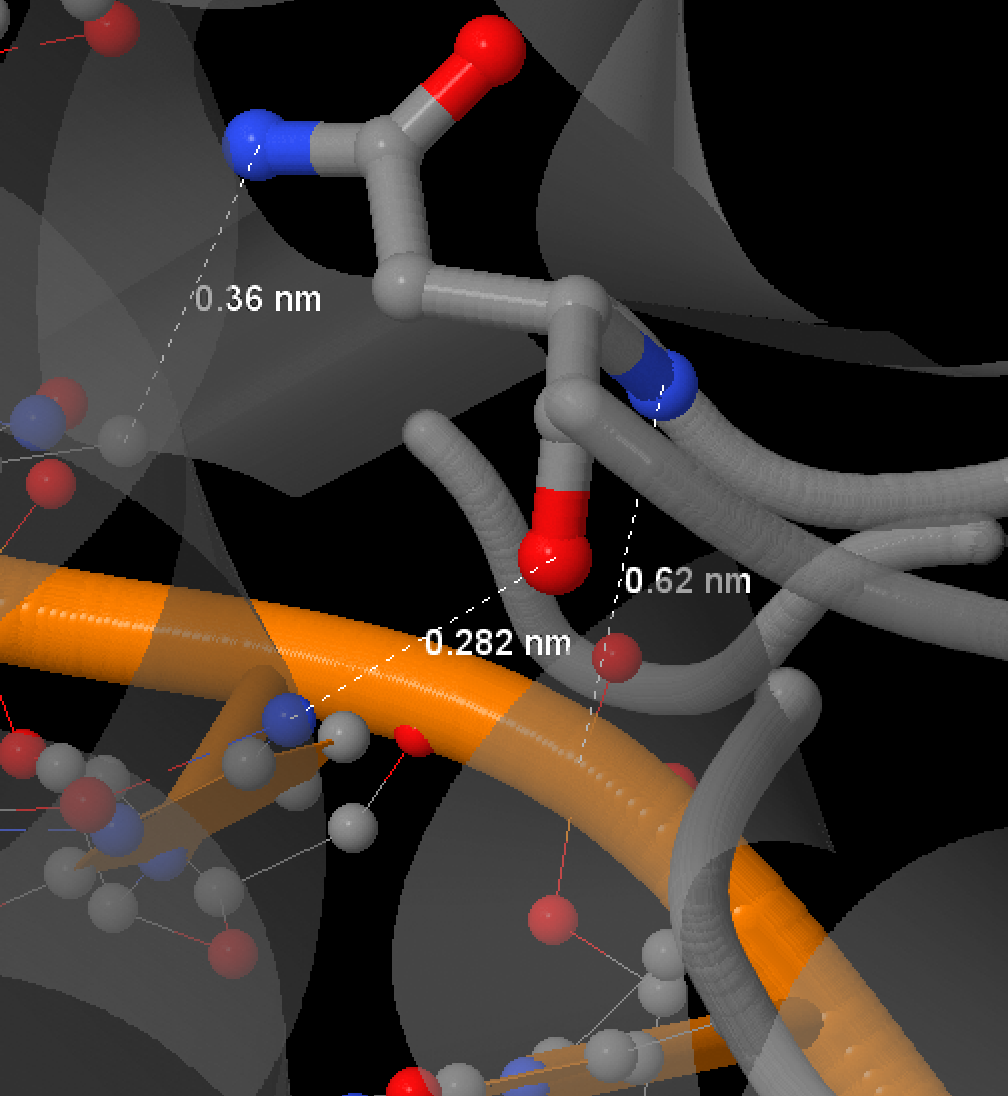
При просмотре изображения было выяснено, что аминокислота аспаргин 140 из цепи B имеет максимальное количество контактов с белком - она упоминается 5 раз. Что интересно, соответственная аминокислота из цепи A уступает лишь на один контакт и располагается симметрично. Возможно, что именно они обеспечивают упаковку ДНК в комплекс. Поэтому в качестве важной для комлекса была рассомтрена аминокислота [Asn]140:A. Изображения этих аминокислот в визуализаторе JMol представлены ниже.