Алгоритмы выравнивания
1. Сравнение выравниваний гомологичных и негомологичных белков
Ход работы:
На сайте UniProt с помощью расширенного поиска(protein name[DE], organism) были найдены интересующие меня гомологичные и негомологичные белки.
Были использованы следующие команды EMBOSS(и для гомологичных, и для негомологичных, несмотря на то, что обычно в теории к
гомологичным применяется глобальное выравнивание, а к негомологичным - локальное):
needle sw:XXX_ORGANIZM sw:YYY_ORGANISM ZZZ.needle -auto (глобальное выравнивание)
water sw:XXX_ORGANIZM sw:YYY_ORGANISM ZZZ.water -auto (локальное выравнивание)
needle sw:XXX_ORGANIZM sw:YYY_ORGANISM ZZZ.needle.msf -aformat msf -auto (в таком формате выравнивания будут открывться в Jalview)
water sw:XXX_ORGANIZM sw:YYY_ORGANISM ZZZ.water.msf -aformat msf -auto
Для заполнения отчётной таблицы из выдачи needle и water я получила следующие характеристики: процент колонок с идентичными буквами,процент колонок с
близкими буквами (для них значение матрицы замен положительно), число гэпов, вес выравнивания. Число инделей[1] мне пришлось посчитать
"глазами" по выравниванию. Их было удобно считать не из выдачи needle, а из выравнивания в Jalview.
Отчётная таблица
С помощью таблицы легко провести сравнительную характеристику парных выравниваний.
Отличия гомологичных белков (введу для них сокращение гб) от негомологичных(пусть они будут нгб), выявленные в ходе выравнивания и
заполнения таблицы:
- Вес выравнивания гб в несколько раз превышает это значение у нгб.
- Можно заметить, что процент колонок с идентичными и схожими буквами у гб, судя по полученным данным, больше 50% (у нгб, соответствеенно меньше 50%).
Интересно, что ,если применять локальное выравнивание к нгб, процент колонок с похожими А.К. у них достигает достаточно высоких значений
(до 40,5 в моём случае)
- Более того у нгб наблюдается огромное кол-во гепов (и почти всегда, как следствие, инделей) при применении выравнивания по алгоритму Нидлмана (у меня максимальное кол-во: 1238).
Однако выравнивание по алгоритму Ватермана сильно сглаживает это значение, оно становится не показательным для проверки белков на гомологию.
2.Сравнение выравниваний
Немного теории:
Парное выравнивание подразделяется на глобальное и локальное. Глобальное выравнивание обычно применяют для последовательностей, гомологичных
по всей длине. В глобальное выравнивание включаются обе входные последовательности целиком. Локальное выравнивание чаще всего применяется,
если последовательности содержат как родственные (гомологичные), так и неродственные участки.
Результатом локального выравнивания является выбор участка в каждой из последовательностей и выравнивание между этими участками.[2]
Ход работы:
Для множественого выравнивания я выбрала белок RECA.
Были использованы следующие команды EMBOSS:
infoseq 'sw:RECA_*' -only -name -out RECA.txt
seqret @SACA.txt RECA.fasta
muscle -in RECA3.fasta -out RECA_alignment.fasta
Множественное выравнивание по мнемонике RECA
 Рис.1 Ссылка [5] поможет разобраться в значении окрашивания выравнивания.
Рис.1 Ссылка [5] поможет разобраться в значении окрашивания выравнивания.
Далее из этих шести гомологичных белков я выбрала два наиболее удаленных друг от друга:RECA_MYCLE и RECA_ECOLI.
В то же окно Jalview я добавила глобальное и локальное выравнивание этих двух белков. Затем покрасила их по группам.
Сравнение выравниваний
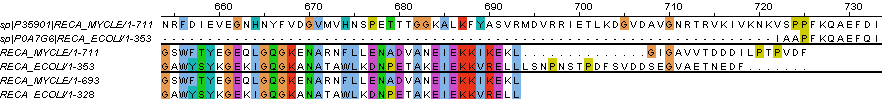
Рис.2
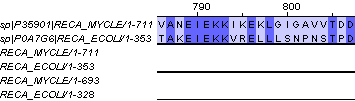
Рис.3
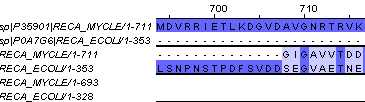
Рис.4
Отличия между парами выравниваний:
- На Рис.2 мы можем видеть что локально выравнивание прерывается на 694 позиции. Однако это не может считаться отличием между выравниваниями.
То есть они одинаковые. Неудивительно, ведь алгоритмы выравниваний needle и water похожи между собой.
- Зато несоответствия между множественным и глобальным выравниванием найти можно. Например на Рис.3 в множественном выравнивании
верхняя последовательность (позиции с 799-807) выровняна с ак: 'LSNPNSTPD',
в то время как в глобальном выравнивании (Рис.4) тот же кусочек верхней последовательносьти (позиции 708-716) сопоставлен 'SEGVAETNE'.
При этом, если обратить внимания на свойства аминокислот, то выравнивание этого участка ('GIGAVVTDD') по алгоритму needle следует признать
более удачным. Тем не менее множественное выравнивание имеет больший биологический смысл тк для него вероятность случайных совпадений меньше.
Здесь представлены ссылки, которыми я пользовалась при изучении темы: