Сигналы и мотивы 2
Задание 1: Вычисление информационного содержания (IC) последовательности Козак в геноме данио рерио и построение LOGO этого сигнала
Мне было дано выравнивание десятка контекстов ATG для десятка белков из статьи Grzegorski et al., PLoS ONE 9(9): e108475, 2014.(5 вариант)
В файле есть пояснения о том, как я вычисляла информационное содержание отдельных букв, колонок, итоговое информационное содержание сигнала.
Для построения LOGO я использовала html ресурс
В LOGO сигнала буквы имеют высоту, равную информационному содержанию букв. IC(j) = высота j-го столбца.
В построенном лого пристствуют небольшие несоответствия высот букв с вычисленными мною информационными содеражаниемя.
Например, в позиции 3 для нуклеотида С, IC которого равно 0,3323, программа не изображает символ С. В то время как в позиции 9 для нуклеотида T, IC которого равно 0,08888 (меньш, чем IC(C, 3) почти в 4 раза ), программа изображает букву T.
Проверка PWM для сайтов регуляции разрывной транскрипции sgmRNA
В предыдущем практикуме с помощью программы MEMЕ я искала сайты регуляции разрывной транскрипции в геноме Betacoronavirus HKU24. Наиболее правдоподобным мне показался мотив, представленный на картинке ниже.
Длина наиболее консервативной части найденного мотива (CS) составляет 6 нуклеотидов. Один сигнал есть в лидерной последовательности, то есть до начала гена ORF1ab полипротеина. Перед 5 из 10 поздними генами есть один сигнал. Кроме того, вероятность найти случайно мотив, обладающий перечисленными свойствами, мала (значения p-value хорошие). Таким образом, можно сказать, что данный мотив похож на идеальный, он подтвержает гипотезу о сигналах, способных регулировать синтез мРНК поздних генов.
Чтобы перепроверить полученный результат, я воспользовалась программой FIMO, которая, насколько я понимаю, делает примерно то же самое, что и MEME. Команда для запуска FIMO из консоли: fimo --norc --motif 1 meme.txt COV_HKU24.fasta Был получен тот же результат, что и при запуске MEME.
Теперь интересно посмотреть, встречается ли этот потенциальный сайт регуляции в геномах двух других короновирусов. К сожалению, геномов того же вида другого штамма в б.д. NCBI не оказалось. Поэтому было решено производить поиск по геномам близкородственных видов.
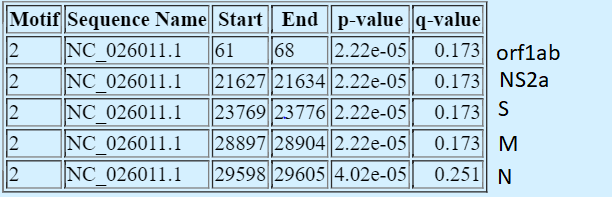
Я выбрала вирус Human coronavirus HKU1 из подрода Embecovirus, к которому принадлежит вирус Betacoronavirus HKU24. В результате запуска FIMO в его геноме был найдено 11 мотивов. На картинке ниже я отметила, где они расположены в геноме Human coronavirus HKU1. Скорее всего найденный ранее мотив является сайтом регуляции для данного вируса.
Вторым короновирусом был выбран Human coronavirus OC43 из того же подрода. Нашлось 3 мотива, первый из upstream области гена ORF1ab (см. картинку ниже), второй расположен внутри гена ORF1ab (то есть не является сайтом регуляции), третий внутри гена S (тоже не является сайтом регуляции). Результат противоречивый, вряд ли найденный мною ранее мотив регулирует разрывную транскрипцию этого вируса.
Вывод:исходя из результатов, полученных для Human coronavirus HKU1, найденный сигнал не является специфичным для Betacoronavirus HKU24.
LOGO для последовательностей Козак коронавируса.
Чтобы построить LOGO, я создала файл с последовательностями из 10 нуклеотидов для каждого из поздних гена, взятых на расстоянии +- 5 нуклеотидов от начала генов. Файл с последовательностями был загружен в MEME, программа построила LOGO, которое можно увидеть ниже.
Если сравнить его с известными LOGO для последовательностей Козак генов человека (см. картинку ниже), то окажется, что в позициях 4, 5 вируса чаще встречаются гуанин и цитозин, как и у человека.