База данных OPM
Анализ структуры иccедуемых белков
В рамках данного практикума проводилась работа по ознакомлению с интерфейсом базы данных ОРМ (Orientation of Proteins in Membranes), содержащей информацию о предположительной локализации того или иного белка относительно мембраны на примере анализа структуры двух белков.
|
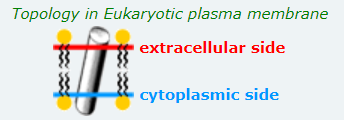
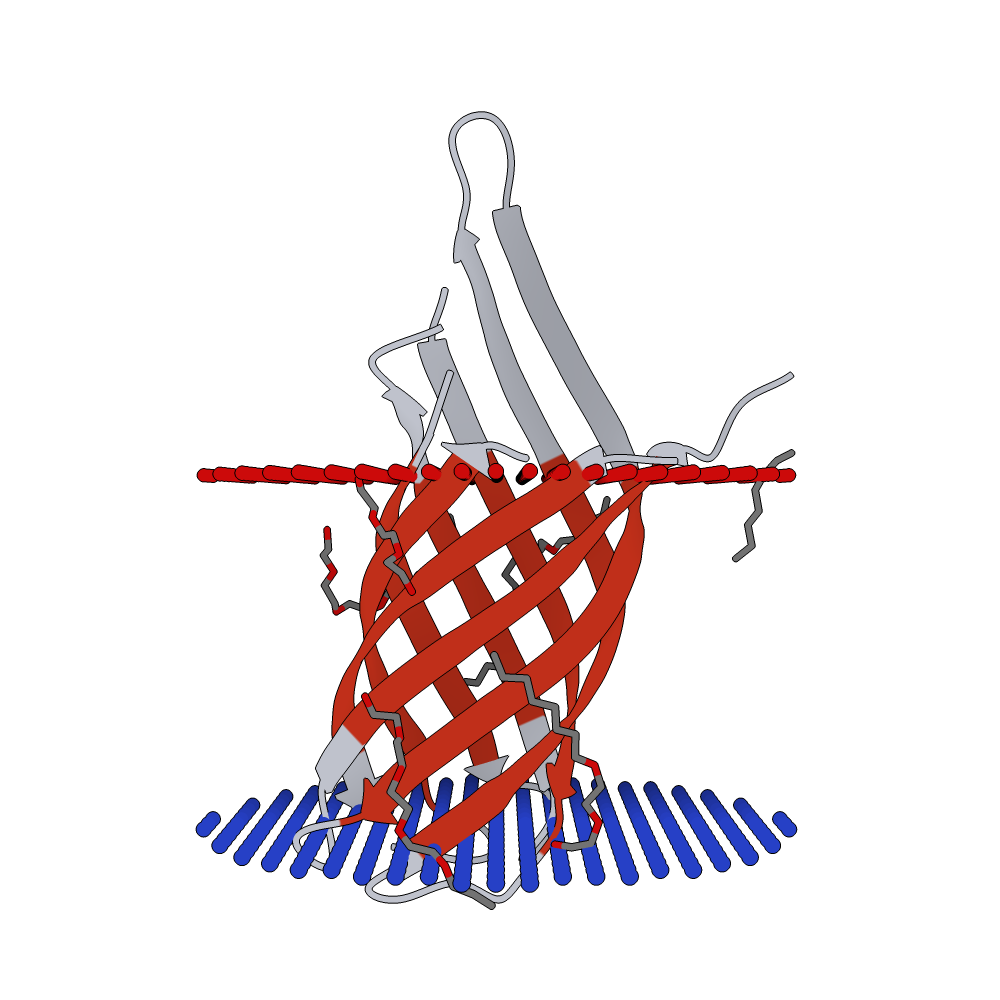
Альфа белок PDBid:4DXK (исследуется одна из его цепей). Этот человеческий трансмембранный белок относится к семейству йонных каналов, сам из себя представляет калиевый канал TREK2.
Он известен тем, что участвует в изменении возбудимости нейронов в ответ на различные стимулы [1].
Ссылка нa страницу OPM |
 Рисунок 1. Визуализация структуры обоих цепей белка TREK2 через NGL
Рисунок 1. Визуализация структуры обоих цепей белка TREK2 через NGL | |
| Таблица 1. Информация о структуре A-цепи TREK2 |
|---|
| Толщина гидрофобной части белка (ангстремы) | Координаты трансмембранных спиралей | Среднее количество остатков в одноq трансмембранной спирали | Мембрана, в которой белок локализован |
|---|
| 32.2 ± 0.7 | 75-95, 185-219, 234-260, 300-328 | 27 | плазматическая мембрана эукариот
 |
|
|
|
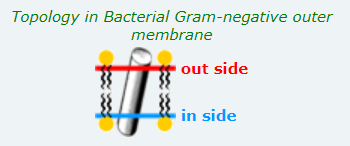
Бета-белок PDBid:4RLC. Этот белок нзывается CarO, представляет из себя кассический образчик бета-бочонка, относится к семейству поринов внешней мембраны. В его состав входит восемь бета-листов. CarO опосреует транспорт малых молекул в Gram- бактериях [2].
Ссылка нa страницу OPM |
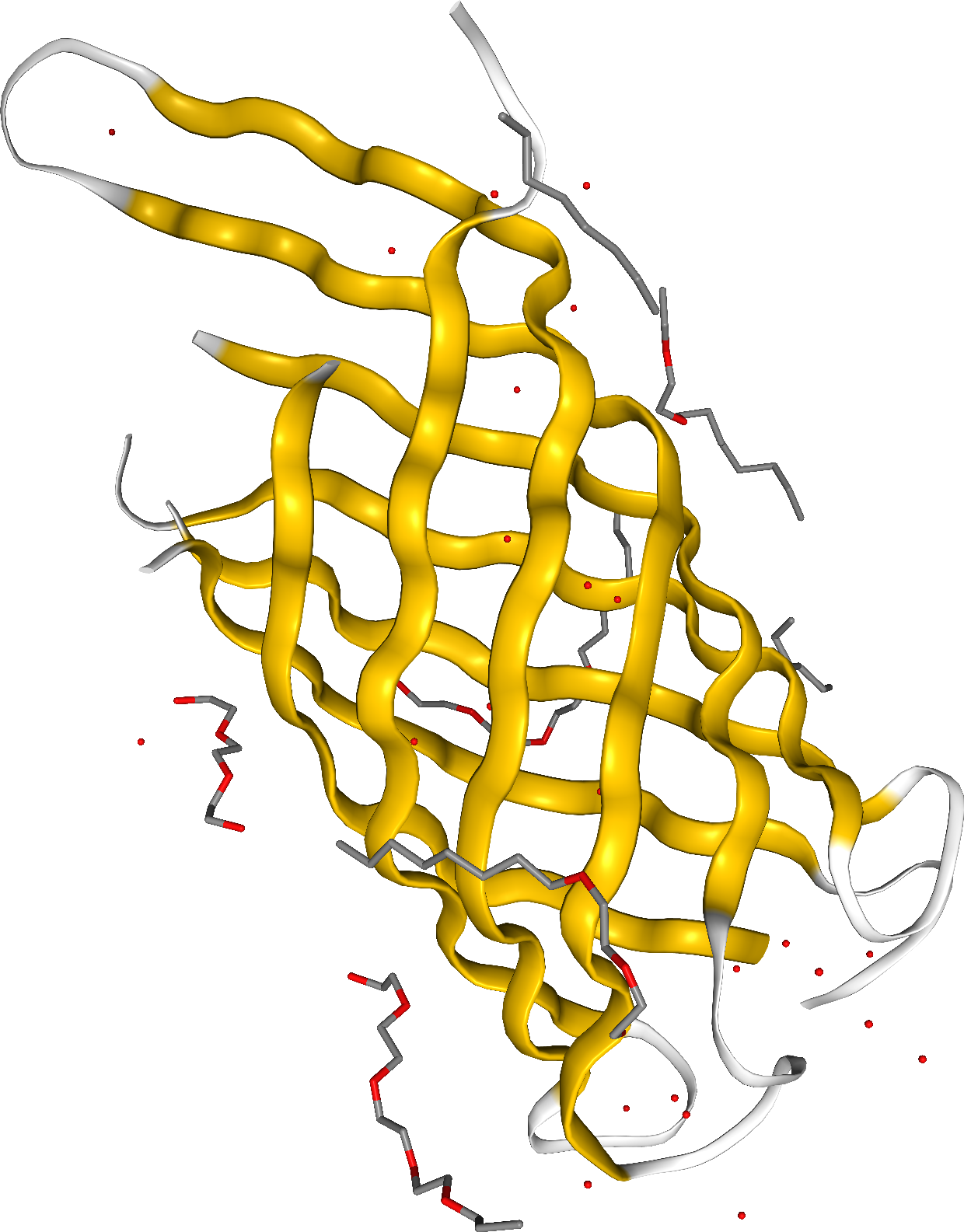 Рисунок 2. Визуализация структуры цепи белка CarO через NGL
Рисунок 2. Визуализация структуры цепи белка CarO через NGL |
| Таблица 2. Информация о структуре CarO |
|---|
| Толщина гидрофобной части белка (ангстремы) | Координаты трансмембранных спиралей | Среднее количество остатков в одном бета-тяже | Мембрана, в которой белок локализован |
|---|
| 24.2 ± 1.3 | 5-14, 28-35, 43-48, 68-77, 84-94, 112-122, 129-137, 150-158 | 9 | внешняя мембрана gram(-)бактерий
 |
|
|
Ниже представлено, как эти TREK2 и CarO располагаются относительно мембран, если p-сторона мембран смотрит вверх:
Анализ предсказания трансмембранных спиралей
Целью данного задания можно считать оценку корректности предсказания сервисов TMHMM и Phobius.
Оба сервиса работали с последовательностью fasta альфа-белка TREK2:
>4XDK:A|PDBID|CHAIN|SEQUENCE
MGLQTVMKWKTVVAIFVVVVVYLVTGGLVFRALEQPFESSQKNTIALEKAEFLRDHVCVSPQELETLIQHALDADNAGVS
PIGNSSNNSSHWDLGSAFFFAGTVITTIGYGNIAPSTEGGKIFCILYAIFGIPLFGFLLAGIGDQLGTIFGKSIARVEKV
FRKKQVSQTKIRVISTILFILAGCIVFVTIPAVIFKYIEGWTALESIYFVVVTLTTVGFGDFVAGGNAGINYREWYKPLV
WFWILVGLAYFAAVLSMIGDWLRVLSKKTKEEVGEAENLYFQ |
Ниже представлены выдачи сервисов:
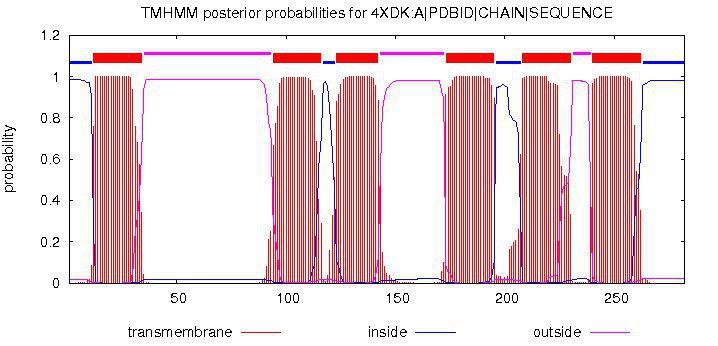 | # 4XDK:A|PDBID|CHAIN|SEQUENCE Length: 282
# 4XDK:A|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 6
# 4XDK:A|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 129.78283
# 4XDK:A|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 21.20494
# 4XDK:A|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.98489
# 4XDK:A|PDBID|CHAIN|SEQUENCE POSSIBLE N-term signal sequence
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 1 11
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 12 34
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 35 93
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 94 116
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 117 122
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 123 142
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 143 172
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 173 195
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 196 207
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 208 230
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 231 239
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 240 262
4XDK:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 263 282 |
Рисунок 3. Выдача TMHMM. Предсказание трансмембранных спиралей. Горизонтальная ось: последовательность белка, вертикальная ось: вероятность того, что данный аминокислотный остаток расположен внутри мембраны, в наружной среде или в цитоплазме. Красные линии - трансмембранная часть белка, синие линии - часть белка внутри клетки, розовые линии - часть белка вне мембраны.
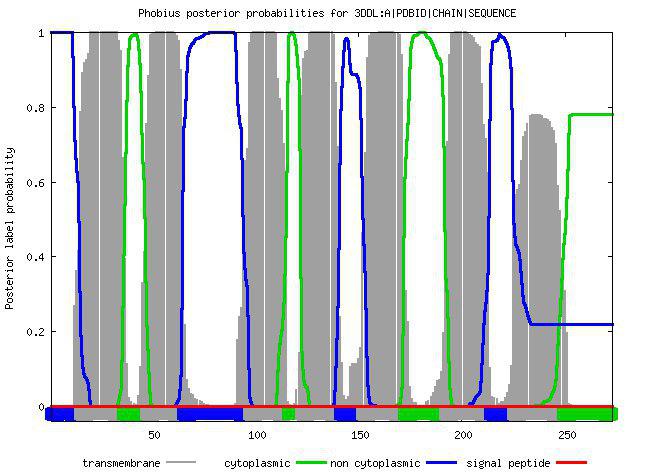 | Prediction of 3DDL:A|PDBID|CHAIN|SEQUENCE
ID 3DDL:A|PDBID|CHAIN|SEQUENCE
FT TOPO_DOM 1 14 NON CYTOPLASMIC.
FT TRANSMEM 15 35
FT TOPO_DOM 36 46 CYTOPLASMIC.
FT TRANSMEM 47 64
FT TOPO_DOM 65 96 NON CYTOPLASMIC.
FT TRANSMEM 97 115
FT TOPO_DOM 116 121 CYTOPLASMIC.
FT TRANSMEM 122 140
FT TOPO_DOM 141 151 NON CYTOPLASMIC.
FT TRANSMEM 152 171
FT TOPO_DOM 172 191 CYTOPLASMIC.
FT TRANSMEM 192 213
FT TOPO_DOM 214 224 NON CYTOPLASMIC.
FT TRANSMEM 225 248
FT TOPO_DOM 249 273 CYTOPLASMIC. |
Рисунок 4. Выдача Phobius. Предсказание трансмембранных спиралей.
Горизонтальная ось: последовательность белка, вертикальная ось: вероятность того, что аминокислотный остаток расположен внутри мембраны, в наружной среде или в цитоплазме.
Серые линии - трансмембранная часть белка, зеленая линия - часть белка внутри клетки, синая линия - часть белка, расположенная вне клетки
На основе полученных данных была составлена сводная таблица:
| Таблица 3. Сравнения предсказаний трансмембранных доменов |
|---|
| OPM: |
(75-95),(185-219),(234-260),(300-328) |
| TMHMM: |
(12-34),(94-116),(123-142),(173-195),(208-230),(240-262) |
|---|
| Phobius: |
(15-35),(47-64),(97-115),(152-171),(192-213),(225-248) |
Как можно наблюдать из выдачи, найденное количество доменов в случае TMHMM и Phobius одинаково, но не совпадает с количеством доменов по данным ОРМ, и координаты их сильно рознятся. Сервисы, использумые в этой работе, не справились с адекватным предсказанием и не предоставили картинку, сопоставимую с ОРМ, то есть доверять им мы не можем в данном случае.
Сравнение методов TMHMM и Phobius
У двух этих методов предсказания трансмембранных участком есть как много общего, так и значительные различия.
Суть TMHMM в использование метода скрытых марковских моделей - статистическая модель, имитирующая работу процесса, похожего на марковский процесс с неизвестными параметрами, и задачей ставится разгадывание неизвестных параметров на основе наблюдаемых. Полученные параметры могут быть использованы в дальнейшем анализе, например, для распознавания образов. По результатам (имеющейся цепи) он строит прогнозирование [3].
Суть Phobius в комбинации выдачи алгоритмов TMHMM и SignalP. SignalP прогнозирует появление сайтов расщепления в данном белке. В таком случае итоговая выдача состоит не только из трансмембранных петель, но и из сигнальных пептидов и промежуточных петель. SignalP используя модель Маркова с тремя переменными- регион вокруг сайта, гидрофобный регион и N-концевой домен, и на основе этих данных модель предполагает, есть ли внутри cleavage-сайты (сайты расщипления) [4]. Главным является тот факт, что Phobius, помимо всего прочего, находит в данном белке ещё и сигнальные пептиды.
Это приводит к значительно схожим выводам двух алгоритмов - так как один заимствует у другого.
И, несмотря на большое сходство методов TMHMM и Phobius, Phobius представляет более полный объем данных, что делает его использование более целесообразным
Анализ структуры иccедуемых белков
В данной части работы необходимо было найти в БД TCBD(Transporter classification database) белки, исследуемые в первом задании. Для альфа-белка ничего не было обнаружено, для бета-бочонка (4rlc) был идентифицирован TC код 1.B.6.1.2:
- 1. Channels/Pores = Каналы/поры)
.B. β-Barrel Porins = β-слойные порины
- 1.B.6. The OmpA-OmpF Porin (OOP) Family = Семейство OmpA-OmpF поринов
- 1.B.6.1.2 OmpF(OprF) porin [om for outer membrane] = OmpF(OprF) порин - порин внешней мембраны
| Таблица 4. Термины GO, относимые к белку TREK2 |
|---|
| Аспект |
Идентификатор GO |
Название термина |
Перевод названия термина |
Код типа достоверности |
| Биологический процесс/Biological process) |
GO:0005980 |
glycogen catabolic process |
катаболический процесс гликогена |
ISS |
| Молекулярная функция/Molecular function |
GO:0007613 |
glucanotransferase activity |
активность глюканотрансферазы |
ISS |
| Биологический процесс/Biological process |
GO:0004134 |
memory |
память |
IEA |
| Биологический процесс/Biological process |
GO:0071805 |
potassium ion transmembrane transport |
трансмембранный перенос калия |
IEA |
| Биологический процесс/Biological process |
GO:0034765 |
regulation of ion transmembrane transport |
регулирование ионно-трансмембранного транспорта |
IEA |
| Биологический процесс/Biological process |
GO:0007165 |
signal transduction |
трансдукция сигнала |
NAS |
| Биологический процесс/Biological process |
GO:0030322 |
stabilization of membrane potential |
стабилизация мембранного потенциала |
IBA |
| Биологический процесс/Biological process |
GO:0006810 |
transport |
транспорт |
TAS |
| Клеточный компонент/Сellular component |
GO:0005887 |
integral component of plasma membrane |
встроенный компонент плазматической мембраны |
IBA |
| Клеточный компонент/Сellular component |
GO:0005886 |
plasma membrane |
плазматическая мембрана |
TAS |
| Молекулярная функция/Molecular function |
GO:0005267 |
potassium channel activity |
активность калиевого канала |
TAS |
| Молекулярная функция/Molecular function |
GO:0022841 |
potassium ion leak channel activity |
активность канала утечки ионов калия |
IBA |
| Молекулярная функция/Molecular function |
GO:0005244 |
voltage-gated ion channel activity |
активность потенциалзависимых ионных каналов |
IEA |
| Таблица 5. Термины GO, относимые к белку CarO |
|---|
| Аспект |
Идентификатор GO |
Название термина |
Перевод названия термина |
Код типа достоверности |
| Биологический процесс/Biological process) |
GO:0008150 |
- |
- |
ND |
| Молекулярная функция/Molecular function |
GO:0003674 |
- |
- |
ND |
Тип кода достоверности ND используется для аннотаций, когда информация о молекулярной функции, биологическом процессе или клеточном компоненте аннотированного гена или генного продукта недоступна.
Использование кода ND указывает на то, что при поиске найти информацию, которая поддерживает создание аннотации к любому термину из Молекулярной функции, биологического процесса или клеточного компонента, не удалось. Остальные коды представлены ниже:
| Таблица 6. Расшифровка кодов достоверности |
|---|
Код типа достоверности |
Расшифровка кода достоверности |
Объяснение |
| ISS |
Inferred from Sequence or structural Similarity |
Буквально "определимая из последовательности или структурного сходства"
Этот код используется всякий раз, когда основанный на последовательности анализ формирует основу для аннотации. ISS следует использовать, если используется комбинация инструментов или методов для определения последовательности, которые дают какие-то доказательства.
На практике аннотации ISS редко, если вообще когда-либо, сделаны исключительно из структурной информации, структурная информация обычно находится на уровне моделирования вторичной структуры или прогнозирования, полученного из информации о последовательности. |
| IBA |
Inferred from Biological aspect of Ancestor |
Дословный перевод "вывод сделан из биологического аспекта предка"
IBA - это тип филогенетических доказательств того, что аспект потомка выведен из-за характеристики аспект гена предков. Вкратце, принципы, лежащие в основе филогенетической аннотации, опираются на филогенетические анализы для передачи аннотаций среди связанных последовательностей на основе общей генерации.
Дополнительная информация может быть найдена в распространении функциональных аннотаций на основе филогенетических веществ в консорциуме генов-онтологов Gaudet et al., 2011. |
| NAS |
Non-traceable Author Statement |
"Неотслеживаемое авторское завление"
Код доказательств NAS должен использоваться в следующих случаях:
1)Когда автор делает заявление, для которого нет ни представленных результатов, ни конкретной ссылки, приведенной в источнике, используемом для создания аннотации.
2)Запись базы данных, которая не цитирует документ (например, запись базы знаний UniProt, отчеты о белке YPD)
3)Заявление в статье (аннотация, введение или обсуждение), которое не ведёт к другой публикации. |
| IEA |
Traceable Author Statement |
Выведено из электронной анотации".
Используется для аннотаций, которые напрямую зависят от вычисления или автоматической передачи аннотаций из базы данных, особенно если анализ выполняется внутренне и не публикуется. Ключевой особенностью, которая отличает этот код доказательств от других, является то, что он сделан не вручную . Используется, когда конкретная аннотация не была проверенна, для анализа точность |
| TAS |
Inferred from Electronic Annotation |
"Отслеживаемое авторское заявление.
Код доказательства TAS охватывает авторские утверждения, которые относятся к цитируемой ссылке, которая является источником первоначального доказательства (экспериментальные результаты, сравнение последовательностей и т. д.). Как правило, аннотации TAS взяты из обзорных статей. Заявления из разделов введения и обсуждения статей первичного исследования также могут быть подходящими, если в качестве источника экспериментальной работы или анализа указывается другая ссылка. |
Отнесение к COG
Как и следовало ожидать, COG для первого альфа-белка мы вновь не нашли:

Для бета-бочонка COG был найден:
| Таблица 7.COG 4RCL (CarO) |
|---|
| Идентификатор COG | COG2885 |
| E-value COG по CDD | 5.55e-48 |
| Интрвал, занятый COG в белке (координаты аминокислотных остатков) | 139-345 |
| Общее число аминокислотных остатков в белке | 350 |
| Название COG/ COG name | OmpA |
| Описание | Outer membrane protein OmpA and related peptidoglycan-associated (lipo)proteins [Cell wall/membrane/envelope biogenesis]/Протеин наружной мембраны OmpA и связанные с ним пептидогликан-ассоциированные (липо)белки [Биогенез клеточной стенки/мембраны/оболчки] |
| Функциональная категория COG/ Functional COG category | M Outer membrane protein OmpA and related peptidoglycan-associated (lipo)proteins/M белок OmbA с наружной мембраной и связанные с ним пептидогликан-ассоциированные (липо)белки |
Визуализация геномного окружения исследуемого COG
Ниже представлено изображение геномного окружения для обнаруженного COG с помощью COGNAT:
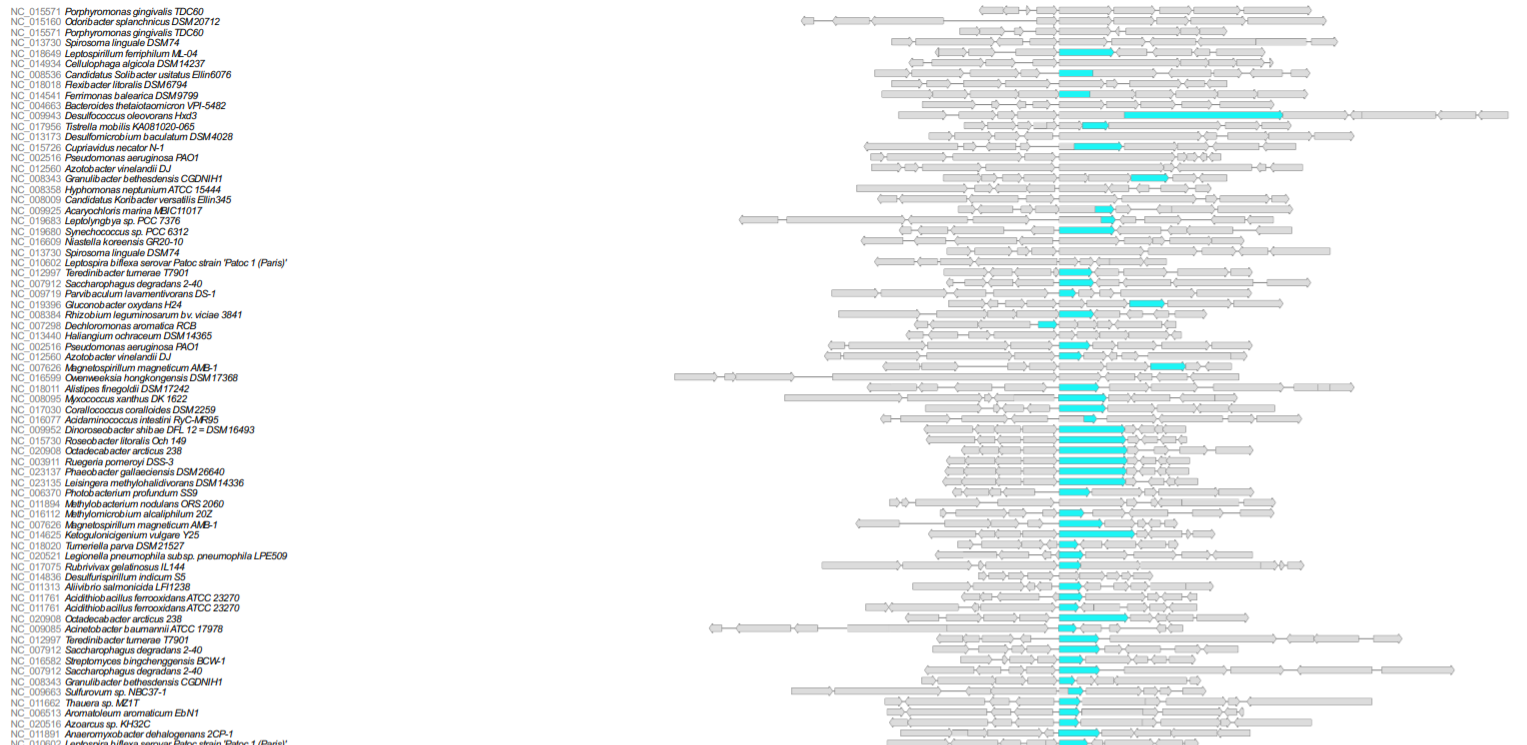 Рисунок 5. Геномное окружение COG2885
Рисунок 5. Геномное окружение COG2885
Параметры, введённые на инпут программы:
-
Search for COG: COG2885
- Neighborhood Size: 9
- Occurrence Threshold: 20%
- Taxonomy(по-дефолту): Нет
Полная выдача представлена в формате pdf: cognat.pdf
На рисунке Списком слева представлены организмы, в которых рассматривается геномное окружение COG. Первыми, серым шрифтом, представлены их идентификаторы в базе данных NCBI, после - видовое наименование(периодически с точностью до штамма). Справа схметично изображены гены - одна стрелка = один ген. Показываются все гены, которые отнесены к выбранному COG в 711 прокариотических геномах и их геномное окружение.
Для изучаемого COG поиск проводился в интервале 9 белков - 4 справа и 4 слева.
Зелёным цветом отмечены гены, встречающиеся более чем в 20% случаев (задано через Threshold), находки не сортировались по таксономии.
Консервативное окружение в данном случае, к сожалению, не наблюдается, что можно легко пронаблюдать на картинке.
Ссылки на ресурсы:
Вернуться назад
На главную страницу
©Solonovich Vera,2017





