Целью данной работой была реконструкция эволюции доменной архитектуры белков, содержащих один и тот же домен Pfam. Для реализации проекта был выбран один из доменов изучаемого в первом семестре белка 4-альфа-глюкотрансферазы. Список доменов данного белка представлен на странице проекта EMBL-EBI, подходящий кандидат выбран по заявленным критериям (в семействе Pfam не более десяти тысяч последовательностей не более чем 1200 видов, домены семейства входят несколько доменных архитектур, для тех известно несколько 3D структур).
Таблица 1. Информация о домене | |||
|---|---|---|---|
| AC, ID | hGDE_central, PF14702 | ||
| Функция домена | |||
| Это центральный домен эукариотического варианта фермента, уменьшающего ветвистость гликогена (glycogen debranching enzyme - GDE). Эукариотический GDE выполняет две функции: 4-альфа-D-глюканотрансферазную и амило-альфа-1,6-глюкозидазную, выполняемую соответственно N- и C-концевыми доменами эукариотического фермента GDE. Центральный домен GDE следует за глюканотрансферазным доменом (GDE_C) и предшествует глюкозидазному (GDE_N). По аналогии с бактериальными GDE, этот домен должен быть вовлечен в субстратное связывание либо для N-концевой глюканотрансферазы, либо для C-концевой глюкозидазы, если не для той или другой разом. | |||
| Количество архитектур | Количество последовательностей | Количество видов | Количество визуализированных структур |
| 43 | 717 | 605 | 4 |
| http://pfam.xfam.org/family/PF14702 | |||
Список сорока трёх доменных архитектур данного домена так же представлен по соответствующей ссылке:
http://pfam.xfam.org/family/PF14702#tabview=tab1
Скаченные из Pfam выравнивания PF14702_full.txt были заружены в JalView и покрашены по BLOSUM62 c порогом консервативности 10. Выравнивание доступно по ссылке/.
Впоследствии была добавлена 3D структура белка 5D06 = Q6FSK0_CANGA остатков 727 - 967. При просмотре в JAlView, PDB id доступен по череде ссылок 'щёлкнуть ID' -> 3D Structure Data в таблице вида Таблица 2 во втором столбце:
| Таблица 2.Found structures summary | ||||||
|---|---|---|---|---|---|---|
| Q6FSK0_CANGA | 5d06 | Crystal Structure of the Candida Glabrata Glycogen Debranching Enzyme (E564Q) in complex with maltopentaose | X-ray diffraction | 3.3 | PF14702, PF14699, PF06202, PF14701 | hGDE_central, hGDE_N, GDE_C, hDGE_amylase |
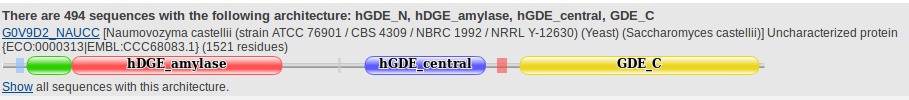
Список всех архитектур представлен на соответствующей странице EMBL-EBI. Всего архитектур, насчитывающих более 20 последовательностей, оказалось четыре. Самая полная архитектура насчитывает 494 последовательности и представляет из себя 4 домена, все вышеперечисленные в функции фермента, поэтому брать мы его не будем - это не интересно.

Ниже - визуализация рассмотренных архитектур. В первой присутствуют амилазный, центральный и с-концевой домены из пятидесяти двух структур, во второй - исключительно неполный фрагмент центрального домена, встречающийся в двадцати структурах.

C помощью скрипта swisspfam-to-xls.py расположенном на сервере в открытом доступе, информация о доменах в последовательностях из файла swisspfam.gz, загруженном на kodomo, была транспонирована в таблицу Excel по идентификатору Pfam AC - PF14702. Ниже представлена команда:
python swisspfam-to-xls.py -i /srv/databases/pfam/swisspfam.gz -z -p PF14702 -o PF14702.xls |
C применением фильтров были выделены AC последовательностей, содержащих данные доменные архитектуры - доменов насчитывается 717, притом, что последовательностей оказалось всего 700. Для каждого из 700 AC из базы Uniprot по ID была скачена с помощью следующего скрипта и обработана в таблице Excel информация о белках:
python uniprot-to-taxonomy.py -i uniprot.txt -o taxonomy.xls |
Из сводной таблицы была составлена таблица, как уже было сказано выше, содержащая AC последовательностей в изучаемых доменных архитектурах. Там же содержится информация по такосномии и длине белковых подуктов. Среди данных представителей были выбраны последовательности, относящиеся к таксону Eukaryota - к сожалению, из нижележащих таксонов более 20 последовательностей имело только Metazoa, потому к двум изученным архитектурам была добавлена первая - самая полная, с наибольшим количеством последовательностей(494). В третью архитектуру включены N-концевой домен, амилаза, центральный домен и С-концевой. О них было рассказано в ранее - PF14699, PF14701, PF14702 B PF06202, соответственно. Таксоном обозначили всё те же Eukaryota, подтаксонами - Fungi и Metazoa. Eukaryota - царство ядерных организмов, стоящее в одной линейке с археями и бактериями, подтаксонами - Fungi и Metazoa, настоящие грибы и настоящие многоклеточные. Выбрали по 20 представителей каждой из архитектур; для каждого подтаксона наличиствует не менее 10 представителей каждой архитектуры. В таблице выбранные последовательности на последнем листе покрашены в красный и вынесены на отдельный лист - chosen_tab. Последовательность, имеющая 3D визуализацию, тоже была добавлена в выборку. Ниже приведены данные про неё, взятые из самой полной таблицы про эту последовательность:
| Таблица 3. Информация по невошедшей последовательности | |||||||
|---|---|---|---|---|---|---|---|
| Sequence_ID | Sequence_AC | Sequence_length | Pfam_AC | From | To | Pfam_seq_num | Description |
| Q6FSK0_CANGA | Q6FSK0 | 1528 | PF06202 | 1037 | 1517 | 1981 | Amylo-alpha-1,6-glucosidase |
Из исходного выравнивания были получены выбранные последовательности с помощью следующей команды, скрипта на kodomo в общем доступе, и файла, содержащего AC из листа chosen_tab:
python filter-alignment.py -i phah.mfa -m chosen.txt -o phah_selс.mfa -a "_" |
Из проекта был получен fasta-файл, после обработки названий python-скриптом, мнемоника каждого на дереве представляет из себя выражение:
[13]_[MF]_AC |
Рисунок филогенетического дерева домена представлен на изображении ниже. Сконструировано дерево с помощью алгоритма Neighbour-Joining, с проверкой bootstrap на 100 копий. Ветви раскрашены по принадлежности к архитектуре и к таксону, таксоны так же обозначены. Далее следует изображение bootstrap-дерева, которое ставит вопрос о валидности нашего дерева.
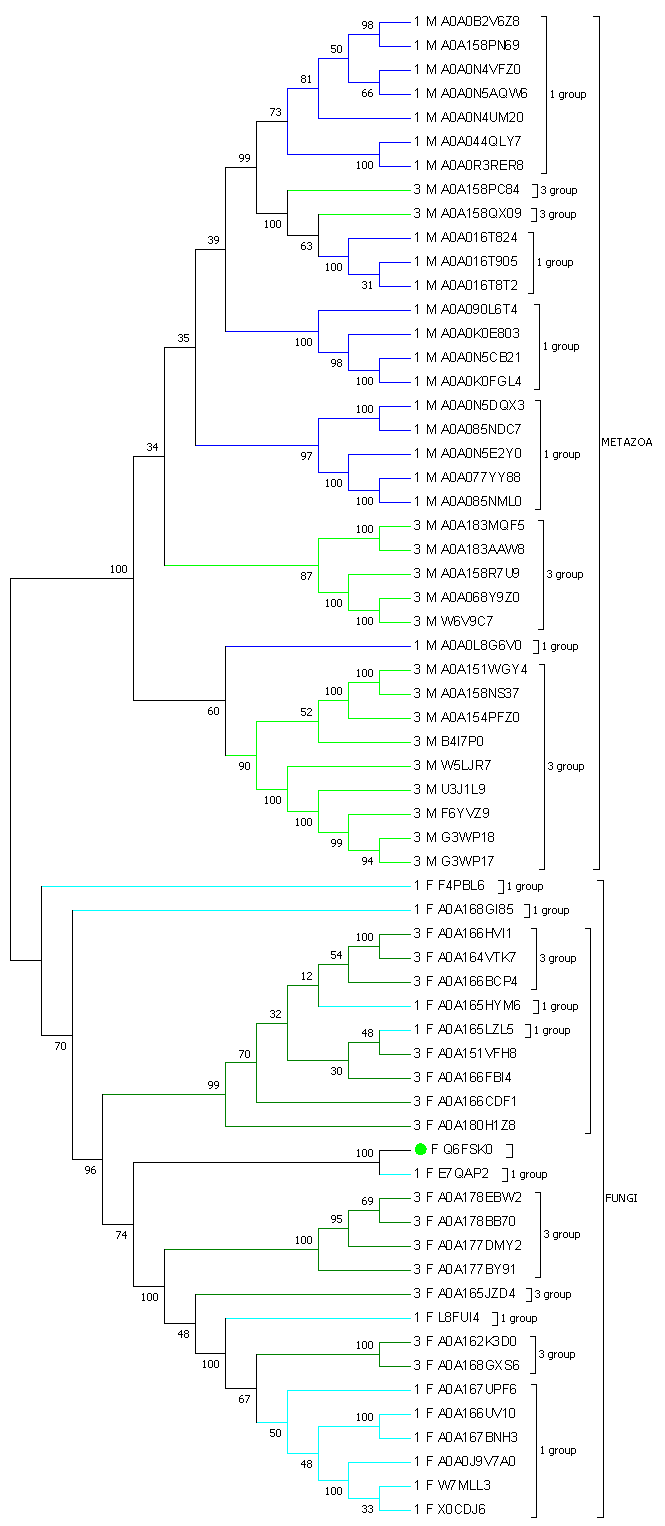 |
(((((((((1_M_A0A0B2V6Z8,1_M_A0A158PN69),(1_M_A0A0N4VFZ0,1_M_A0A0N5AQW6)),1_M_A0A0N4UM20),(1_M_A0A044QLY7,1_M_ A0A0R3RER8)),(3_M_A0A158PC84,(3_M_A0A158QX09,(1_M_A0A016T824,(1_M_A0A016T905,1_M_A0A016T8T2))))),(1_M_A0A090L6T4,(1_M_A0A0K0E803,(1_M_A0A0N5CB21,1_M_A0A0K0FGL4)))),((1_M_A0A0N 5DQX3,1_M_A0A085NDC7),(1_M_A0A0N5E2Y0,(1_M_A0A077YY88,1_M_A0A085NML0)))),((3_M_A0A183MQF5,3_M_A0A183AAW8),(3_M_A0A158R7U9,(3_M_A0A068Y9Z0,3_M_W6V9C7)))),(1_M_A0A0L8G6V0,((((3_ M_A0A151WGY4,3_M_A0A158NS37),3_M_A0A154PFZ0),3_M_B4I7P0),(3_M_W5LJR7,(3_M_U3J1L9,(3_M_F6YVZ9,(3_M_G3WP18,3_M_G3WP17)))))),(1_F_F4PBL6,(1_F_A0A168GI85,(((((((3_F_A0A166HVI1,3_F _A0A164VTK7),3_F_A0A166BCP4),1_F_A0A165HYM6),(3_F_A0A166FBI4,(1_F_A0A165LZL5,3_F_A0A151VFH8))),3_F_A0A166CDF1),3_F_A0A180H1Z8),((F_Q6FSK0,1_F_E7QAP2),((((3_F_A0A178EBW2,3_F_A0 A178BB70),3_F_A0A177DMY2),3_F_A0A177BY91),(3_F_A0A165JZD4,(1_F_L8FUI4,((3_F_A0A162K3D0,3_F_A0A168GXS6),(1_F_A0A167UPF6,((1_F_A0A166UV10,1_F_A0A167BNH3),(1_F_A0A0J9V7A0,(1_F_W7 MLL3,1_F_X0CDJ6))))))))))))); |
| Перестройки доменной архитектуры |
|
Эволюция доменных архитектур представляется нам, по итогам исследования, следующим образом: На нескольких ветвях происходили вставки или делеции доменов, организующих архитектуры. В результате, по ходу эволюции, какие-либо части последовательности, ответственные за связыание и работу глюкозидазы и глюкотрансферазы, были утрачены или приобретены. Если обращаться к частным примерам, то, например, в кладе Metazoa по узлам расхождение верхней группы (с первой доменной архитектурой) и второй группы началось с приобретения нового домена (третья архитектура подразумевает N-концевого домена), но через два ответвления снова происходит потеря N-концевого домена, и последовательности возвращаются к первой архитектуре. Выше, по ссылке в заголовке, приведено изображение, в котором по предположению, что изначально N-концевого домена не существовало, зелёным показаны места возникновения домена, синим - потери его. |
На главную страницуВернуться назад
©Solonovich Vera,2017