-
Всего 647 283 находки.
Счёт более 40 имеют 41 743 находки
более 50 - 2 594
более 60 - 603
Счёт 100 имеют 12 находок
-
Составила сводную таблицу - количество строк уменьшилось. Примерно в 400 белках было найдено по два участка.
Файл Excel со сводной таблицей
-
Характеритстики списка найденых белков (все белки):
Число верных находок ("True positive hits", TP) - 58
Число ложных находок ("False positive hits", FP) - 10685
Число ненайденных белков подсемейства ("False negatives", FN) - 0
Чувствительность TP/(TP+FN) - 1
Селективность TP/(TP+FP) - 0,0054
Надо заметить, что кроме рибосомального белка S11, нашлось множество других белков. Я посчитала характеристики списка только 690 белков RS11_?????.
Характеритстики списка найденых белков (только S11):
Число верных находок ("True positive hits", TP) - 58
Число ложных находок ("False positive hits", FP) - 632
Число ненайденных белков подсемейства ("False negatives", FN) - 0
Чувствительность TP/(TP+FN) - 1
Селективность TP/(TP+FP) - 0,084
-
У созданнного на прошлом занятии паттерна селективность и чувствительность равнялись 1. Чтобы добиться такого же значения селективности у профиля
, необходимо поставить порог 93. При таком пороге значение чувствительности у профиля (как и для всех белков, так и для S11)- 0,89.
-
ROC-кривая:
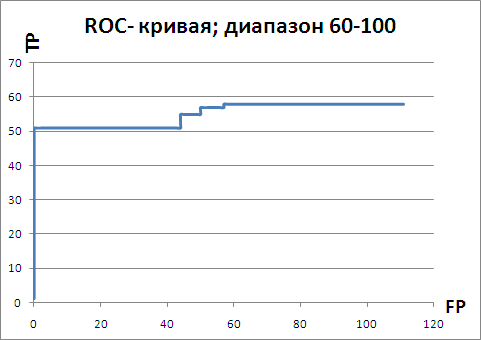
© Медведева
Далее я ограничилась находками, со счётом более 45 (10743 находки)