 -- 646 сруктур и
-- 646 сруктур и  -- 511 структур
-- 511 структурВ общем: 126 среди эукариот
897 среди бактерий
1 среди вирусов
8 из архей
Филогенетическое дерево:
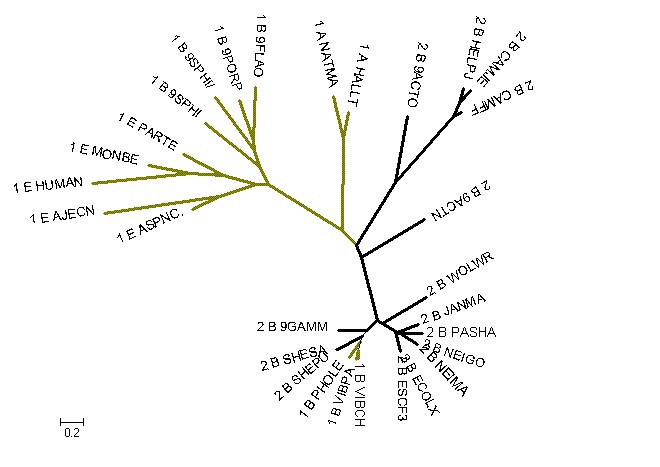
Оливковый цвет - группа (А), чёрный - (А+В).
Скобочная формула:
(1_B_VIBPA:0.07914,(1_B_PHOLE:0.16681,((2_B_SHEPU:0.00695, 2_B_SHESA:0.00798):0.23959,(2_B_9GAMM:0.24727,(((((((1_E_ASPNC.:0.25530, 1_E_AJECN:1.00019):0.33058,((1_E_HUMAN:0.84220,1_E_MONBE:0.36931):0.28303, 1_E_PARTE:0.38677):0.29216):0.10801,(1_B_9SPHI:0.59234,(1_B_9SPHI/:0.56468, (1_B_9PORP:0.36791,1_B_9FLAO:0.46052):0.08180):0.14461):0.17819):0.74438, (1_A_NATMA:0.36512,1_A_HALLT:0.22140):0.79684):0.17580,(2_B_9ACTO:0.58216, ((2_B_HELPJ:0.13149,2_B_CAMJE:0.14619):0.12522,2_B_CAMFF:0.07623):0.75868):0.63298):0.10926, 2_B_9ACTN:0.62805):0.56291,(2_B_WOLWR:0.43873,((2_B_JANMA:0.18909, (2_B_PASHA:0.18688,(2_B_NEIGO:0.00528,2_B_NEIMA:0.00083):0.16585):0.02955):0.01905, (2_B_ECOLX:0.00000,2_B_ESCF3:0.00000):0.17715):0.13480):0.04800):0.12198):0.06497):0.05090):0.08022,1_B_VIBCH:0.06912);
Мысли выводы и замечания:
С выбором последовательностей возникла проблема - оказалось, что архитектура (А+В) встречается исключительно у бактерий. Поэтому я постаралась взять больше бактерий и для второй архитектуры (A).
Одну последовательность (A0M1H1_GRAFK) пришлось исключить из выравнивания.
На картинке в Pfam для (А+В) идёт сначала Pro_dh, а затем Aldedh. Однако, на выравнивании общий домен для второй группы лежит перед доменом Pro_dh.
Нехорошо, что 4 бактерии из группы 1 оказались в ветке с эукариотами и археямм. Наверное, это результат плохого выравнивания. По построенному дереву нельзя установить родственную связь между бактериями из разных групп. Тем не менее, по нему видно, что бактерии сначала приобрели структуру с двумя доменами, а затем, вторично, один домен потеряли. Это говорит в пользу гипотезы о "слабых связях".
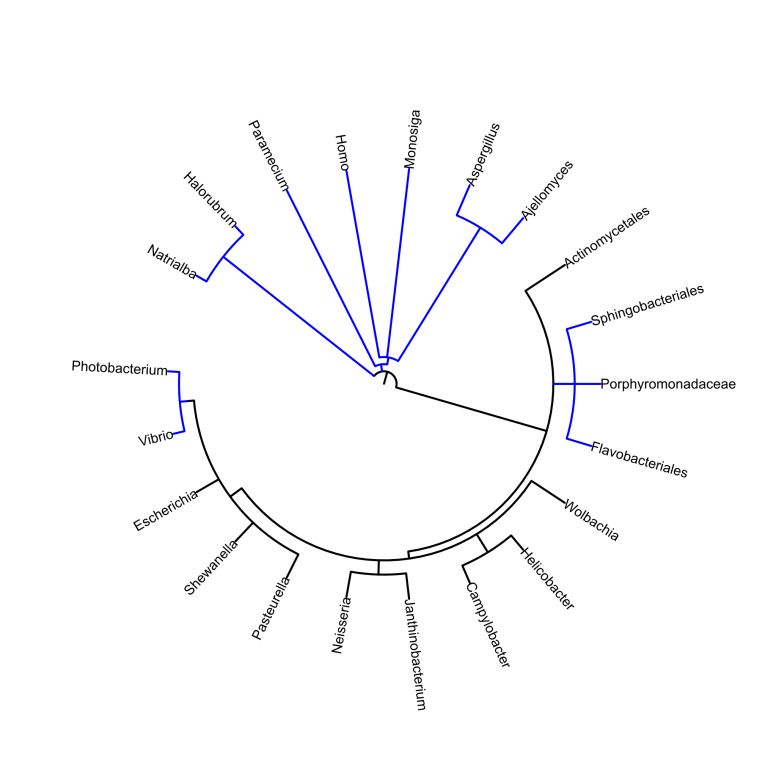
Это дерево построено с помощью iTOL. Синие - (А+В), чёрные - (А). Видно что две группы бактерий в ходе эволюции потеряли второй домен.
© Медведева