| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
Эволюционные домены. Банки Pfam и InterPro.
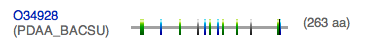
Доменная архитектура белка PDAA_BACSU в соответствии с банком Pfam
- Предсказание сколько теоретически раз слово "light" встречается в банке SwissProt: для этого перемножим вероятности нахождения букв слова и все это умножим еще на количество букв: 0.0966*0.0596*0.0707*0.0227*0.0534*191670831=94.6 => 95 раз.
- Определение сколько раз оно точно встречается в банке SwissProt: Для этого используем сервис PROSITE и получаем 94.
Результаты совпали. Как видно по найденному, слово "light" встречается в самых разных местах последовательностей => его расположение случайно.
Нахождение вероятных гомологов белка PDAA_BACSU в банке SwissProt с помощью паттернов
Создаем слабый и сильный партерны и получаем с помощью них гомологи (табл. 1).
| ||||||||||||||||
| Таблица 1. Поиск вероятных гомологов белка PDAA_BACSU по различным паттернам. |
Использование ни одного паттерна не дало мне найти последовательности гомологов из множественного выравнивания. Почему? Не потому, что у меня плохие гомологи или плохие паттерны, а потому что используемые последовательности находятся в UniProtKB/TrEMBL. Пробуем найти их там: очень долго грузит, видимо, очень много последовательностей даже на сильный паттерн.
Нахождение всех мотивов PROSITE в последовательности белка PDAA_BACSU
Чтобы найти мотивы белка заходим в ScanProsite, там поле слева вводим индефикатор PDAA_BACSU или O34928 и убираем галочку напротив поля "Exclude patterns with a high probability of occurrence" - это нужно для того, чтобы найти все мотивы, а не только те, которые специфичны для этого белка (ведь поле называется "Исключить моделей с высокой вероятностью возникновения"). Находим. Результаты по нахождению вы можете посмотреть в таблице 2, а так же на рисунках 1 и 2 видна организация белка и расположение на нем мотивов соответственно.