| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
Построение и визуализация электронной плотности (ЭП)
Выберем белок, полученный экспериментально, для изучения по публикациям в PubMed. Мне заходелось найти какой-нибудь белок человека, поэтому я остановила свой выбор на белке, который регулирует экспрессию генов, связываясь с нуклеотидными последовательностими - белок 1 гистидиновой триады (human histidine triad nucleotide-binding protein 1), его идентификатор в PDB 3TW2. Его разрешение 1.38 анстрема.
Проверим выполнение требований к белку:
- Автор модели структуры положил в PDB экспериментальные данные, а не только модель, поэтому белок есть на сервисе EDS: 3TW2. А также есть файл "структурных факторов" (Strucure factors).
- Есть подходящие структурные гомологи белка (проверка с помощью сервера PDBeFold => launch => Query - PDB код 3TW2). Сводная таблица с результами. В этой таблице видно, что есть 55 белков гомологов данного белка, у которых значение RMSD лежит в интервале [0.8,3] и значение Nalgn - [68,125] (значения означают, что есть от 50% до 90% аминокислотных остатков 3TW2). RMSD - оценка среднего расстояния между Cальфа - атомами соответствующих остатков при суперпозиции структур. А Nalgn - число сопоставленных остатков, т.е. длина выравнивания.
Построение изображение ЭП вокруг полипептидной цепи
С сайта EDS был скачан файл с картой электронной плотности. Для визуализации электронной плотности был использован PyMol. В элементарную ячейку попала одна молекула белка, что можно видеть на рисунке 1.
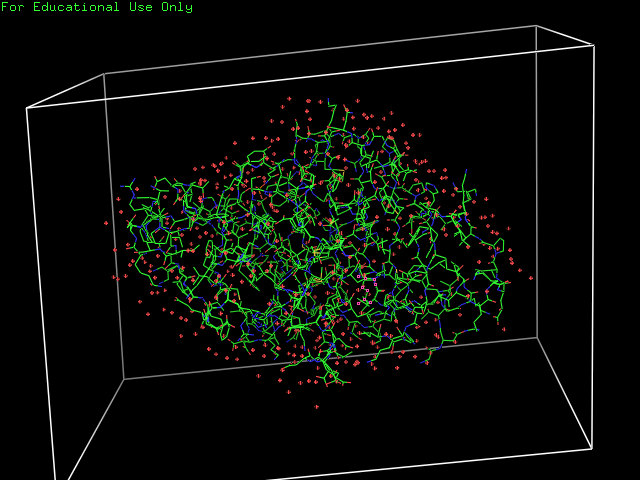 |
| Рис. 1. Молекула белка 1tw2 в элементарной ячейке |
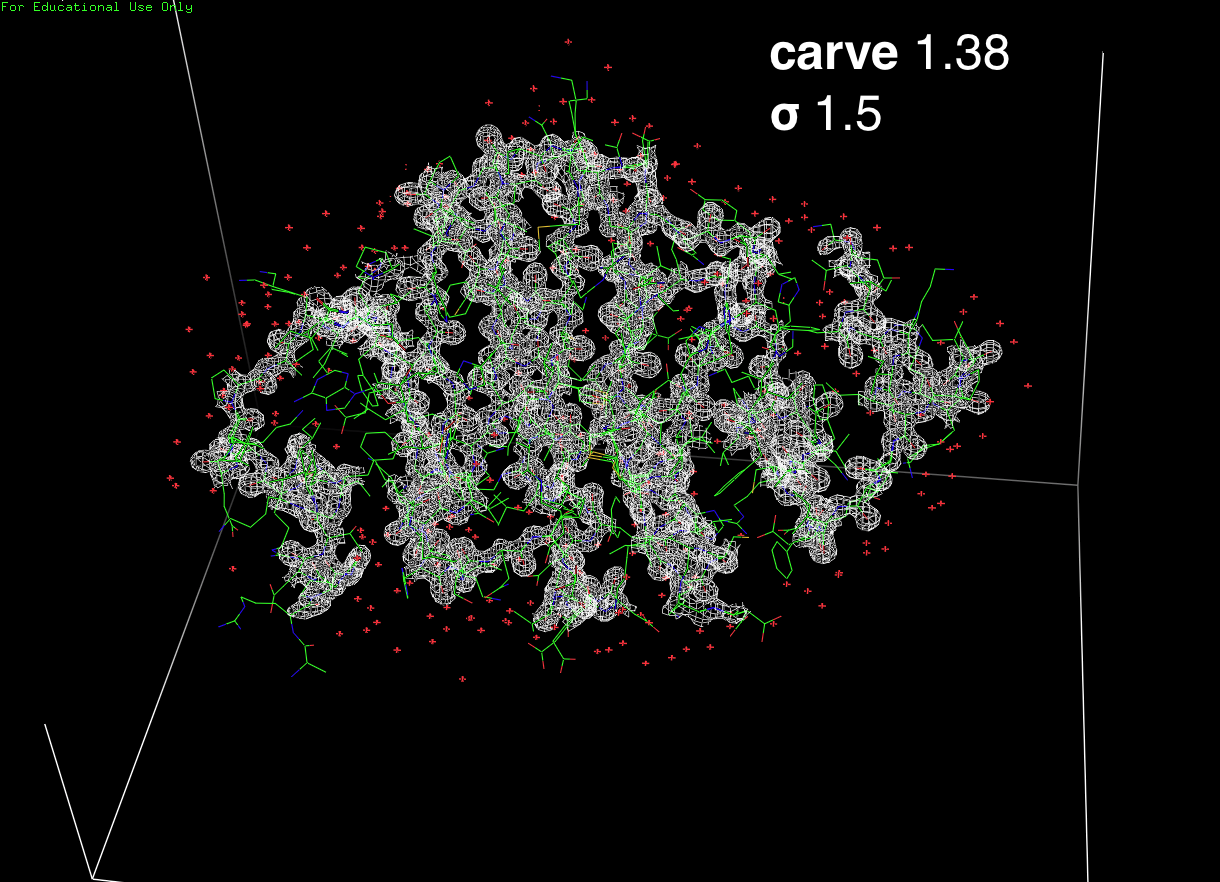
При визуализации используются не физические единицы измерения — "заряд/ангстремы в кубе", а статистические — превышение сигнала над шумом Z. Для вычисления Z рассчитывается среднее значение ЭП по ячейке кристалла M и среднее квадратичное отклонение S (сигма). Для каждой точки вычисляется Z = (R – M)/S. Здесь R — значение ЭП в данной точке пространства (x,y,z) в физических единицах, Z — значение ЭП "в сигмах" в той же точке. Далее были получены изображения ЭП вокруг полипептидной цепи с различными уровнями подрезки: расстояниями от выбранного множества (carve, ангстремы) и значениями S (рис. 2.1) с помощью комманд:
isomesh backbone_map1, 3tw2-map, 1, backbone, carve=1.38
isomesh backbone_map2, 3tw2-map, 1.5, backbone, carve=1.38
isomesh backbone_map3, 3tw2-map, 2, backbone, carve=1.38
isomesh backbone_map4, 3tw2-map, 2, backbone, carve=4
 |
 |
 |
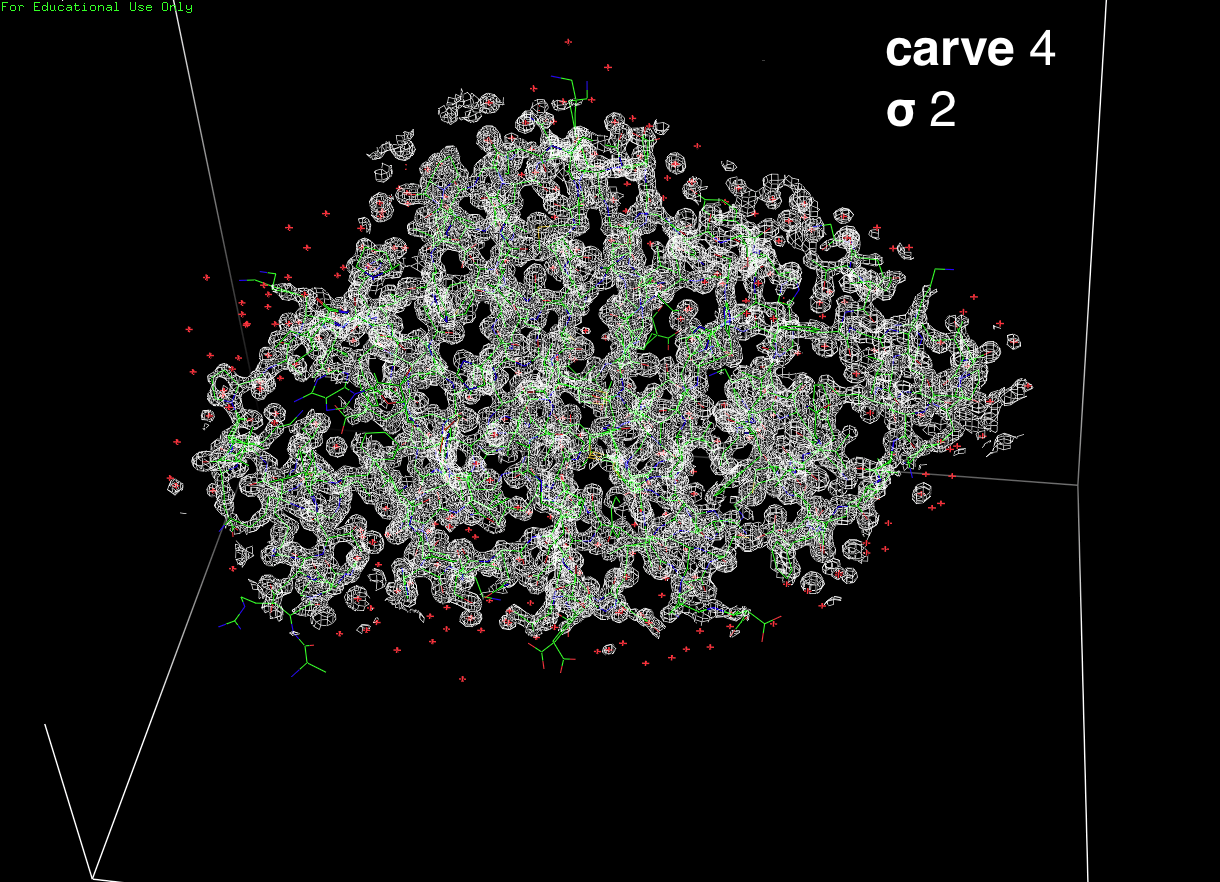 |
| Рис. 2.1 Изображения ЭП 1tw2 с различными уровнями подрезки. | |
Посмотрим повнимательнее на то, что получается при carve = 4 (рис. 2.2).
Построение изображение ЭП вокруг трех-пяти аминокислотных остатков
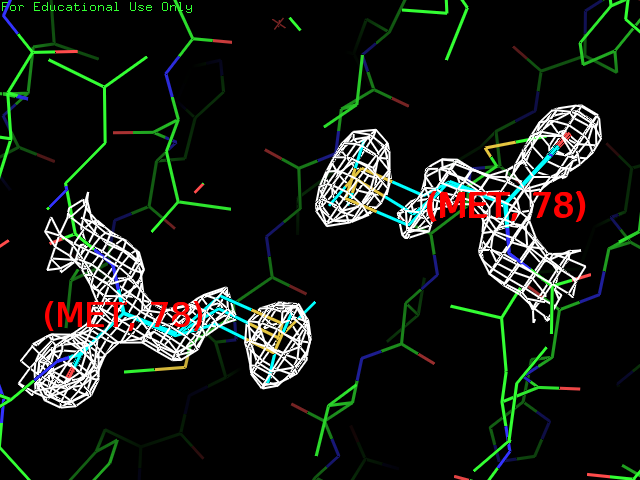
Для интереса взяли разные аминокислотные остатки.
 |
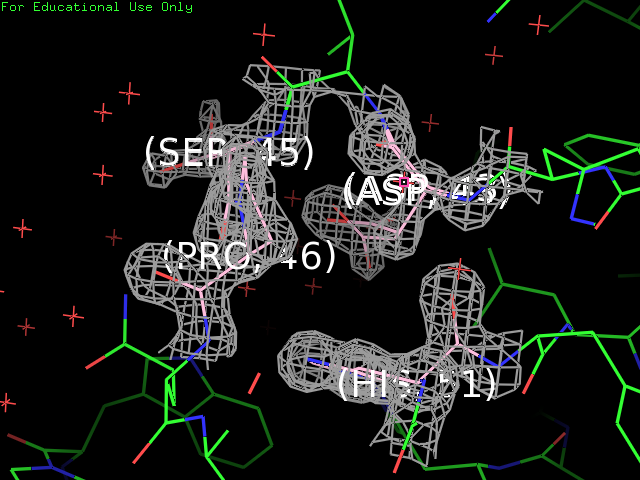 |
| Рис. 3. Изображения ЭП некоторых остатков 1tw2 с carve=2 и S=2. | |
Разрешение структуры почти атомарное и, как видно из рисунка 3, визуализация электронной плотности позволяет предположить расположение центров отдельных атомов.
Для визуализации использовались в частности эти команды:
select 2met, (chain A or chain B) and resi 78
isomesh backbone_map5, 3tw2-omap, 2, 2met, carve=2
label n. CA and i. 78, "(%s, %s)" % (resn, resi)
set label_color, red, /2met
set label_size, -1
Визуализация электронной плотности вокруг аминокислотных остатков данного белка показывает, что разрешение структуры хорошее, чтобы наблюдать возможные различные конформации отдельных аминокислотных остатков. И в целом координаты атомов соответствуют электронной плотности как видно на рисунке 1, но похоже, что структура подвижна, поэтому многие концевые остатки не вписаны в электронную плотность даже при значениях carve 4 и сигма 2.
.png)
.png)
