Навигация по сайту: |
A- и В- формы ДНК. Структура РНКЗадание 1. Программа fiber пакета 3DNAС помощью программы fiber были сгенерированы дуплексы A-DNA, B-DNA и Z-DNA длиной 20 пар нуклеотидов:
*Возможное объяснение того, что в Z-форму входят только С и G в том, что поскольку Z-форма менее устойчива чем А- и B- формы[S. Neidle, Principles of Nucleic Acid Structure, doi: 10.1016/B978-0-12-369507-9.X5001-8, p.68-69.], поэтому можно предположить, что в ее состав могут входить только нуклеотиды, которые формируют три водородные связи. Задание 2. Расположение цитозина в экспериментальной структуре. Характеристики различных форм спирали ДНКРассматривалось расположение цитозина 9 в А-цепи A-формы ДНК (PDB-ID кристаллической структуры 3v9d). На рисунке ниже атомы, обращенные в сторону большой бороздки, покрашены красным, в сторону малой — синим. В сторону большой бороздки обращены атомы С4, N4, С5, С6. В сторону малой бороздки обращены атомы N1, C2, O2, N3. Были проанализированы экспериментальные структуры А-(3v9d), B-(1bna) и Z-(1tne) форм ДНК:
**Слишком маленький фрагмент, чтобы оценить. Задание 3. Структура тРНКТорсионные углыС помощью find_pair и analyze пакета 3DNA была получена информация о структуре пролиновой(CGG) тРНК(PDB-ID: 1H4S). Данные о торсионных углах тРНК и различных форм ДНК были проанализированы с помощью MS Excel(ссылка на файл). Было выяснено, что по геометрии дуплексы тРНК больше всего похожи на А-форму ДНК, что соответсвует представлениям о предпочтительности этой формы для двухцепочечной РНК. Ту же информацию можно было найти и в файле "1H4S_old.out" выдачи analyze для нашей структуры. 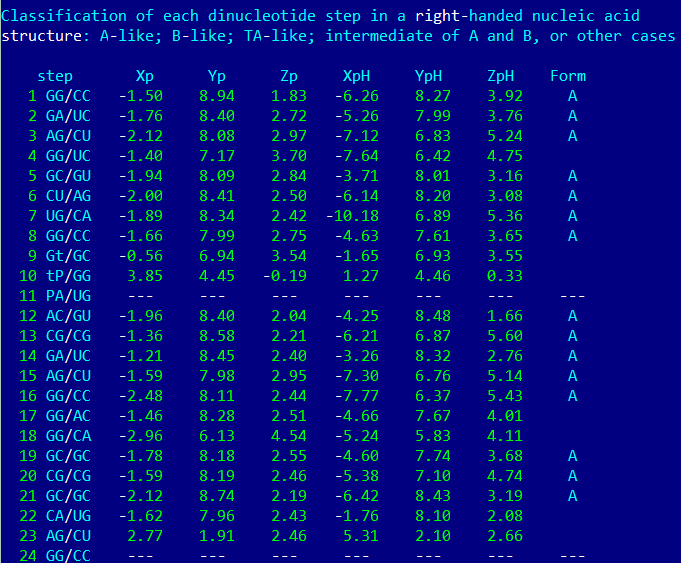
Стебли тРНКСогласно сайту 3DNA, в состав стебля могу входить только канонические или wobble пары, будем иметь это в виду. Ниже схематично представлена последовательность тРНК, стебли выделены цветом. Пара 49G-U65 является wobble-парой, поэтому она входит в стебель. 4G-----C695G-----C68 6A-----U67 7G-----C66 49G-*---U65 50C-----G64 51U-----A63 52G-----C62 53G-----C61 54[5MU]-**--G58 55[PSU]-**+-G18 38A-----U32 39C-----G31 40G-----C30 41A-----U29 42G-----C28 43G-----C27 44G-**--A26 10G-----C25 11C-----G24 12G-----C23 13C-----G22 14A-**--U8 15G-**+-C48 19G-----C56 Пары 54[5MU]-G58 и 55[PSU]-G18 образованы неканоническими основаниями: тимином и псевдоуридином. Пара 44G-A26 является парой двух пуринов, в паре 14A-U8 аденин образует водородную связь атомом N7, пара 15G-C48 является обратной Уотсон-Криковской. Таким образом, перечисленные пары являются неканоническими. Дополнительно третичную структуру стабилизирует изолированная каноническая пара 19G-C56. Стекинг-взаимодействияС помощью программы stack2img были построены изображения лучшего и худшего стекинг-взаимодействий в молекуле тРНК. |