Навигация по сайту: |
Геномное окружение. База данных GOПолучение информации о КОГе, к которому относится белокБудем анализировать белок ALV08439.1 из далекого первого семестра. С помощью CDD получили, что наш белок относится к COG4558 c e-value 3.41e-76. Данный COG обнаруживается в белке с 11 по 294 остаток (при длине самого белка в 298 остаток). COG4558 называется "ABC-type hemin transport system, periplasmic component" и имеет категорию P (Inorganic ion transport and metabolism) или, говоря по-русски, "Переплазматическая компонента транспортной системы гемина по типу ABC", категория "Транспорт и метаболизм неорганических ионов". Визуализация геномного окруженияС помощью программы COGNAT получим геномное окружение нашего COG-a в 711 прокариотических геномах. Параметры запуска: COG: COG4558, Neighborhood Size: 11, Occurrence Threshold (%): 20, Taxonomy: Да. Использующаяся легенда: 
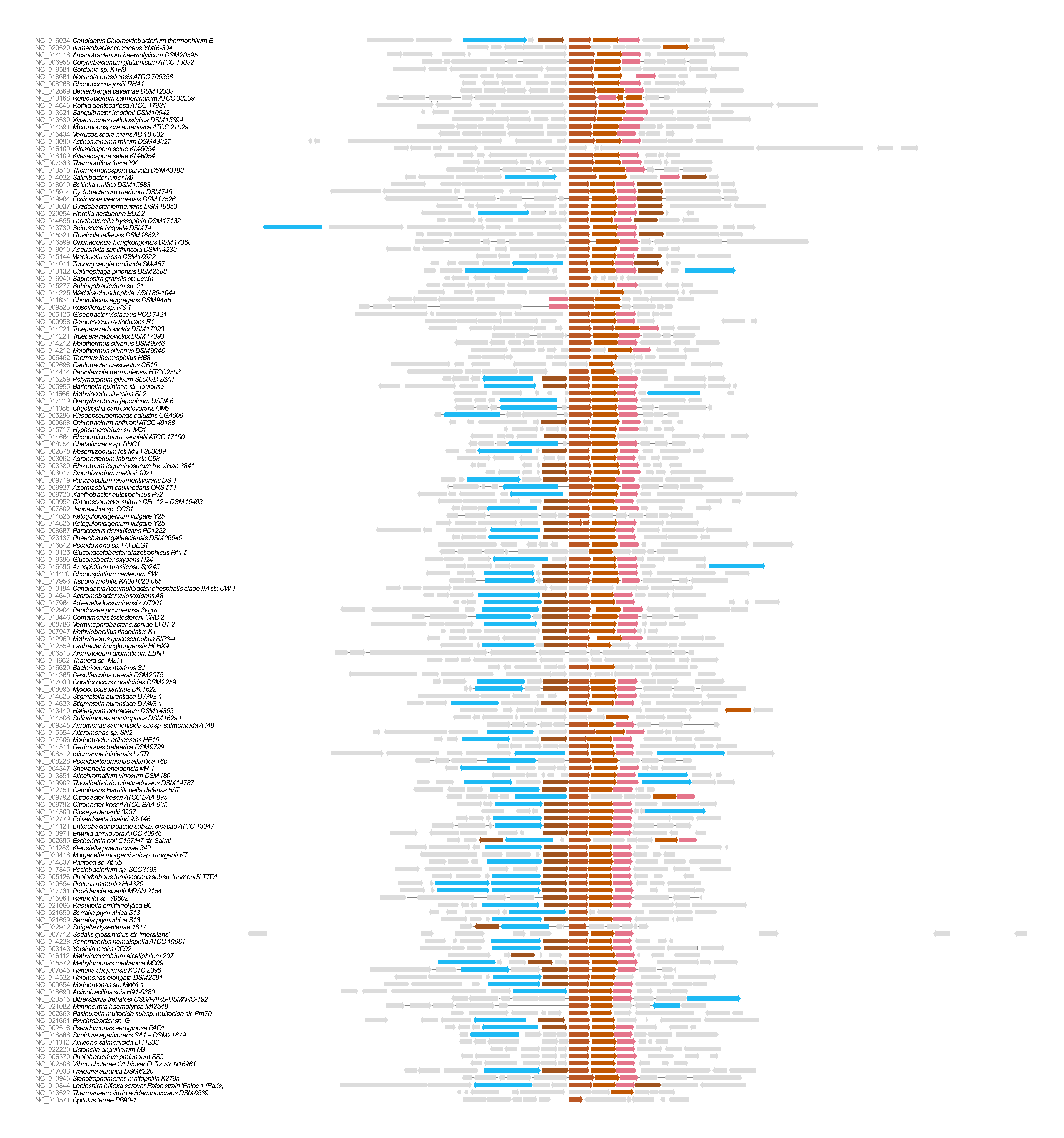
Ссылка на файл выдачи COGNAT. Отнесение белка ALV08439.1 из Roseateles depolymerans KCTC 42856 к терминам GOНаиболее похожим на наш оказался белок VC_A0913 из организма Vibrio cholerae O1 biovar El Tor str. N16961. Полное название — Hemin ABC transporter, periplasmic hemin-binding protein HutB. Похоже на ALV08439.1. Что касается цифр, то P-value находки составило P = 1.4e-32, что вполне достаточно, чтобы считать его гомологом. Интересно, что при включенном "Blast filter" часть последовательности запроса замаскировалась буквой Х. При отключении этого фильтра получаем для этого же VC_A0913 P-value = 8.9e-38 за счет дополнительных match-ей в этом регионе. Наверное замаскированный участок был участком малой сложности. Таблица 1. Термины GO, отнесенные к белку с идентификатором Uniprot Q9KL36 (Q9KL36_VIBCH).
Таблица 2. Описание кодов достоверности, использованных в Таблице 1.
Таким образом, в этом практикуме мы идентифицировали кластер ортологичных генов, куда входит наш белок, посмотрели на геномное окружение ортологов этого гена в разных организмах и попытались найти термины GO для нашего белка. Найдя в базе данных GO белок наиболее похожий на рассматриваемый, получили термины GO для рассматриваемого белка. Термины перечислены в таблице 1, расшифровка типов достоверности в таблице 1 представлена в таблице 2. |