Домены и профили
Что было сделано:
- был составлен список белков Uniprot с заданным составом доменов Pfam
- был построен hmm профиль семейства белков с выбранной архитектурой
- была проверена работа hmm профиля семейства белков
- было проведено сравнение филогенетических деревьев двух доменов из выбранной доменной архитектуры
Список белков Uniprot с заданным составом доменов Pfam
Сначала выберем домен. Пусть это будет PF01740 (family: STAS). Вот немного информации о домене:
- AC: PF01740
- ID: STAS
- Название: Sulphate Transporter and AntiSigma factor antagonist
- Число последовательностей среди бактерий: 19098
- Функция белков домена: Sulphate Tansporter и bacterial AntiSigma factor antagonist
- Ссылка на страницу домена в Pfam
Выберем какую-нибудь двухдоменную архитектуру, содержащую выбранный нами домен. Пусть это будет двухдоменная архитектура, содержащая помимо выбранного уже домена домен PF04960 (family: Glutaminase). :
- AC: PF08448
- ID: PAS_4
- Название: PAS fold
- Число последовательностей среди бактерий: 4823
- Функция белков домена: структурный белок
- Ссылка на страницу домена в Pfam
Архитектура PAS_4, STAS встречается у 111 белков. Составим таблицу с информацией о всех бактериальных белках с выбранной архитектурой. Для этого зайдем в Uniprot и вобьём такой запрос: database:(type:pfam pf08448) database:(type:pfam pf01740) taxonomy:"Bacteria [2]". В настройках Columns выбираем те колонки, которые нас интересуют:
- Organism (Names & Taxonomy)
- Protein names (Names & Taxonomy)
- Length (Sequences)
- Taxonomic lineage (FAMILY) (Taxonomic lineage)
- Taxonomic lineage (GENUS) (Taxonomic lineage)
- Taxonomic lineage (PHYLUM) (Taxonomic lineage)
- Pfam (Family and domain)
По запросу вышло 625 результатов. Далее скачиваем таблицу в формате Excel. Вот ссылка на полученную таблицу.
hmm профиль семейства белков
Для того, чтобы составить hmm профиль, набрали 51 последовательность. Это делалось так: сначала по длине последовательности построили гистограмму на Python, наиболее характерной длиной данных последовательностей оказалась 390-440 п.о. На полученную диаграмму можно посмотреть на картинке ниже. Далее последовательности фильтровались по доменам, отбирались только те последовательности, которые не содержали дополнительных доменов. Из оставшихся 66 последовательностей случайным образом отобрали 51 последовательность. Она представлена на листе "selected" таблицы Excel
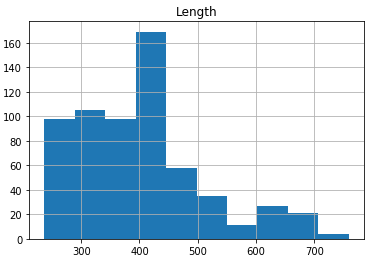
Далее выбранные в
Uniprot последовательности, содержащие один из доменов, понадобилось скачать и выровнять. Выравнивание было произведено
с помощью muscle в программе MEGA.
После этого провели ревизию последовательностей: был найден самый N-концевой консервативный блок, и вырезаны все позиции до него;
удалили последовательности, не содержащие этого N-концевого блока. На картинке ниже представлен N-конец отредактированного выравнивания.

С-концы после последнего С-концевого консервативного блока тоже были вырезаны. На картинке ниже представлен С-конец отредактированного выравнивания.

Сохранили файл с полученным выравниванием.
Далее для построения профиля была использована команда hmm2build пакета HMMER.
hmm2build -g profile.hmm alignment.fasta
Для калибровки профиля использована команда hmm2calibrate.
hmm2calibrate profile.hmm
Программой hmm2seqrch был проведен поиск по всем белкам Uniprot, включающим домен, с пороговым E-value 0,001. Для проведения поиска по всем белкам Uniprot, включающими первый домен из моей архитектуры, нужно было получить fasta-файл с последовательностями всех белков с доменом STAS, найденных выражением database:(type:pfam pf01740) taxonomy:"Bacteria [2]" (Uniprot выдал 79500 результатов - в два раза меньше по сравнению с запросом о втором домене).
hmm2search profile.hmm --domE 0.001 pf01740.fasta > info.txt
Файл с откалиброванным профилем.
Файл с информацией о выравниваниях.
Была выдана информация о 6957 последовательностях из 79500.
Далее проводилось сравнение списков с помощью Excel. C помощью таблицы Excel была построена ROC-кривая и кривая весов. Ссылка на таблицу с графиками.
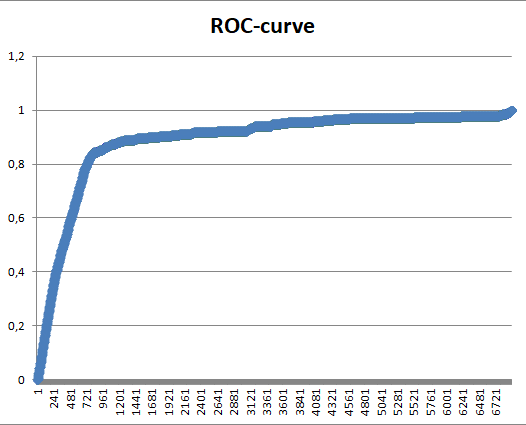 ROC-кривая
ROC-кривая
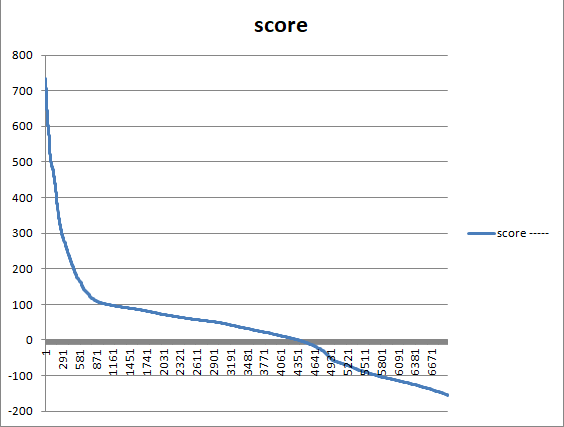 Распределение весов находок.
Распределение весов находок.
Площадь под графиком ROC-кривой составила 0,88. Это значит, что метод поиска двухдоменных архитектур довольно хорош.
В качестве порога выбрали вес, равный 113,1, evalue, равный 6,30E-30.
Таблица "истинности"
| # | two-domed | not two-domed |
|---|---|---|
| true | 592 | 33 |
| false | 6365 | 72543 |

