Мотивы и профили II
- Что было сделано:
- было вычислено информационное содержание (IC) последовательности Козак в геноме Danio rerio и построено LOGO этого сигнала
- была проведена проверка PWM для сайтов регуляции разрывной транскрипции sgmRNA
Вычисление информационного содержания (IC) последовательности Козак в геноме Danio rerio
Дано было выравнивание десятка контекстов ATG из статьи Grzegorski et al., PLoS ONE 9(9): e108475, 2014. Я выполнила задание из варианта 5 вот с таким выравниванием:
aatcatgac taacatggc aaccatggt aagaatggc caccatggt cagcatgga aaacatgga caacatgat cacaatggt cagcatggc cacaatggc
Для подсчета IC необходимо было сначала посчитать количество нуклеотидов в позициях выравнивания.
Затем посчитать частоты нуклеотидов в позициях выравнивания. Далее считалось информационное содержание
каждого нуклеотида в колонке, а затем суммарное информационное содержание. Подробнее
ознакомиться с процессом поиска информационного содержания выравнивания можно здесь.
После этого, воспользовавшись сервисом webLOGO,
я построила LOGO выравнивания. Результат можно найти на картинке ниже.

Проверка PWM для сайтов регуляции разрывной транскрипции sgmRNA
Проверим мотив, полученный в предыдущем практикуме с помощью сервиса MEME. Как ни печально, мои мотивы были во многом далеки от идеала. Поэтому пришлось делать их поиск сначала. Запустив MEME, я ввела fasta-файл с upstream областями, а также ввела следующие параметры:
- Select the site distribution: Zero or One Occurence Per Sequence (zoops)
- Select the number of motifs: 3
- How wide can motifs be? 6 - 10
- Can motif sites be on both strands? search given strand only
- What should be used as the background model? 0-order model of sequences
MEME выдал мне вот эту html-страничку и больше всего мне понравился вот этот мотив - CTCAACTAAA - из 10 нуклеотидов, присутствующий во всех семи upstream областях (во всех последовательностях по одному сигналу!), на картинке ниже. Он более всех соответствует тому самому мотиву из статьи - CTAAAC Вы поймете мой восторг, когда посмотрите на позиции 1-6 мотива ниже. А еще радует глаз положение данных мотивов (обозначено красным) перед старт-кодоном. У этого мотива достаточно мальенькое E-Value (6.3e-002), чтобы утверждать, что находка не случайна.
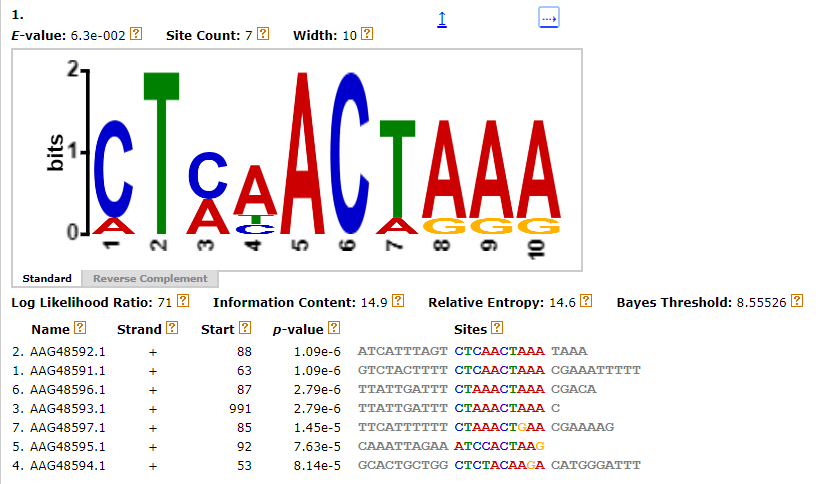
Все, что мне потребовалось сделать - это сабмитнуть данный мотив в FIMO. Это очень удобно делать прямо с html-странички, выданной в МЕМЕ. Пользовалась следующими параметрами:
- scan given strand only
- E-Value: 0.0001
Вот, что мне выдал FIMO:
| Motif ID | Ближайший ген | Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|---|---|
| 1 | внутри ORF1ab | NC_002645.1 | + | 63 | 72 | 1.39e-06 | 0.019 | CTCAACTAAA |
| 1 | S | NC_002645.1 | + | 20556 | 20565 | 1.39e-06 | 0.019 | CTCAACTAAA |
| 1 | M | NC_002645.1 | + | 24980 | 24989 | 3.15e-06 | 0.0287 | CTAAACTAAA |
| 1 | S (в конце, перед 4а) | NC_002645.1 | + | 24043 | 24052 | 1.36e-05 | 0.0926 | ATCAACTAAA |
| 1 | N | NC_002645.1 | + | 25669 | 25678 | 1.95e-05 | 0.107 | CTAAACTGAA |
| 1 | внутри replicase polyprotein 1ab | NC_002645.1 | + | 3279 | 3288 | 3.98e-05 | 0.155 | CTCAACAAGA |
| 1 | replicase polyprotein 1ab | NC_002645.1 | + | 15151 | 15160 | 3.98e-05 | 0.155 | CTCAACAAGA |
| 1 | в конце S, перед 4а | NC_002645.1 | + | 24433 | 24442 | 9.31e-05 | 0.282 | CTCTACAAGA |
| 1 | Е | NC_002645.1 | + | 24740 | 24749 | 9.31e-05 | 0.282 | ATCCACTAAG |
В 2 сигналах из 9 содержатся все 6 нуклеотидов CS, в остальных 7 сигналах содержатся 5 из 6 нуклеотидов CS.
Все сигналы, за исключением одного ([15151..15160]) находятся почти вплотную к своим генам. Среди всех сигналов
нашлись даже сигналы, находящиеся внутри или поблизости полипротеина. Не во всех генах присутствовал единственный сигнал.
В итоге, подтвердились сигналы для 5 из 6 поздних генов и 1 для лидирующего гена.
Было построено LOGO для последовательностей сигнальных мотивов поздних генов коронавируса. Можно увидеть его на картинке ниже.

Далее был выполнен поиск найденного в МЕМЕ мотива для двух геномов коронавирусов. Один геном я взяла от другого штамма 229E-related bat coronavirus.
Вот, что мне выдал FIMO:
| Motif ID | Alt ID | Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|---|---|
| 1 | KT253271.1 | + | 63 | 72 | 1.39e-06 | 0.0399 | CTCAACTAAA | |
| 1 | KT253271.1 | + | 26183 | 26192 | 3.15e-06 | 0.0453 | CTAAACTAAA | |
| 1 | KT253271.1 | + | 20571 | 20580 | 8.95e-06 | 0.0858 | CTCAACTAAG | |
| 1 | KT253271.1 | + | 28120 | 28129 | 1.36e-05 | 0.0975 | ATCAACTAAA | |
| 1 | KT253271.1 | + | 26872 | 26881 | 1.95e-05 | 0.112 | CTAAACTGAA | |
| 1 | KT253271.1 | + | 22937 | 22946 | 3.34e-05 | 0.16 | CTCCACAAAA |
Сигналы этого коронавируса похожи на сигналы моего. Для 2 из 6 последовательностей имеется мотив, представленный в ссылке - СТАААС.
Даже коордиинаты мотивов примерно похожи.
Второй геном я взяла от близкородственного вида - Rousettus aegyptiacus bat coronavirus 229E-related.
Вот что для него выдал FIMO:
| Motif ID | Alt ID | Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|---|---|
| 1 | MN611517.1 | + | 53 | 62 | 1.39e-06 | 0.0192 | CTCAACTAAA | |
| 1 | MN611517.1 | + | 20576 | 20585 | 1.39e-06 | 0.0192 | CTCAACTAAA | |
| 1 | MN611517.1 | + | 25572 | 25581 | 3.15e-06 | 0.029 | CTAAACTAAA | |
| 1 | MN611517.1 | + | 23705 | 23714 | 8.95e-06 | 0.0618 | CTCAACTAAG | |
| 1 | MN611517.1 | + | 24633 | 24642 | 1.36e-05 | 0.0749 | ATCAACTAAA | |
| 1 | MN611517.1 | + | 26264 | 26273 | 1.95e-05 | 0.0899 | CTAAACTGAA | |
| 1 | MN611517.1 | + | 25023 | 25032 | 3.62e-05 | 0.137 | CTCTACAAAA | |
| 1 | MN611517.1 | + | 3290 | 3299 | 3.98e-05 | 0.137 | CTCAACAAGA |
Результат так же схож с выдачей FIMO моего коронавируса.
Cкорее всего, сигнальные мотивы моего коронавируса специфичны для вида.
