
 |
Учебный сайт Юдиной А.С. |
Главная |
Обо мне |
Семестры |
HMM профиль. Выбор целевого семейства.Для построения профиля была определена выборка из домена PF00932 у представителей Chordata (ранг: тип).
Построение профиля.Полученные последовательности были выравнены в программе Jalview (Рис.1). Выравнивание было поправлено вручную: убраны плохо выровнявшиеся концы последовательностей, в начале добавлены и удалены гэпы для достижения большего числа консервативных позиций. Две последовательности были удалены из выравнивания. Ссылка на проект выравнивания: LTD_align. 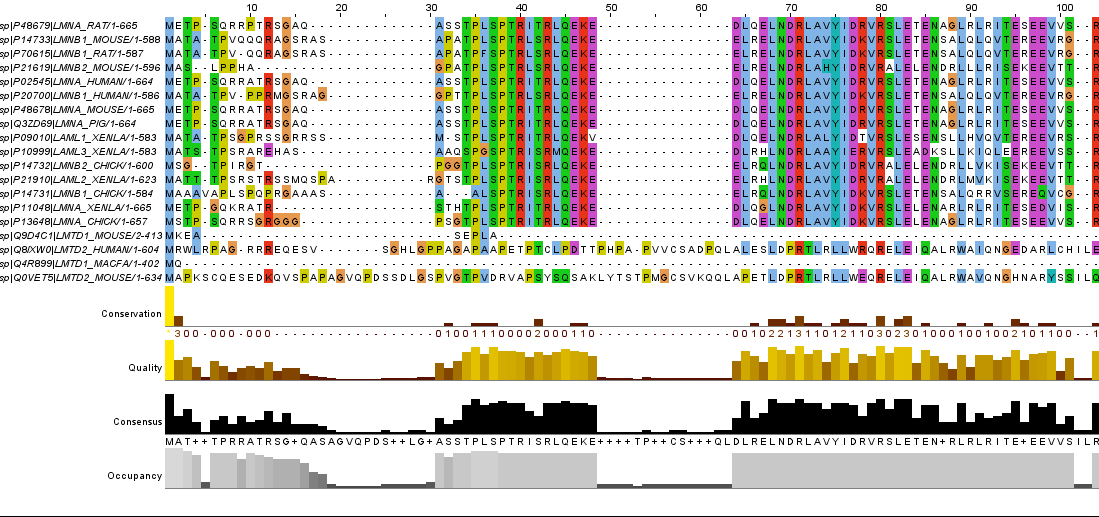
Рис.1 Этапы построения профиля:
Поиск по профилю в Swissprot.С помощью команды hmm2search --domE 1000 --domT -50 profile_new.out /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > find_new.out
среди записей банка данных Swissprot были найдены последовательности, соответствующие построенному профилю.
Найденная данные по доменам были добавлены в исходную таблицу Ecxel. С помощью функции ВПР были отмечены
последовательности, изначально найденные в Uniprot.
Построение ROC кривой.Для разных выбранных порогов были вычислены коэффициенты Sensitivity (True Positive/ (True Positive + False negative)) и Specificity (True Negative/(False Positive + True Negative)). По величинам Sensitivity и 1-Specificity построена ROC кривая (Рис.2). 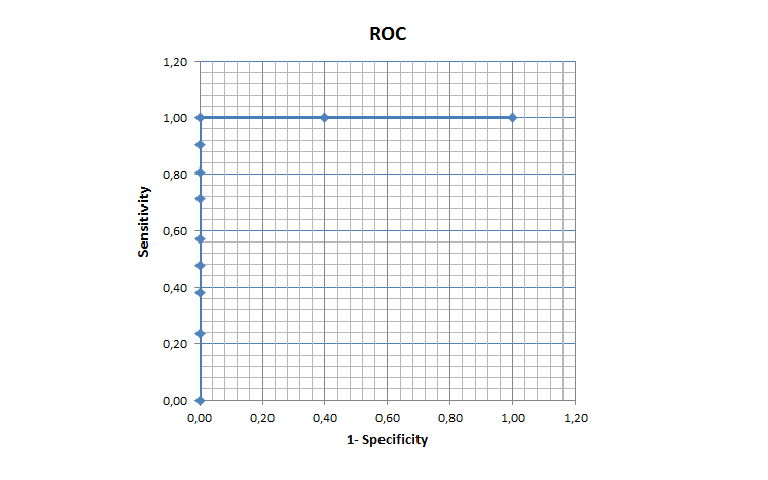
Рис.2 По гистограмме (Рис.3) весов найденных в Swissprot последовательностей определен порог. 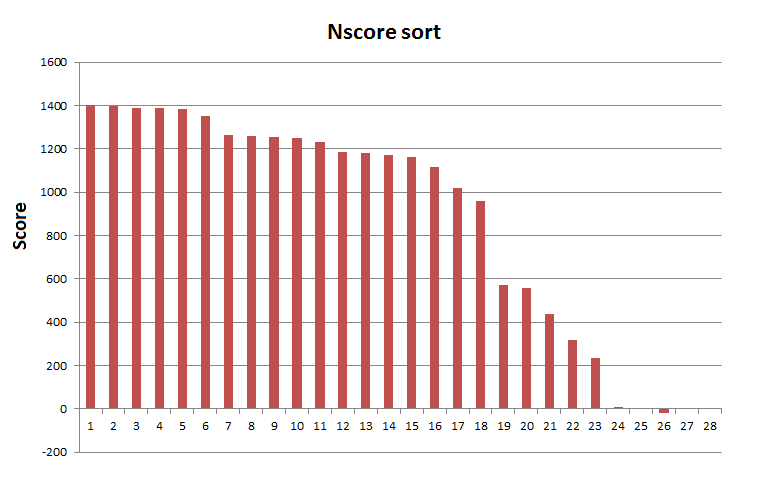
Рис.3 Порог: 436.
Вывод: Профиль достаточно точен для определения принадлежности белка к семейству, так как
из найденных в Swissprot 26 белков с помощью профиля был найден 21 белок (они же и были ранее найдены в Uniprot) с точностью 1.
Вид ROC-кривой подтверждает такой вывод.
|
||
© Юдина Анастасия, 2016