Формы ДНК. Структура РНК
Задание 1
С помощью инструментов пакета 3DNA были построены модели структур A-, B- и Z-формы дуплекса ДНК, последовательность одного из цепей которого — пять повторов GATC.
Файлы с полученными моделями:
Для построения была использована команда ‹fiber -a-seq=GATCGATCGATCGATCGATC gatc-a.pdb› в случае с А-формой, опция -а заменялась на -b и -z для B- и Z-формы соответственно.
Задание 2
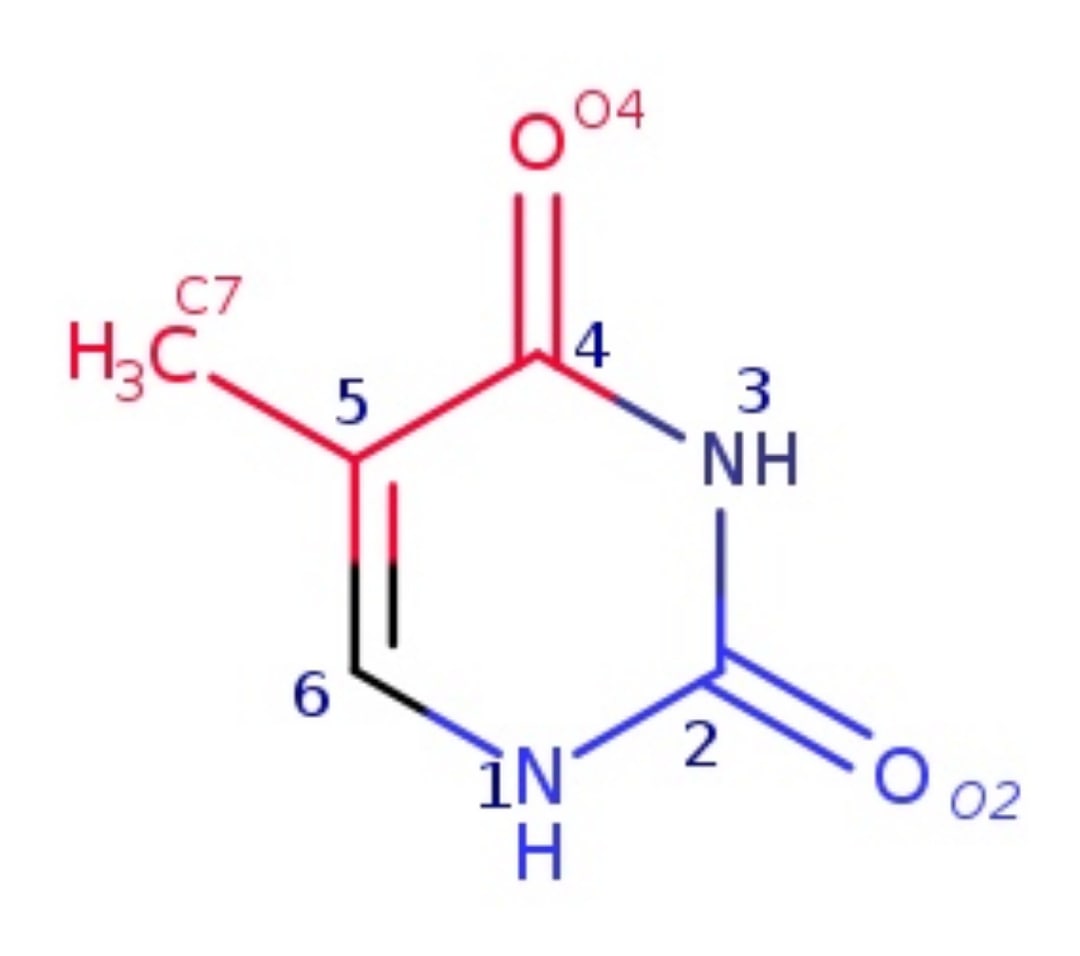
В цепи B экспериментально полученной структуры А-формы ДНК (3V9D) было выбрано 7 основание — тимин, который выделяется скриптом с участком сахаро-фосфатного остова в апплете ниже.
Далее были определены атомы, явно обращённые в сторону большой бороздки (C4, C5, C6, O4), и атомы, явно обращённые в малую (N1, C2, O2). На Изображении 1 представленная нарисованная в MarvinSketch молекула тимина, атомы которой окрашен в зависимости от обращённости к определённой бороздке. Синим покрашены направленные в сторону малой, а красной в сторону большой.
Также были измерены параметры различных форм ДНК из экспериментально полученных структур (1tne для Z-формы, 1bna для B-формы, 3v9d для A-формы). Результаты измерений приведены в таблице ниже.
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28.09 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7.98(3:A.P 32:B.P) | 17.21 (29:B:P-9:A:P) | 7.2 (33:B.P-11:A.P) |
| Ширина малой бороздки | 16.81 (10:A:P-33:B:P) | 13.1 (30:B:P-14:A:P) | 16.08 (28:B.P-12:A.P) |
Задание 3. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
В этой части практикума работа ведётся с т-РНК 1G59. К сожалению, пакет 3DNA позволяет работать только со старым форматом pdb, поэтому сначала необходимо для конвертации воспользоваться командой remediator ‹--old 1g59.pdb > 1g59_old.pdb›.
Далее с помощью команды ‹find_pair -t 1g59_old.pdb stdout | analyze› были получены файлы, в одном из которых (1g59_old.out) можно найти информацию о торсионных углах и водородных связях.
Определение торсионных углов
Ниже представлена таблица созначениями торсионных углов для первой цепи.
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- -157.0 59.6 98.9 -153.8 -79.8 -171.3
2 G -84.9 -162.0 51.6 96.1 -155.3 -68.9 -154.4
3 C -69.6 -179.4 51.3 92.5 -152.5 -74.0 -148.7
4 C -60.1 176.5 47.0 90.4 -161.9 -83.7 -149.4
5 C -60.4 168.9 51.9 89.1 -157.7 -69.5 -161.6
6 C -66.0 -176.8 55.6 96.2 -161.6 -76.4 -165.7
7 A -73.5 174.6 73.2 128.9 -65.1 -113.2 -144.7
8 G 59.7 151.9 31.3 97.1 -148.8 -79.0 -170.4
9 G -75.8 176.5 63.6 91.3 -160.6 -64.4 -165.4
10 G -63.8 -170.7 41.7 90.7 -162.9 -97.4 -156.2
11 G 130.2 -154.5 -168.9 104.6 -118.0 -26.4 -178.1
12 G -109.6 56.1 178.5 110.9 -115.0 -87.5 167.2
13 U 0.9 155.6 -12.8 97.3 -136.3 -77.9 -152.5
14 U -69.9 156.0 60.4 88.9 -110.2 -75.4 -153.1
15 A -69.8 173.1 54.9 94.0 -146.6 -61.6 -167.3
16 G -69.5 177.9 49.6 93.0 -157.1 -77.9 -156.7
17 G -61.8 178.8 44.4 93.8 -157.3 -74.7 -154.8
18 C -81.4 175.2 64.5 87.3 -159.0 -69.1 -157.2
19 C -70.5 -179.2 55.4 87.5 -157.7 -73.2 -154.6
20 G -66.2 172.9 62.0 87.9 -158.6 -79.5 -164.1
21 A -60.2 -176.6 42.0 88.8 -141.2 -66.9 -145.2
22 G -154.9 159.7 45.6 93.8 -144.5 -73.1 -175.6
23 U -68.1 -171.7 46.2 93.1 -153.8 -79.1 -166.8
24 C -59.7 -176.0 41.7 91.1 -151.4 -71.1 -147.9
25 U 120.0 -154.7 -146.4 92.3 -161.4 -79.7 -170.3
26 U -37.9 -148.7 56.4 96.3 -148.2 -73.8 -140.9
27 A -68.2 -169.7 52.4 91.3 -134.5 -68.0 -162.7
28 G -78.8 162.3 72.4 99.5 24.2 165.6 -147.4
29 G -147.7 -111.4 52.7 121.2 58.6 139.0 -62.9
30 G --- -161.2 55.0 98.3 -155.1 -75.7 -163.9
31 G -78.8 -165.2 44.9 96.0 -151.7 -71.0 -155.8
32 C -65.5 174.8 48.7 92.0 -152.0 -75.1 -146.4
33 C -57.5 175.0 44.6 91.2 -159.7 -84.2 -151.5
34 C -64.4 170.1 55.5 88.1 -158.5 -69.8 -162.5
35 C -64.6 -177.7 55.1 96.5 -162.4 -76.1 -163.7
36 A -75.6 175.3 74.8 130.5 -66.4 -112.1 -145.1
37 G 60.1 151.0 30.0 93.2 -147.9 -78.5 -173.7
38 G -69.3 177.2 61.9 92.2 -158.5 -66.6 -166.9
39 G -61.2 -173.6 40.7 90.2 -159.8 -99.2 -157.4
40 G 126.3 -151.0 -166.9 104.6 -119.8 -27.5 179.1
41 G -110.5 56.4 178.7 112.4 -118.9 -89.0 166.5
42 U 9.3 148.6 -19.7 92.5 -131.1 -75.9 -152.2
43 U -71.9 158.4 68.3 89.9 -114.2 -74.7 -146.5
44 A -67.6 172.6 51.8 92.5 -147.5 -58.4 -167.8
45 G -79.3 -179.5 57.9 90.6 -156.5 -77.8 -157.2
46 G -60.6 177.9 43.0 92.2 -156.5 -75.3 -157.2
47 C -76.6 174.7 60.8 88.5 -159.3 -69.1 -153.4
48 C -68.9 -178.7 54.0 87.1 -156.5 -73.1 -153.3
49 G -64.2 171.0 62.2 89.1 -155.9 -81.0 -163.1
50 A -59.9 -178.7 41.1 87.0 -141.1 -67.4 -146.0
51 G -163.4 166.4 48.1 97.2 -147.7 -72.6 -174.0
52 U -66.5 -172.1 46.0 93.2 -153.5 -78.6 -159.6
| α | β | γ | δ | ε | ζ | χ | |
|---|---|---|---|---|---|---|---|
| A-форма (теор.) | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B-форма (теор.) | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
Из отрицательных значений углов α и β цепей РНК очевидно, что сравнение по средним значениям этих углов с формами ДНК не представляется возможным. Однако можно заметить, что модули этих углов зачастую достаточно близки к таковым у A- и B-форм ДНК.
Углы дельта и хи ближе к A-форме, а эпсилон по модулю к B-форме.
Водородные связи
Ниже представлена таблица с информацией о водородных связях.
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.011) ....>B:.501_:[..G]G-----C[..C]:.572_:B<.... (0.010) |
2 (0.008) ....>B:.502_:[..G]G-*---U[..U]:.571_:B<.... (0.015) |
3 (0.011) ....>B:.503_:[..C]C-----G[..G]:.570_:B<.... (0.007) |
4 (0.010) ....>B:.504_:[..C]C-----G[..G]:.569_:B<.... (0.008) |
5 (0.010) ....>B:.505_:[..C]C-----G[..G]:.568_:B<.... (0.007) |
6 (0.011) ....>B:.506_:[..C]C-----G[..G]:.567_:B<.... (0.007) |
7 (0.012) ....>B:.507_:[..A]A-----U[..U]:.566_:B<.... (0.014) |
8 (0.007) ....>B:.549_:[..G]G-----C[..C]:.565_:B<.... (0.010) |
9 (0.006) ....>B:.550_:[..G]G-----C[..C]:.564_:B<.... (0.011) |
10 (0.007) ....>B:.551_:[..G]G-----C[..C]:.563_:B<.... (0.010) |
11 (0.007) ....>B:.552_:[..G]G-----C[..C]:.562_:B<.... (0.010) |
12 (0.009) ....>B:.553_:[..G]G-----C[..C]:.561_:B<.... (0.010) |
13 (0.016) ....>B:.554_:[..U]U-**--A[..A]:.558_:B<.... (0.008) |
14 (0.017) ....>B:.555_:[..U]U-**+-G[..G]:.517_:B<.... (0.015) x
15 (0.008) ....>B:.538_:[..A]A-**--C[..C]:.532_:B<.... (0.012) |
16 (0.009) ....>B:.539_:[..G]G-----C[..C]:.531_:B<.... (0.010) |
17 (0.012) ....>B:.540_:[..G]G-----C[..C]:.530_:B<.... (0.010) |
18 (0.012) ....>B:.541_:[..C]C-----G[..G]:.529_:B<.... (0.011) |
19 (0.011) ....>B:.542_:[..C]C-----G[..G]:.528_:B<.... (0.006) |
20 (0.008) ....>B:.543_:[..G]G-----C[..C]:.527_:B<.... (0.010) |
21 (0.007) ....>B:.544_:[..A]A-**--G[..G]:.526_:B<.... (0.006) |
22 (0.010) ....>B:.510_:[..G]G-----C[..C]:.525_:B<.... (0.010) |
23 (0.015) ....>B:.511_:[..U]U-----A[..A]:.524_:B<.... (0.013) |
24 (0.012) ....>B:.512_:[..C]C-----G[..G]:.523_:B<.... (0.007) |
25 (0.017) ....>B:.513_:[..U]U-*---G[..G]:.522_:B<.... (0.010) |
26 (0.014) ....>B:.508_:[..U]U-**--A[..A]:.546_:B<.... (0.008) |
27 (0.009) ....>B:.514_:[..A]A-**--A[..A]:.521_:B<.... (0.009) |
28 (0.008) ....>B:.515_:[..G]G-**+-C[..C]:.548_:B<.... (0.009) x
29 (0.008) ....>B:.518_:[..G]G-----C[..C]:.556_:B<.... (0.015) +
30 (0.009) ....>D:.501_:[..G]G-----C[..C]:.572_:D<.... (0.010) |
31 (0.010) ....>D:.502_:[..G]G-**--U[..U]:.571_:D<.... (0.016) |
32 (0.010) ....>D:.503_:[..C]C-----G[..G]:.570_:D<.... (0.008) |
33 (0.009) ....>D:.504_:[..C]C-----G[..G]:.569_:D<.... (0.006) |
34 (0.009) ....>D:.505_:[..C]C-----G[..G]:.568_:D<.... (0.009) |
35 (0.008) ....>D:.506_:[..C]C-----G[..G]:.567_:D<.... (0.008) |
36 (0.009) ....>D:.507_:[..A]A-----U[..U]:.566_:D<.... (0.015) |
37 (0.011) ....>D:.549_:[..G]G-----C[..C]:.565_:D<.... (0.012) |
38 (0.008) ....>D:.550_:[..G]G-----C[..C]:.564_:D<.... (0.010) |
39 (0.009) ....>D:.551_:[..G]G-----C[..C]:.563_:D<.... (0.014) |
40 (0.007) ....>D:.552_:[..G]G-----C[..C]:.562_:D<.... (0.011) |
41 (0.011) ....>D:.553_:[..G]G-----C[..C]:.561_:D<.... (0.009) |
42 (0.017) ....>D:.554_:[..U]U-**--A[..A]:.558_:D<.... (0.008) |
43 (0.019) ....>D:.555_:[..U]U-**+-G[..G]:.517_:D<.... (0.022) x
44 (0.011) ....>D:.538_:[..A]A-**--C[..C]:.532_:D<.... (0.013) |
45 (0.013) ....>D:.539_:[..G]G-----C[..C]:.531_:D<.... (0.008) |
46 (0.008) ....>D:.540_:[..G]G-----C[..C]:.530_:D<.... (0.009) |
47 (0.010) ....>D:.541_:[..C]C-----G[..G]:.529_:D<.... (0.008) |
48 (0.010) ....>D:.542_:[..C]C-----G[..G]:.528_:D<.... (0.006) |
49 (0.009) ....>D:.543_:[..G]G-----C[..C]:.527_:D<.... (0.012) |
50 (0.009) ....>D:.544_:[..A]A-**--G[..G]:.526_:D<.... (0.009) |
51 (0.010) ....>D:.510_:[..G]G-----C[..C]:.525_:D<.... (0.012) |
52 (0.018) ....>D:.511_:[..U]U-----A[..A]:.524_:D<.... (0.009) |
53 (0.011) ....>D:.512_:[..C]C-----G[..G]:.523_:D<.... (0.008) |
54 (0.016) ....>D:.513_:[..U]U-*---G[..G]:.522_:D<.... (0.007) |
55 (0.015) ....>D:.508_:[..U]U-**--A[..A]:.546_:D<.... (0.008) |
56 (0.010) ....>D:.514_:[..A]A-**--A[..A]:.521_:D<.... (0.010) |
57 (0.010) ....>D:.515_:[..G]G-**+-C[..C]:.548_:D<.... (0.008) x
58 (0.012) ....>D:.518_:[..G]G-----C[..C]:.556_:D<.... (0.032) +
Note: This structure contains 18[15] non-Watson-Crick base-pairs.
По данным в таблице выше можно определить номера нуклетидов, образующих стебли, и сопоставить их с существующими названиями (с чем помогла эта статья):
- Акцепторный: 501–507/572–566
- Т-стебель: 549–553/565–561
- Антикодоновый стебелль: 538–544/532–526
- D-стебель: 510–513/525–522
Также можно найти неканонические пары оснований:
- 502G–571U
- 554U–558A
- 555U–517G
- 538A–532C
- 544A–526G
- 513U–522G
- 508U–546A
- 514A–521A
- 515G–548C
Стекинг-взаимодействия
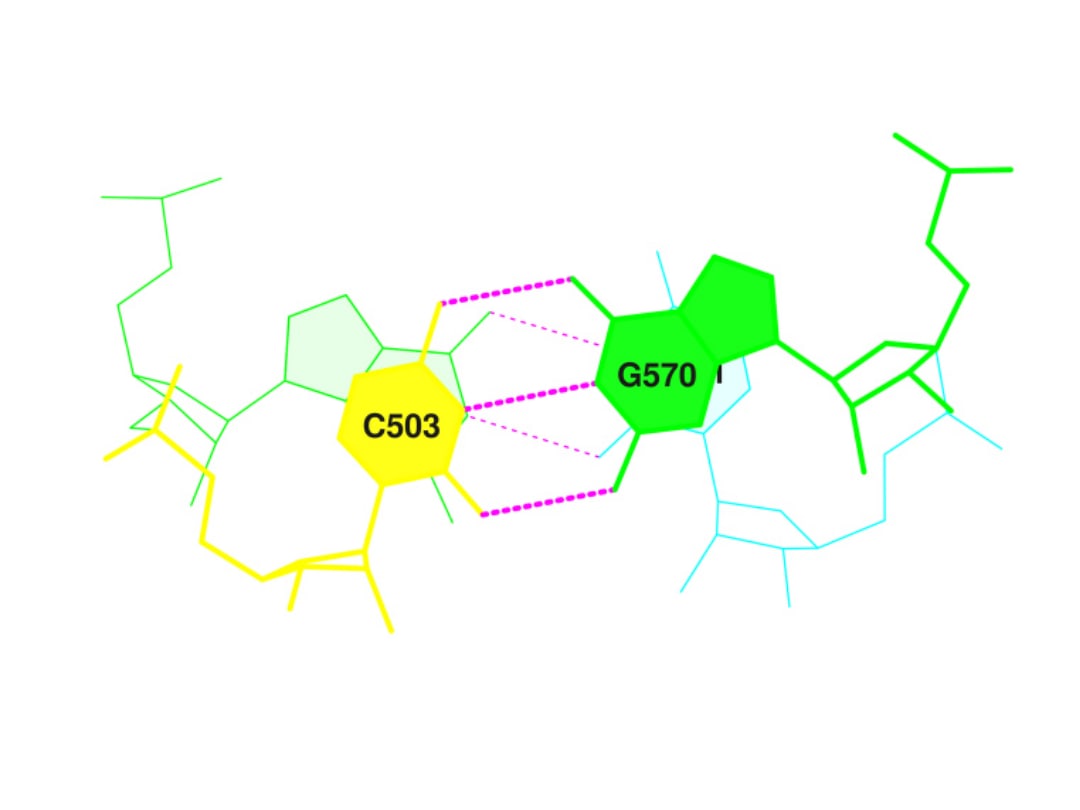
В файле 1g59_old.out в разделе Overlap area были найдены пары азотисных оснований с наибольшим перекрыванием. Они находились на втором шаге.
Далее с помощью команды ‹ex_str -2 stacking.pdb step2.pdb› была вырезана соответствующая 2 шагу структура.
На следующем шаге было построено изображение этой структуры с помощью команды ‹stack2img -cdolt step2.pdb step2.ps›. Результат после конвертации представлен на изображении ниже: