| Главная | Семестры | Обо мне | Ссылки |
|---|
Поиск структур, сходных с белком RadA (идентификатор PDB 3NTU), с помощью сервиса PDBeFold
PDBeFold - сервис, позволяющий найти и совместить полипептидные цепи, похожие на данную.
На вход требуется идентификатор PDB нужного белка и название его цепи.
Белок репарации и рекомбинации RadA состоит из 6 одинаковых цепей, файл PDB содержит информацию об одной из них (:A), так что выбирать не пришлось.
Было просмотрено 95775 записей, содержащих 247218 цепей, при этом сервис работал около 7 минут.
В результате нашлось 27 структур, одна из которых - исходная цепь белка RadA с идентификатором 3ntu.
Все находки с их параметрами можно посмотреть в таблице Excel (загрузить).
В рамке - исходная структура, желтым выделена выбранная мной для сравнения цепь. Мой выбор остановился на записи FILAMENT OF OCTAMERIC RINGS OF DMC1 RECOMBINASE FROM HOMO SAPIENS (идентификатор PDB 4hyy, цепь :D). Исходный белок RadA получен из организма археи Methanococcus voltae, а найденная цепь - структурная единица рекомбиназы человека DMC1. Биологическая единица рекомбиназы человека DMC1, состоящая из 8 таких цепей, показана на рис. 1.
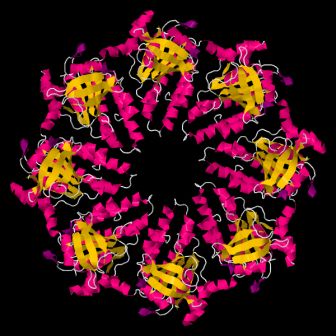
Рис. 1. Биологическая единица рекомбиназы человека DMC1. Загружено с http://www.rcsb.org/pdb/explore/explore.do?structureId=4hyy
Судя по названию, белки RadA и DMC1 выполняют одну и ту же функцию: участвуют в процессе рекомбинации ДНК. Отметим параметры сходства их структур, посчитанные сервисом PDBeFold.
Таблица 1. Сравнение структур белка RadA и цепи D рекомбиназы человека DMC1
| Параметр находки | Расшифровка параметра | Значение |
|---|---|---|
| RMSD | Root Mean Square Deviation, среднее квадратичное всех расстояний между соответствующими C-α атомами | 1,17 |
| N_align | Число сопоставленных C-α атомов в исходной структуре и в находке | 215 (из 310) |
| N_g | Число участков расхождения полипептидных цепей (N- и C-концевые расхождения не считаются) | 7 |
| %seq | Процент совпадающих аминокислотных остатков | 46 |
На рис. 2 представлено сопоставление цепей исходного белка и находки. Можно заметить, что сходство вторичных структур велико, несмотря на небольшой процент совпадения аминокислот. Также у цепи D белка DMC1 отсутствует структура, расположенная на рисунке справа от основной части белка RadA. Ранее мной была выдвинута гипотеза, что эта структура отвечает за связывание белка с ДНК. Возможно, из-за того, что DMC1 состоит из 8 цепей, его сайт связывания был перемещен на другое место.
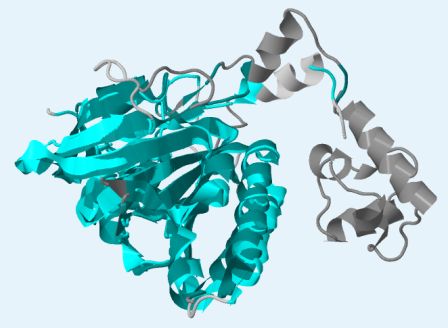
Рис. 2. Совмещение цепей белков RadA и DMC1. Голубым цветом показаны совпадающие участки структур, серым - различающиеся. Получено с помощью сервиса PDBeFold и Jmol
Рассмотрим участки цепей обоих белков для более точного сравнения структур (см. рис. 3). Я выделила в Jmol участки 80-140 цепи RadA (CHAIN1) и 101-150 цепи DMC1 (CHAIN2). Командой SELECT WITHIN(2,CHAIN1.CA) AND CHAIN2 OR WITHIN(2,CHAIN2.CA) AND CHAIN1 можно выделить все С-альфа атомы обеих цепей, которые находятся на расстоянии не более 2 ангстрем друг от друга. Для сравнения, расстояние между соседними С-альфа атомами одной цепи составляет 3,6 ангстрема. В результате было обнаружено по 56 атомов на каждой цепи, на рис. 3 они показаны шариками. Выделен также участок расхождения цепей. Внутри него цепи имеют разную длину, совпадающих аминокислот практически нет. Результат выравнивания участков, полученный с помощью сервиса http://xylian.igh.cnrs.fr/bin/align-guess.cgi.
>_ RadA 125-144 20 aa vs.
>_ DMC1 146-155 10 aa
scoring matrix: , gap penalties: -12/-2
15.0% identity; Global alignment score: -17
10 20
395641 PEFLFYDEEAVSKGEVAEPK
: . : :
_ P----------GAGGYPGGK
10
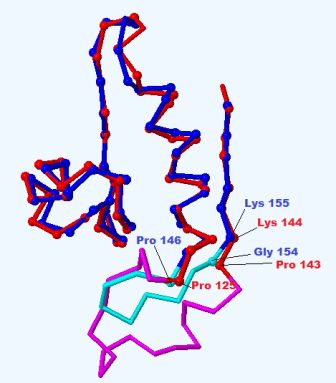
Рис. 3. Совмещение участков цепей белков RadA и DMC1. Красным цветом показана цепь RadA, синим - DMC1, сиреневым и голубым - соответственно участок расхождения цепей RadA и DMC1. Шариками отмечены С-альфа атомы обеих цепей, находящихся на расстоянии не более 2 ангстрем друг от друга. Подписаны совпадающие и несовпадающие аминокислоты на концах участка. Получено с помощью сервиса PDBeFold и Jmol