| Главная | Семестры | Обо мне | Ссылки |
|---|
Работа в Uniprot
Uniprot - это база последовательностей белков. Для каждого белка существует отдельная запись, в которой, помимо аминокислотной последовательности, есть много полезной информации. Например, это название и таксономия организма, из которого получен белок, данные о статьях, где он описан, и, что полезно, названия записей о белке в других базах данных.
В базе данных RefSeq запись о белке RadA находится под названием YP_003707477.1. Для того чтобы узнать AC (Accession number, один из идентификаторов записи белка в Uniprot), я использовала сервис ID Mapping на сайте Uniprot. Для записи о белке RadA из организма Methanococcus voltae, штамм А3: ID D7DTP4_METV3, AC D7DTP4.
Вся информация в записях Uniprot распределена на поля, каждое со своим названием, состоящим из двух букв. Есть несколько способов получить нужную информацию из записи о белке. Например, открыть файл с записью с помощью команды less в командной строке bash, или же использовать grep с указанием метки поля и того, что она начинается в первой позиции (в моем случае grep ^XX d7dtp4_metv3.entret, где XX - название нужного поля).
Ортологи белка - это белки из геномов организмов того же рода.
Для поиска ортологов моего белка в Uniprot я использовала расширенный поиск (Advanced Search). Запрос выглядит так:
name:"DNA repair and recombination protein RadA" AND taxonomy:"Methanococcus [2184]"
Результат поиска - 10 последовательностей (см. рис. 1). Далее я выбрала записи из полностью секвенированных геномов (Show only entries from a complete proteome set).
Число находок сократилось до 8, в их числе есть и исходная с AC D7DTP4.
Для дальнейшей работы нужно было выбрать двух ортологов моего белка.
Оказалось, все 8 записей содержат белки, существование которых не было подтверждено, оно лишь обосновано гомологией с известным белком.
Поэтому я решила использовать для описания один из действительно обнаруженных белков.
При этом его запись не относится к полностью секвенированным геномам, ее AC P0CW58.
Таблица с кратким описанием моего белка и двух его ортологов: загрузить.
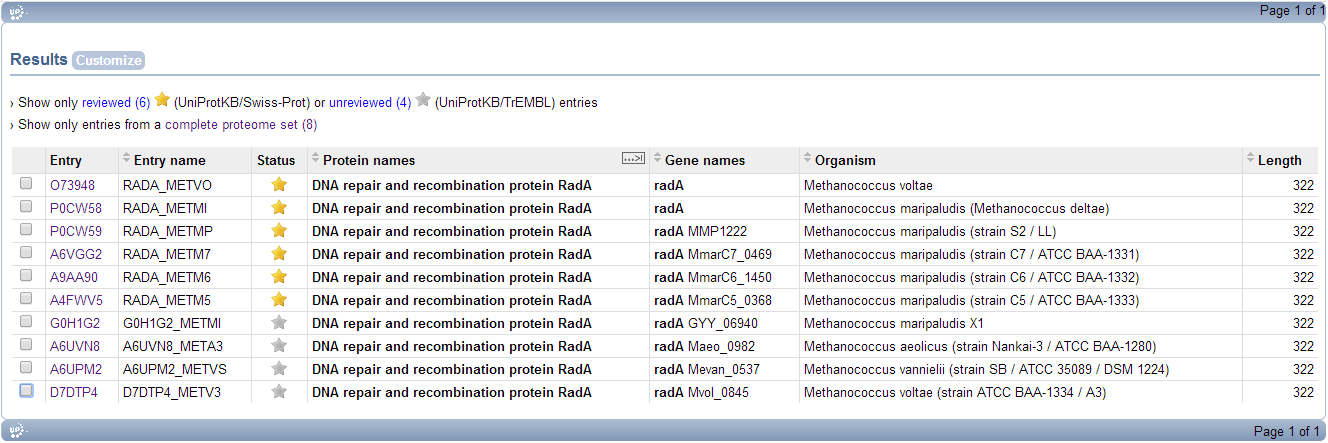
Рис. 1. Результат поиска ортологов белка RadA на сайте Uniprot.
Для того чтобы ответить на 3 вопроса из списка, выданного нам (ссылка), я использовала запись с AC P0CW58, так как она содержит больше дополнительной информации.
Вопрос №1.
Какие участки белка (напишите номера аминокислотных остатков) участвуют в связывании лиганда? Какого?
105-112 а.о. отвечают за связывание с АТФ. Все а.о. этого участка: GMFGSGKT.
Вопрос №2.
Опишите функцию и состав комплекса, в который входит данный белок.
Белок участвует в репарации и рекомбинации ДНК. Он образует филамент с одноцепочечной ДНК, после этого гидролизует молекулу АТФ и активизирует обмен участками между гомологичными цепями.
Таким образом, в состав комплекса входят: белок RadA, одноцепочечная ДНК, АТФ.
Вопрос №3.
Предложите мутации, влияющие на связывание с ДНК.
В связывании с ДНК большую роль играет пространственная структура белка.
В элементах вторичной структур я заметила повороты (TURN).
Это аминокислотные остатки №№ 91-93 (LAG) и 132-134 (LEG).
Повороты (изгибы) бывают первого и второго типов.
В изгибе 2 типа третьим аминокислотным остатком должен быть глицин, что как раз имеется в нашем случае.
На рис. 2 показан механизм образования поворотов первого и второго типов.
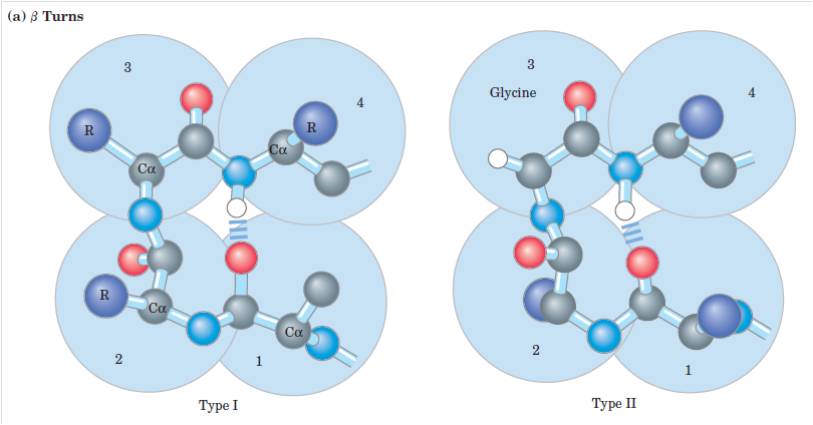
Рис. 2. Образование поворотов первого и второго типов в полипептидной цепи. Голубым цветом показаны атомы азота, серым - атомы углерода, красным - атомы кислорода, белым - водорода, синим - боковые радикалы.
Мне кажется, в данном случае поворот возникает из-за взаимных отталкиваний крупных боковых радикалов. Исходя из этого предположения, при замене этих аминокислот на ту, что содержит самый маленький боковой радикал (глицин), поворот исчезнет.
К сожалению, из трех белков только один с AC P0CW58 имеет подтвержденное существование, поэтому в его записи содержится информация о вторичной структуре белка и о сайтах связывания. С помощью сайта Uniprot я сделала выравнивание трех последовательностей и выделила те участки, о которых шла речь выше - сайт связывания АТФ и повороты. Цветом выделены только те участки, что имеют аннотацию в записи белка. Выравнивание показано на рис. 3.

Рис. 3. Выравнивание последовательностей белка RadA и его ортологов. Желтым цветом отмечены участки поворотов в структуре белка, зеленым - аннотированный сайт связывания АТФ.
Заметим, что сайт связывания АТФ довольно консервативен у всех трех последовательностей, значит, можно сделать вывод о том, что все три белка присоединяют АТФ в одном и том же месте. А вот участки поворотов менее консервативны. Первый поворот (91-93 а.о.), возможно, имеет место быть, так как замена аланина на глицин не имеет функциональной значимости, а вот насчет второго (132-134 а.о.) сказать то же самое сложно.
В базе данных Uniprot есть два раздела: Swiss-Prot и TrEMBL.
В TrEMBL находятся последовательности белков, которые были аннотированы автоматически, например, исходя из гомологии с уже известным белком.
Если же запись TrEMBL была проверена экспертом, она переходит в раздел Swiss-Prot.
Там записи более "высокого качества": как правило, они содержат дополнительную информацию, да и вероятность сущестования белка существенно выше.
На рис. 1 показаны результат поиска ортологов моего белка. Видно, что из 10 находок 6 записей принадлежат разделу Swiss-Prot, а 4 - TrEMBL. К сожалению, мой белок как раз среди этих четырех.
Работа в DOOR2
DOOR2 (Database of prOcaryotic OpeRons) - база данных предсказанных оперонов прокариот. Точность предсказаний - более 90%.
Есть и подтвержденные экспериментально опероны.
Оперон – кластер из генов, лежащих в ДНК непосредственно друг за другом и закодированных на одной цепи. Как правило, при транскрипции все гены оперона считываются в виде одной длинной молекулы мРНК, с котором потом синтезируются отдельные субъединицы. Это - один из механизмов точной регуляции нужного соотношения количеств производимых субъединиц.
В 1 семестре я изучала оперон АТФ-синтазы археи Methanococcus voltae (ссылка на страницу) с помощью геномного браузера на сайте NCBI.
Тогда я объединила в оперон все ближайшие сонаправленные гены АТФ-синтазы, всего 10 штук.
Посмотрим, выдаст ли DOOR2 тот же результат.
По запросу "Methanococcus voltae A3 atp synthase" нашлось 3 оперона, содержащих 4 гена (см. рис. 4).
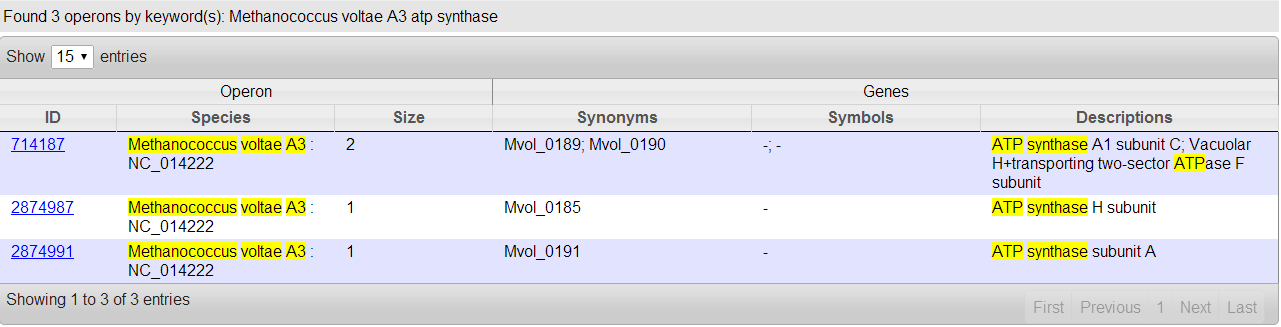
Рис. 4. Результат поиска по запросу "Methanococcus voltae A3 atp synthase" в DOOR2.
Область генома, включающая в себя опероны АТФ-синтазы, можно посмотреть здесь.
Можно сделать вывод, что предсказание DOOR2 является точным, но не полным.
Далее я попробовала найти оперон белка RadA (ссылка на работу прошлого семестра). По запросу "RadA methanococcus voltae" DOOR2 выдал 1 оперон с 1 геном Mvol_0845 (см. рис. 5).
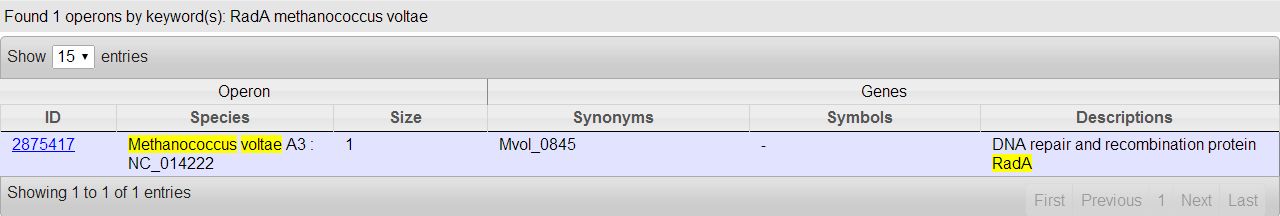
Рис. 5. Результат поиска по запросу "RadA methanococcus voltae" в DOOR2.
Область генома с данным геном можно посмотреть здесь.