| Главная | Семестры | Обо мне | Ссылки |
|---|
A- и В- формы ДНК. Структура РНК
Задание 1
Для начала с помощью программы fiber пакета 3DNA были получены PDB-файлы A-, B- и Z-форм дуплекса ДНК.
Команда для получения А-формы выглядит так:
fiber -a fiber_A.pdb
Задание 2
На рис. 1-3 показана А-форма ДНК. Требовалось выделить
- сахарофосфатный остов ДНК;
- все нуклеотиды;
- все аденины;
- атом N7 во всех гуанинах и/или только в первом по последовательности.
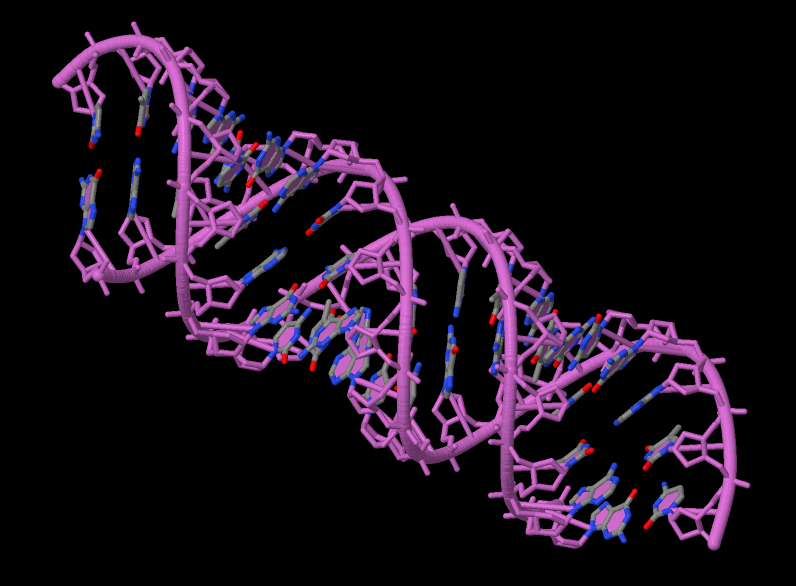
Рис. 1. А-форма ДНК. Фиолетовым выделен сахарофосфатный остов, азотистые основания покрашены по типу атомов
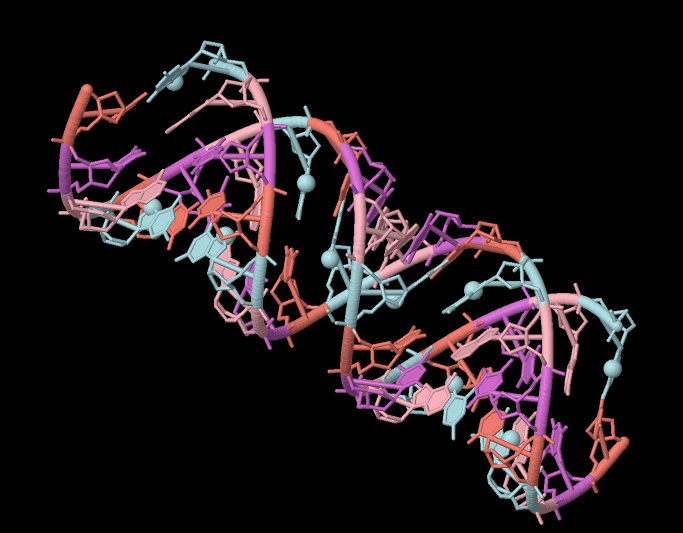
Рис. 2. А-форма ДНК. Голубым цветом покрашены нуклеотиды с гуанином, розовым - с аденином, фиолетовым - с тимином, коралловым - с цитозином. Атомы N7 гуанина показаны шариками
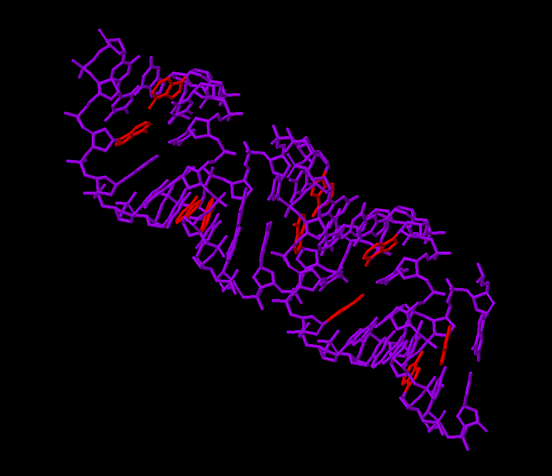
Рис. 3. А-форма ДНК. Красным цветом выделены все аденины
Далее были получены структуры ДНК и РНК с сайта PDB.
ДНК: 1LQ1.pdb
РНК: 1C0A.pdb
Проверим структуры на наличие разрывов. На рис. 4 представлена ДНК из файла 1LQ1.pdb. Есть один двуцепочечный разрыв. Ближайшие к нему азотистые основания сильно отклонены в сторону, то есть причина не в ошибке рентгеноструктурного анализа. Видимо, разрыв связан с деятельностью белка.

Рис. 4. Структура ДНК из файла 1LQ1.pdb. Есть двуцепочечный разрыв в середине
На рис. 5 показана структура т-РНК из файла 1C0A.pdb. Разрывов нет.
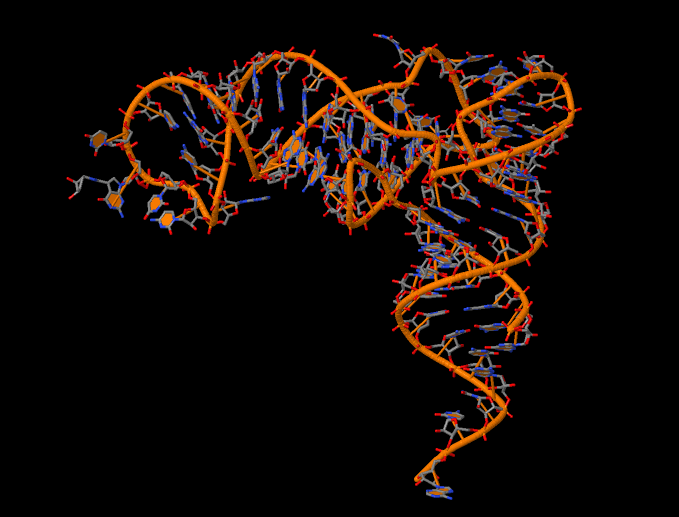
Рис. 5. Структура т-РНК из файла 1C0A.pdb. Разрывов не обнаружилось
Для дальнейшей работы сохраним координаты только ДНК и РНК в отдельных файлах.
ДНК: 1LQ1_dna.pdb
РНК: 1C0A_rna.pdb
Задание 3
A-, B- и Z-формы ДНК отличаются тем, что одни и те же атомы направлены к разным бороздкам. На рис. 6 показана структура В-формы ДНК. Выделены атомы цитозина, которые смотрят в сторону малой бороздки: O2, N1, C2. В Z-форме ДНК они тоже направлены к малой бороздке, а вот в А-форме, напротив, к большой.
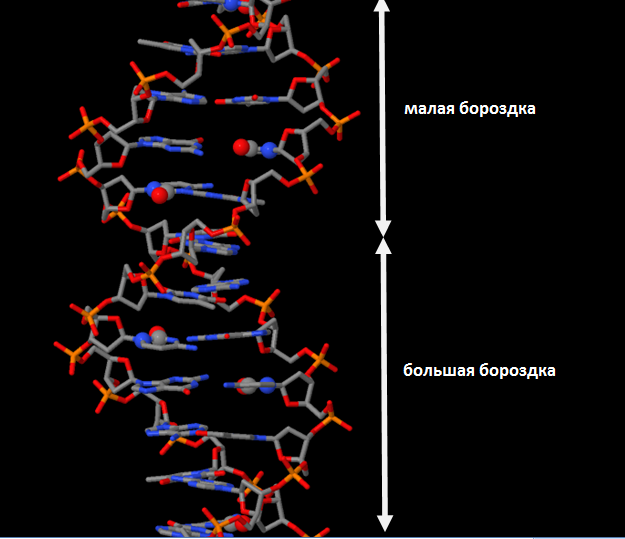
Рис. 6. В-форма ДНК. Выделены атомы цитозина O2, N1, C2
Изображение цитозина в ChemSketch приведено на рис. 7. Файл с работой в ChemSketch: загрузить.

Рис. 7. Цитозин в В-форме ДНК
Для дальнейшего сравнения A-, B- и Z-форм ДНК заполним таблицу 1.
Таблица 1. Сравнение основных параметров разных форм ДНК
| А-форма | В-форма | Z-форма | |
|---|---|---|---|
| тип спирали | правая | правая | левая |
| Шаг спирали, Å | 28,03 | 33,75 | 43,50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки, нм | 1,681 (от g9:a.p до a34:b.p) | 1,721 (от t7:a.p до t31:b.p) | 1,517 (g11:a.p c30:b.p) |
| Ширина малой бороздки, нм | 0,798 (от g9:a.p до a26:b.p) | 1,169 (от t31:b.p до a14:a.p) | 0,72 (g11:a.p g33:b.p) |
Для определения шага спирали измерялось расстояние между атомами фосфора, расположенных друг под другом. Результат показан на рис. 8.
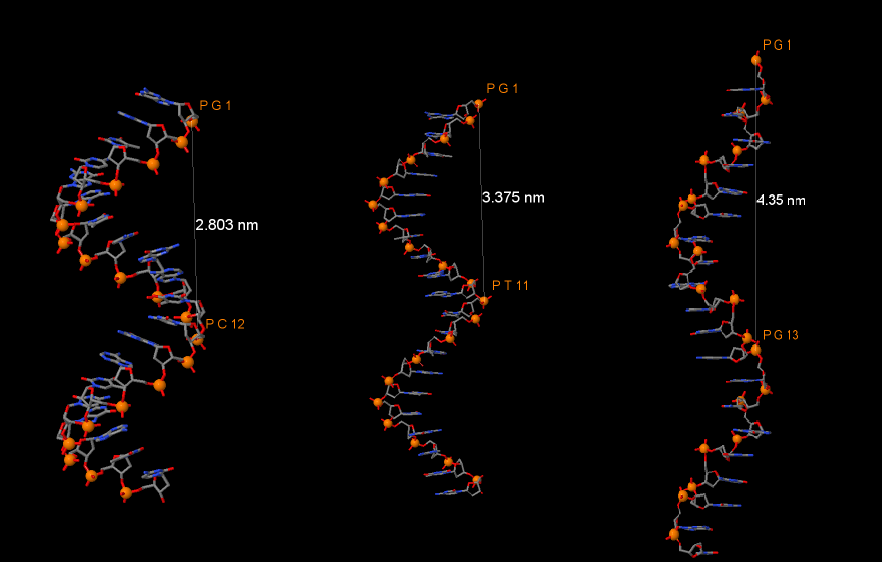
Рис. 8. Измерение шага спирали для A-, B- и Z-формы ДНК соотвестственно
Задание 4
Посчитаем торсионные углы нуклеотида с цитозином и сравним значения для А-, B- и Z-формы ДНК, а также для т-РНК, структура которой приведена выше. Результат - в таблице 2. Названия торсионных углов можно посмотреть на рис. 9.
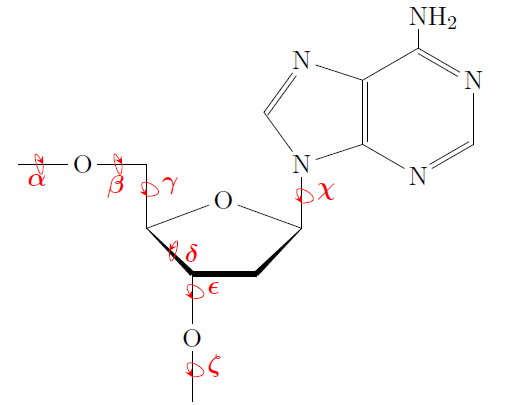
Рис. 9. Торсионные углы нуклеотида
Таблица 2. Торсионные углы в разных формах ДНК и РНК
| Форма | α | β | γ | δ | ε | ζ | χ |
|---|---|---|---|---|---|---|---|
| А-форма | -51,7 | 174,8 | 41,7 | 79 | -147,8 | -75 | -157,2 |
| В-форма | -29,9 | 136,3 | 31,2 | 143,3 | -140,8 | -160,5 | -98 |
| Z-форма | -139,5 | -136,8 | 50,9 | 137,6 | -96,5 | 82 | -154,3 |
| т-РНК | -57,5 | 179,5 | 40,7 | 81,9 | -157,2 | -71,1 | -157,3 |
В наибольшей степени различаются значения углов β и ζ. По значениям торсионных углов можно сделать вывод, что т-РНК находится в А-форме. Действительно, из-за ОН-группы она не может принимать другие формы.
Для получения информации о файлах с сайта PDB были выполнены следующие команды bash:
remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb''
find_pair -t XXXX_old.pdb stdout | analyze
В файле под названием XXXX_old.out можно найти информацию о торсионных углах каждого нуклеотида.
Для заданных ДНК и РНК данные были сведены в таблицу Excel: загрузить
В таблице желтым цветом выделены самые отклоняющиеся значения углов и номера самых "деформированных" нуклеотидов.
Также в файле XXXX.out есть информация о водородных связях между азотистыми основаниями.
Для т-РНК данные приведены в таблице 3.
Неканонические пары оснований: 604G-669U, 649G-665PSU, 655PSU-618G, 638C-632C, 644G-626A, 610G-625U, они выделены в таблице жирным.
Есть водородные связи, которые не образуют стебли: 654_5MU-658A, 655PSU-618G, 614A-608_4SU, 615G-648C, 619G-656C. Они стабилизируют структуру РНК.
Таблица 3. Водородные связи во вторичной структуре т-РНК
| Атом № | Атом № | Стебель | Спираль | |||
|---|---|---|---|---|---|---|
| 1 | 601 | G | C | 672 | акцепторный | |
| 2 | 602 | G | C | 671 | акцепторный | |
| 3 | 603 | A | U | 670 | акцепторный | |
| 4 | 604 | G | U | 669 | акцепторный | |
| 5 | 605 | C | G | 668 | акцепторный | |
| 6 | 606 | G | C | 667 | акцепторный | |
| 7 | 607 | G | C | 666 | акцепторный | |
| 8 | 649 | G | PSU | 665 | Т | |
| 9 | 650 | C | G | 664 | Т | |
| 10 | 651 | G | C | 663 | Т | |
| 11 | 652 | G | C | 662 | Т | |
| 12 | 653 | G | C | 661 | Т | |
| 13 | 654 | 5MU | A | 658 | ||
| 14 | 655 | PSU | G | 618 | Смена цепи | |
| 15 | 638 | C | C | 632 | антикодоновый | |
| 16 | 639 | G | C | 631 | антикодоновый | |
| 17 | 640 | C | G | 630 | антикодоновый | |
| 18 | 641 | A | U | 629 | антикодоновый | |
| 19 | 642 | G | C | 628 | антикодоновый | |
| 20 | 643 | G | C | 627 | антикодоновый | |
| 21 | 644 | G | A | 626 | антикодоновый | |
| 22 | 610 | G | U | 625 | D | |
| 23 | 611 | U | A | 624 | D | |
| 24 | 612 | U | A | 623 | D | |
| 25 | 613 | C | G | 622 | D | |
| 26 | 614 | A | 4SU | 608 | ||
| 27 | 615 | G | C | 648 | Смена цепи | |
| 28 | 619 | G | C | 656 | Изолированная пара |
Используя информацию о стекинг-взаимодействиях из файла XXXX.out, можно найти пары азотистых оснований с максимальной площадью перекрывания. Чтобы получить изображение стекинг-взаимодействия для т-РНК, были выполнены команды:
ex_str -12 stacking.pdb step12.pdb
stack2img -cdolt step12.pdb step12.ps
Полученная картинка - на рис. 10.
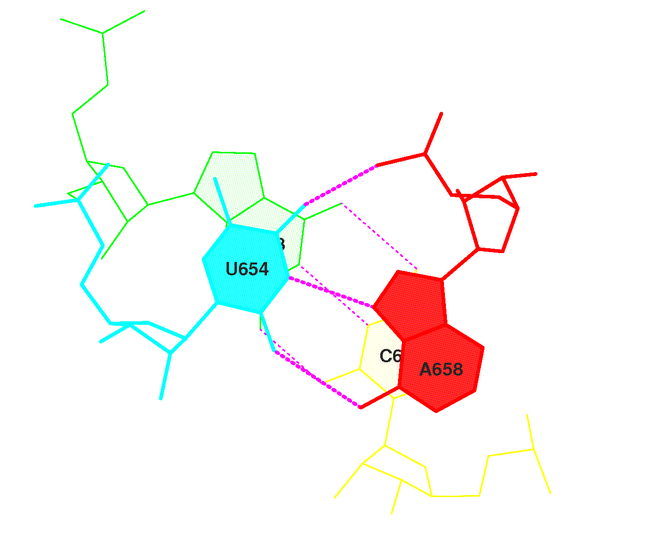
Рис. 10. Стекинг-взаимодействие азотистых оснований т-РНК с максимальной площадью перекрывания