| Главная | Семестры | Обо мне | Ссылки |
|---|
Восстановление предкового состояния доменной архитектуры
Выбор объектов изучения
Для восстановления предкового состояния доменной архитектуры был выбран домен Rad51 белка, с которым я работала в курсе биоинформатики, а именно белка репарации и рекомбинации RadA. В работе второго семестра было выполнено описание этого домена: ссылка на работу. Некоторую дополнительную информацию о домене можно найти в Pfam по идентификатору PF08423.
Всего известно 29 доменных архитектур, в которые входит Rad51. Таблица со всеми структурами на сайте Pfam: здесь. Из этой таблицы следует, что преобладающее большинство белков содержат либо только домен Rad51, либо два домена: Rad51 и HHH_5. HHH_5 (Helix-Hairpin-Helix) - это неспецифический ДНК-связывающий домен. Так как белки с доменом Rad51 отвечают за репарацию и рекомбинацию ДНК, наличие ДНК-связывающего домена для них весьма желательно. Как показывает таблица с доменными архитектурами, действительно, большинство белков с Rad51 содержат также домен HHH_5. Из всех доменных архитектур были выбраны две самые распространенные: только домен Rad51 (1253 последовательности в Pfam) и Rad51+HHH_5 (586 последовательностей Pfam).
Белки с доменом Rad51 встречаются у архей и эукариот.
Я решила выбрать два класса архей с таким доменом: Archaea - Euryarchaeota - Methanococci&Methanomicrobia, так как в классе Methanococci находится организм Methanococcus voltae, с которым я работала в курсе биоинформатики. Было решено взять последовательности, представляющие структуру с единственным доменом, из Methanococci, а структуру с двумя доменами - из Methanomicrobia.
Также к выбранным последовательностям была добавлена еще одна, с двумя доменами и известной 3D-структурой (идентификатор PDB 3NTU), из организма Methanococcus voltae.
Длины доменов в обоих случаях составляют около 200 аминокислот.
Последовательности хорошо выравниваются относительно друг друга: много консервативных позиций, мало гэпов.
Это неудивительно для двух близкородственных классов.
Таблица Excel всего практикума: загрузить
На листе "Домен" можно найти описание всех белков из Uniprot, включающих домен Rad51. Лист "Общая таблица" - таблица всех доменных архитектур с указанием таксономии. На лист "Выбранные археи" перенесены отобранные последовательности.
Проект JalView с выравниванием отобранных последовательностей: загрузить
В проекте использована раскраска по группам ClustalX с порогом консервативности 70%.
Добавлена 3D-структура для последовательности RadA_METVO.
Окно "rad51_to_tree" - это выравнивание только домена Rad51.
Построение филогенетического дерева
Дерево было построено с помощью MEGA алгоритмом Maximum Likelihood, с количеством bootstrap реплик, равном 100.
К последовательностям были добавлены коды: 1 для однодоменной архитектуры, 2 для двухдоменной.
С обозначает представителей Methanococci; M - Methanomicrobia. Результат показан на рис. 1.
Дерево с указанными длинами ветвей: загрузить PDF
Скобочная формула дерева: загрузить
Как видно из дерева, два класса находятся в разных кладах. Красным выделена ветвь, отделяющая класс Methanomicrobia, а таже ветвь, отделяющая организмы с двухдоменной архитектурой. К предковой форме, видимо, ближе класс Methanococci с однодоменной архитектурой, а домен HHH_5 был приобретен позднее. Также интересно отметить, что две последовательности организма Methanococcus voltae (RADA_METVO и D7DSA9_METV3) находятся довольно обособленно от других последовательностей. Одна из этих последовательностей содержит два домена, вторая - один. В классе Methanococci действительно есть представители с двухдоменной архитектурой. Скорее всего, здесь имеет место ошибка аннотации: не был найден домен HHH_5 в последовательности D7DSA9_METV3.
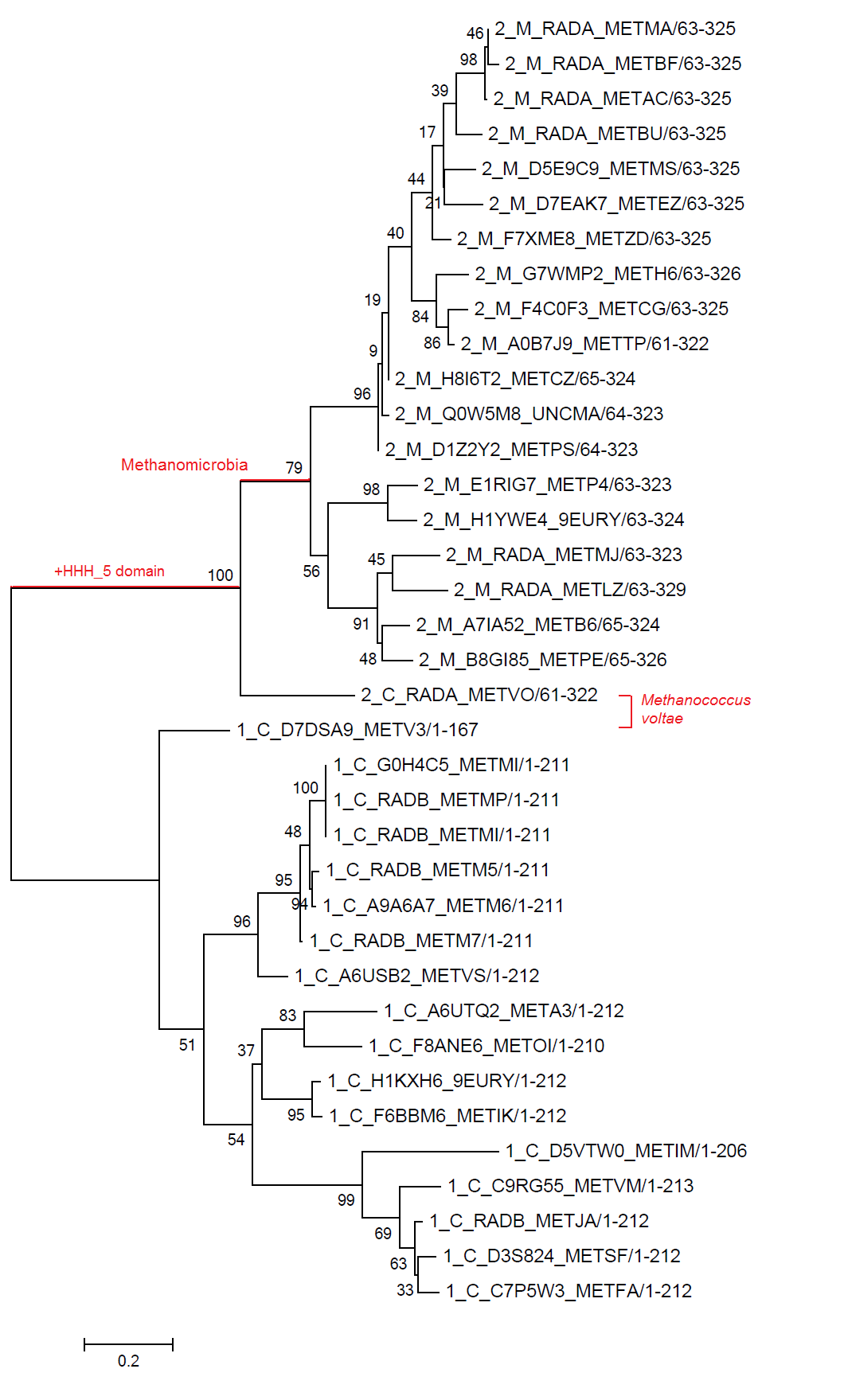
Рис. 1. Филогенетическое дерево домена Rad51. Получено с помощью MEGA
Построение профиля подсемейства
Для построения профиля HMM были выбраны последовательности с двухдоменной архитектурой, т.к. они образуют отдельную кладу на дереве.
Кроме того, по профилю был проведен поиск среди всех последовательностей из Uniprot с доменом Rad51.
Файл с выравниванием последовательностей подсемейства: загрузить.
Файл со всеми последовательностями из Uniprot, содержащими домен Rad51: загрузить.
Команды для построения профиля HMM с помощью пакета HMMER 2.3.2:
hmm2build hmm.out m2.fasta
hmm2calibrate hmm.out
hmm2search hmm.out all_seqs.fasta > hmm.txt
Профиль HMM: загрузить.
Найденные по профилю последовательности: загрузить.
По профилю нашлись все 20 последовательностей выбранного подсемейства. В таблице Excel на листе "Данные HMM" эти находки выделены зеленым цветом.
На рис. 2 представлена ROC-кривая, характеризующая поиск по построенному профилю.
Так как E-value худшей находки составляет 1,3E-153, неудивительно, что кривая выглядит таким образом: слишком "сильный" получился профиль HMM.
Скрипт Python для получения изображения: загрузить.
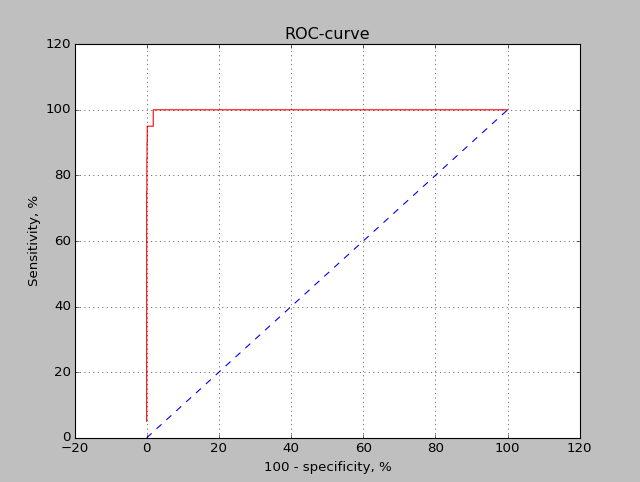
Рис. 2. ROC-кривая (красного цвета) и прямая y=x для сравнения (синего цвета)
Пороговое значение E-value отмечено на листе таблицы Excel "Данные HMM" красным цветом. В таблице 1 приведена информация о находках с учетом выбранного порога. Как уже было сказано, полученный профиль HMM очень "сильный", то есть скорее можно ожидать ложноотрицательный результат, чем ложноположительный.
Таблица 1. Качество предсказаний, полученных с помощью построенной модели
| На самом деле | принадлежит подсемейству | не принадлежит | сумма |
|---|---|---|---|
| Выше порога по профилю | 18 | 3 | 21 |
| Ниже порога | 2 | 1978 | 1980 |
| Сумма | 20 | 1981 | 2001 |