
Цель данной работы - создать множественное выравнивание белков-гомологов CRH_BACSU. Для получения выравнивания нужно, в первую очередь, создать выборку анализируемых белков, что и описано в первом разделе. Процессу создания собственно выравнивания посвящен второй раздел.
Процесс поиска был разбит на два этапа: создание черновой выборки, состоящей из любых более или менее подходящих последовательностей и
дальнейшая ее "чистка" - выкидывания слабо гомологичных последовательностей (например,- белков-паралогов) при пробных выравниваниях.
В свою очередь, процесс поиска гомологов подразделялся на поиск по прокариотам и, для удобства, по эукариотам - отдельно.
Для прокариот параметры поиска BLAST по таксонам были следующими: Eukarya exclude, Firmicutes exclude (см. таблица 1).
Исключение прокариотического филума Firmicutes связано с тем, что сама B. subtilis относится к этому таксону и поэтому, следует ожидать, что
для полученных хитов гомология будет настолько высокой с CRH_BACSU, что полученное выравнивание будет слабо информативным.
Т.е. из него невозможно будет получить никакой информации о широком полиморфизме белка с одной стороны, и о его консервативности
с другой стороны.
Примерно представляя объемы информации по гомологам данного белка из предыдущего задания по BLAST ,
сразу был поставлено количество хитов, равное 5000. При пороге e-value=5 выводилось 3210 хитов (см. таблицу 1).
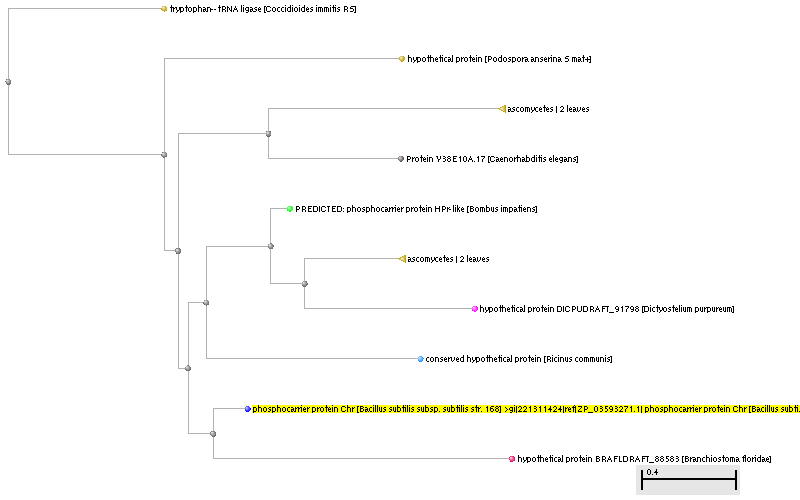
Для гомологов было построено филогенетическое дерево (рис. 1), которое, однако, не было интерактивным - невозможно было переходить
по таксонам-ветвям.

|
|
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | blastp (protein-protein BLAST) | Reference proteins (refseq_protein) | Eukarya, Fermicures - exclude | 5 | 5000 |
| По прокариотам | blastp (protein-protein BLAST) | Reference proteins (refseq_protein) | Филлумы, перебором | 3 | 10 |
| По эукариотам | blastp (protein-protein BLAST) | Reference proteins (refseq_protein) | только Eukarya | 10 | 100 |
Результатом описанных манипуляций стала сформированая черновая выборка (таблица 2). В выборке содержится 7 архейных белков, 13 эукариотических и 37 бактериальных гомологов. В сумме - 57 последовательностей.
| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | Euryarchaeota | Halococcus hamelinensis 100A6 Haloferax volcanii DS2 |
2 |
| Korarchaeota | Candidatus Korarchaeum cryptofilum OPF8 | 2 | |
| Nanoarchaeota | Nanoarchaeum equitans Kin4-M | 1 | |
| Thaumarchaeota | Candidatus Nitrosoarchaeum limnia SFB1 Nitrosopumilus maritimus SCM1 |
2 | |
| Bacteria | Actinobacteria | Atopobium rimae ATCC 49626 Rubrobacter xylanophilus DSM 9941 |
2 |
| Aquificae | Hydrogenobaculum sp. HO Hydrogenobaculum sp. Y04AAS1 |
2 | |
| Chloroflexi | Chloroflexus aurantiacus J-10-fl Ktedonobacter racemifer DSM 44963 |
2 | |
| Cyanobacteria | Oscillatoriales cyanobacterium JSC-12 Moorea producens 3L |
2 | |
| Deferribacteres | Flexistipes sinusarabici DSM 4947 Deferribacter desulfuricans SSM1 |
2 | |
| Deinococcus-Thermus | Deinococcus maricopensis DSM 21211 Deinococcus geothermalis DSM 11300 |
2 | |
| Dictyoglomi | Dictyoglomus thermophilum H-6-12 Dictyoglomus turgidum DSM 6724 |
2 | |
| Elusimicrobia | Elusimicrobium minutum Pei191 uncultured Termite group 1 bacterium phylotype Rs-D17 |
2 | |
| Fibrobacteres/Acidobacteria group | Fibrobacter succinogenes subsp. succinogenes S85 Candidatus Chloracidobacterium thermophilum B |
2 | |
| Fusobacteria | Sebaldella termitidis ATCC 33386 Leptotrichia goodfellowii F0264 |
2 | |
| Gemmatimonadetes | Gemmatimonas aurantiaca T-27 | 1 | |
| Nitrospirae | Leptospirillum ferriphilum ML-04 Candidatus Nitrospira defluvii |
2 | |
| Planctomycetes | Rhodopirellula baltica SH 1 Rhodopirellula baltica WH47 |
2 | |
| Proteobacteria | Thiobacillus denitrificans ATCC 25259 Allochromatium vinosum DSM 180 |
2 | |
| Spirochaetes | Turneriella parva DSM 21527 Spirochaeta thermophila DSM 6192 |
2 | |
| Synergistetes | Anaerobaculum mobile DSM 13181 Anaerobaculum hydrogeniformans ATCC BAA-1850 |
2 | |
| Tenericutes | Mycoplasma synoviae 53 Mycoplasma suis (strain KI_3806 |
2 | |
| Thermodesulfobacteria | Thermodesulfatator indicus DSM 15286 | 2 | |
| Thermotogae | Thermotoga naphthophila RKU-10 Thermotoga sp. RQ2 |
2 | |
| Eukaryotes | Metazoa | Bombus impatiens Amphimedon queenslandica Drosophila simulans Caenorhabditis elegans |
3 |
| Fungi | Aspergillus oryzae RIB40 Aspergillus flavus NRRL3357 Gibberella zeae PH-1 |
3 | |
| Viridiplantae | Ricinus communis Medicago truncatula |
2 | |
| Amoebozoa | Dictyostelium purpureum | 1 | |
| Diplomonadida | Giardia lamblia ATCC 50803 | 1 | |
| Choanoflagellida | Monosiga brevicollis MX1 | 1 | |
| Alveolata | Paramecium tetraurelia strain d4-2 Tetrahymena thermophila |
2 |
После составления выборки, были получены fasta-файлы последовательностей гомологов (ссылка на исходник).
Файл с последовательностями использовался для получения множественного выравнивания
при помощи программы Muscle .
Черновое выравнивание оказалось очень большим (рис. 3 полноразмерное изображение можно найти по этой ссылке ),
однако уже на нем различимы консервативные блоки.
 Рис 3. Начальной выравнивание. ( полноразмерное изображение можно найти по этой ссылке)
Рис 3. Начальной выравнивание. ( полноразмерное изображение можно найти по этой ссылке)
В процессе улучшения, из выравнивания были удалены некоторые последовательности, очевидно негомологичные CRH_BACSU:
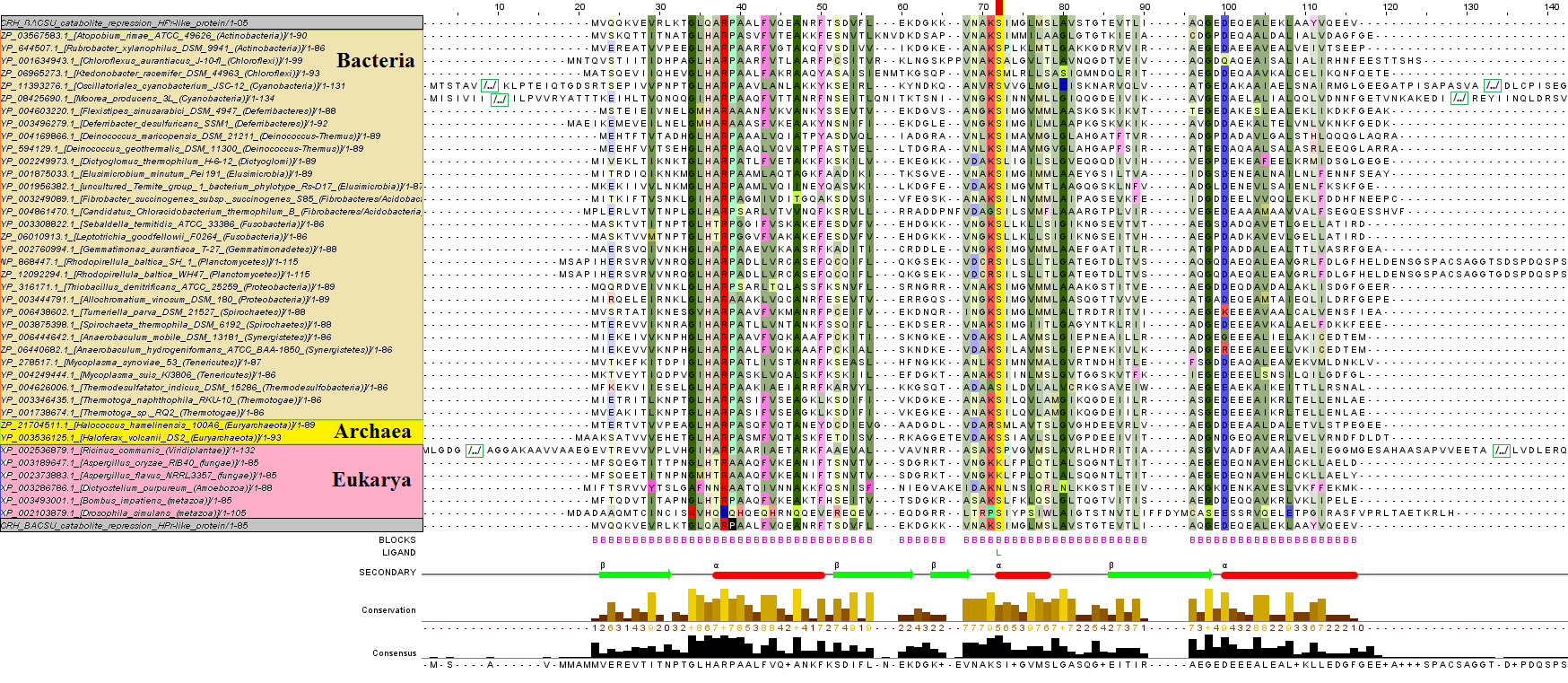 Рис 4. Оптимизированное выравнивание. ( полноразмерное изображение можно найти по этой ссылке ).
Рис 4. Оптимизированное выравнивание. ( полноразмерное изображение можно найти по этой ссылке ).
Из выравнивания очевидно, что белок CRH_BACSU черезвычайно консервативный: среди всех групп организмов гомологи очень слабо отличаются между собой.
В основном, невыровненными остаются концевые участки последовательностей, тогда как кор молекулы похож у всех гомологов.
Это, возможно, связано с тем, что белок представляет собой плотную глобулу, образованную преимущественно серединой молекулы и поэтому
такая структурно-значимая часть является более консервативной.
Интерес представляет также второй участок без гэпов, который четко выражен, но при этом не имеет консервативной структуры.
В множественном выравнивании имеется несколько колонок гэпов, которые, однако, никак не коррелируют с элементами
вторичной структуры (аннотация 3 рис 4).
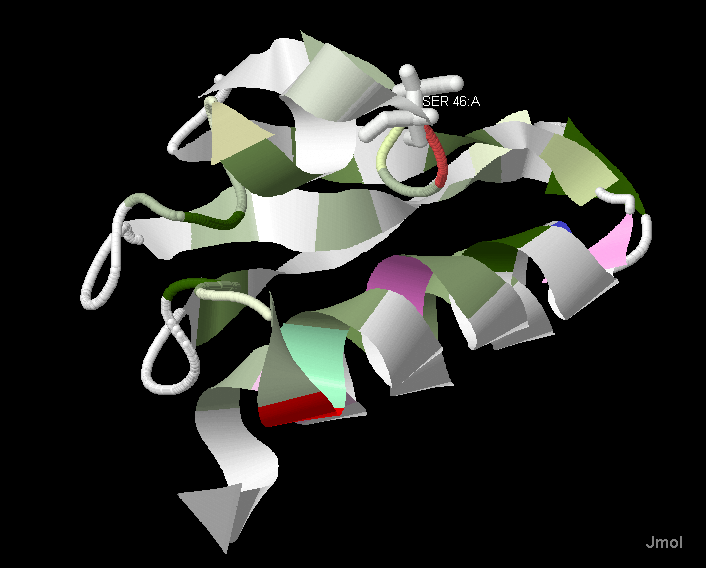
При помощи JalView можно связать последовательность fasta с трехмерной структурой того же белка из PDB (рис 5). При помощи такого инструмента удобно
искать связи между консервативностью определенных блоков или позиций и расположением их на пространственной структуре молекулы.
Уникальность белка CRH_BACSU в том, что за связывания фосфата отвечает одна единственная аминокислота - 46 серин, про роль
других ничего не известно.
В нашем случае, была попытка проанализировать свойства 46 серина, связывающего лиганд - фосфат. Однако, ни с точки зрения консервативности
этой позиции в выравниании (это не самая консервативная позиция в белке), ни с точки зрения расположения аминокислоты на пространственной структуре
белка (см. рис 5) ничего установить не удалось.
 |