
участвующих в переносе фосфата и являющихся частью фосфоенолпируват зависимой
фосфотрансферазной системы (phosphotransferase system (PTS)) транспорта сахаров у бактерий
Для поиска родственных белков, а также для выяснения возможных функций неизвестного белка можно провести анализ его последовательности. В результате мы получим набор более или менее консервативных сайтов, мотивов, доменов. Что это такое?
Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов | Положение в последовательности белка CRH_BACSU | Клан |
| 1. | PF00381 | PTS-Hpr | Семейство доменов названо по семейству маленьких цитоплазматических белков, участвующих в переносе фосфата и являющихся частью фосфоенолпируват зависимой фосфотрансферазной системы (phosphotransferase system (PTS)) транспорта сахаров у бактерий |
1–84 | - |
Как видно из данных таблицы, весь белок CRH_BACSU является единым доменом PTS-Hpr. Для этого домена не удалось найти клана (суперсемейства)
к которому он относился бы. Возможно, это отдельно стоящее семейство доменов, характерных для консервативных белков-переносчиков фосфата.
При помощи Pfam можно построить множественные выравнивания хитов. Для примера выравнены только участки, содержащие домены PTS-Hpr в хитах. Файл с выравниваниями
Считается, что именно домены это структурно-функциональные единицы эволюции белков. Т.е. если домены встретились вместе в одном белке,
то это еще ничего не означает: в других белках они могут быть организованы в другую архитектуру или вообще встретиться по отдельности.
В этом разделе мы проанализируем встречаемость некоторых доменов в белках.
Белок CRH_BACSU не очень подходит для этой цели: у него есть всего 1 домен в структуре и его встречаемость мы уже описали в таблице 1.
В качестве альтернативы мы возьмем белок Q1C131_YERPA -переносчик фосфата в фосфоенолпируват зависимой
фосфотрансферазной системе у Yersinia pestis (возбудитель чумы).
Доменное строние этого белка приведено на рисунке 1, как видно, оно несколько разнообразнее, чем у CRH_BACSU.

Рис 1. Доменная архитектура белка Q1C131_YERPA
Для сравнения мы возьмем количество белков, архитектура которых содержит только один выбранный домен (и больше ничего).
Первый домен - уже знакомый нам PTS-Hpr, второй - PTS_EIIA_2, который также относится к фосфоенолпируват зависимой
фосфотрансферазной системе переноса сахаров, но только EIIA 2.
| Домен | Архитектура белков | Количество белков с такой архитектурой |
| PTS-Hpr |  |
5718 |
| PTS_EIIA_2 |  |
9065 |
Как видно из данных таблицы 2, существует множество белков, имеющих архитектуру из единственного домена.
Банк Pfam предоставляет возможность создать дерево, на котором будут обозначены организмы и таксоны,
в которых встречаются белки с данным доменом.
Однако, подобная операция невозможна для широко распространенных доменов: например для домена PTS-Hpr, при запросе дерева, было выдано
сообщение: This family has 3582 species, which means that displaying the tree is not feasible.
If you really need to see a representation of the tree for this entry, please contact us and we will be happy
to discuss ways to generate it for you. (т.е. этот домен встречается в белках 3582 видов и такой объем
невозможно отобразить на одном дереве)
В качестве альтернативы дереву, сервис PFAM предлагает круговое (розеточное) отображение таксонов (Sunburst) - см рисунок 2.
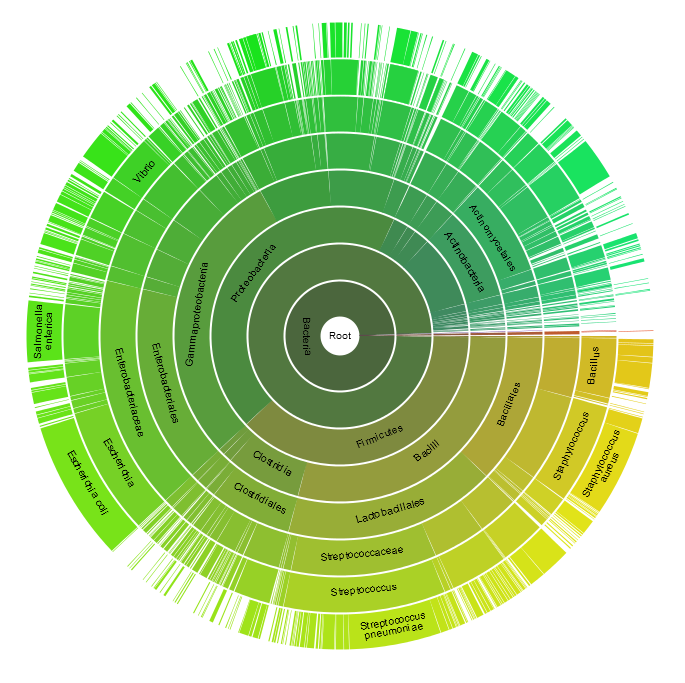
Рис 2. Розеточное отображение разнообрязия видов, имеющих белки с доменом PTS-Hpr.
Аналогичная ситуация для некоторых других, рандомно выбранных, доменов (например по PTS_EIIA_2 более 3000 видов не отображается).
Наконец, прошел анализ по домену NACHT (PF05729) - 661 вид. Этот домен отвечает за нуклеозидтрифосфатазную активность,
встречается во множестве белков с совершенно различными функциями.
|
Таксон
|
Количество белков с доменом NACHT.
|
|
| Эукариоты (Eukariota) | Зеленые растения (Viridiplantae) | 3 |
| Грибы (Fungi) | 84 | |
| Животные (Metazoa) | 117 | |
| Остальные эукариоты | 20 | |
| Археи (Archaea) | 11 | |
| Бактерии (Bacteria) | 426 | |
| Вирусы (Virus) | 0 | |
Рассуждения о распространенности домена NACHT среди организмов
Домен NACHT встречается во всех известных доменах клеточных организмов: в бактериях, археях и эукариотах. Его встречаемость в бактериях и
эукариотах соизмерима (см. таблицу 3), в археях - меньше. Для вирусных белков присутствие домена не зарегистрировано.
InterPro (an integrated resource of protein families, domains and functional sites) это интегрированная база данных,
собранная по данным из Pfam, Prosite, etc. о семействах белков, доменах, мотивах, сайтах и т.п.
Для белка CRH_BACSU была получена карта всех мотивов, сайтов и доменов описаных в InterPro (см рис 3).
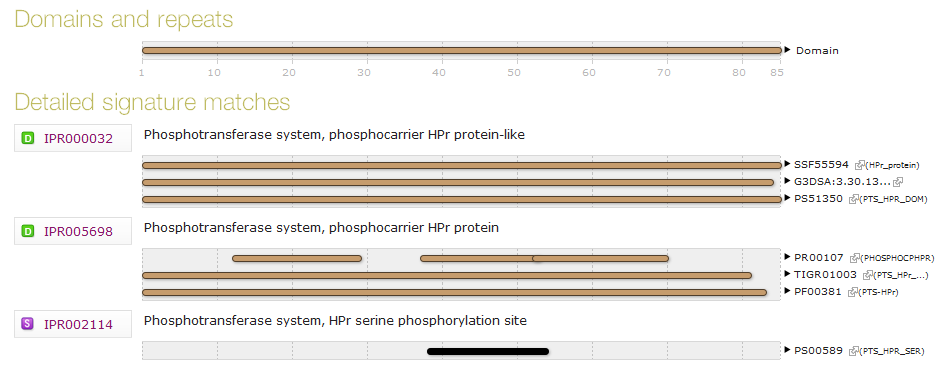
Рис 3. Карта мотивов, сайтов и доменов описаных в InterPro для белка CRH_BACSU
Самый короткий мотив на карте - PTS_HPR_SER (АС PS00589), описаный в банке PROSITE.
Он состоит из 16 аминокислот и является сайтом фосфорилирования серина в домене PTS Hpr.
Самый длинный мотив из 85 аминокислот - собственно домен белка, т.е. весь белок (SSF55594 в банке SUPERFAMILY,
PS51350 в банке PROSITE).
В InterPro также интегрированы известные структуры белков из разных банков.
Так, для CRH_BACSU приведены 3 полностью идентичные структурные черты (Structural features): из PDB, из SCOP
(The Structural Classification of proteins) и из CATH. Все они представляют собой полные 85 аминокислотные домены.
Однако, как мы помним, PFAM предлагал нам 84 аминокислотный, неполный (обрезанный с краю) домен. Последняя аминокислота потерялась, скорее
всего потому, что она наиболее вариабельна для этогй группы белков. Поэтому, при составлении множественного выравнивания и выделения
эволюционных доменов PFAM искусственно укоротил структурный домен.