Сравнение пространственной структуры белков дегидратазы (PDB-идентификатор 4E3E) и rv0216 (PDB-идентификатор 2BI0)
Для поиска схожих по структуре белков и совмещения их полипептидных цепей пользуются сервисом PDBeFold. Полученные совмещённые структуры можно просмотреть с помощью программы Jmol. Две полученные цепи соответствуют последовательности каждого из двух сравниваемых белков и отображаются одновременно. Таким образом можно найти участки сходства и расхождения в трёхмерных структурах.
Сравнение структур используется не только для поиска различий в структурах белков со схожими функциями, но также и для самого предположения этих функций.
Входным параметром для поиска совмещения была структура цепочки А белка дегидратазы из организма бактерии Chloroflexus aurantiacus, штамм J-10-fl (PDB-идентификатор 4E3E). Из 97183 было найдено 68 более-менее похожих на данную структур. Среди полученных находок наиболее подходящей явилась цепочка А из белка rv0216 из генома бактерии Mycobacterium tuberculosis штамм H37Rv (PDB-идентификатор 2BI0). Параметры, которые были получены в результате поиска и по котором проводился отбор наиболее подходящего для сравнения кандидата, этого белка приведены в Таблице 1.
| Наименование параметра | Значение параметра |
| %seq (процент совпадающих аминокислотных остатков среди всех сопоставленных) |
27 |
| Nalign (число сопоставленных Cα-атомов во входной цепочке и в находке) |
289 (82,3% от числа остатков в исходной структуре) |
| RMSD (мера сходства структур) |
1,65 |
| Ng (число участков расхождения полипептидных цепей) |
17 |
Rv0216 — предполагаемый белок (conserved hypothetical protein) из генома бактерии Mycobacterium tuberculosis штамм H37Rv. К группе conserved hypothetical protein относят белки, кодирующие гены которых присутствуют в геноме бактерии, но при этом функция этих белков неизвестна.
Пространственная структура данного белка очень схожа со структурой гидратаз, которые катализируют R-специфичную гидратацию 2-эноил коэнзима А бактерий и эукариот [1].
Организмы, из которых были получены белки rv0216 и дегидратаза, относятся к разным родам и видам. В то же время домен найденного белка относится к семейству доменов типа MaoC (MaoC-like domain), что характерно и для дегидратазы.
Исходя из сказанного выше, можно предположить, что белок rv0216 выполняет функцию, схожую с функцией белка дегидратазы.
Участок совмещения цепей (для цепи А, соответствующей rv0216, — от 210 до 227 аминокислотного остатка; для цепи В, соответствующей дегидратазе, — от 216 до 231) представлен на Рисунке 1. Фиолетовым цветом изображена цепочка белка дегидратазы, зелёным — rv0216. На данном участке хорошо совмещаются 14 аминокислотных остатков, два из которых абсолютно совпадают (это лейцин и аргинин).
Для того, чтобы получить данное изображение я использовала скрипт, созданный мной самой. Скачать исходный pdb-файл с последовательностями можно отсюда.
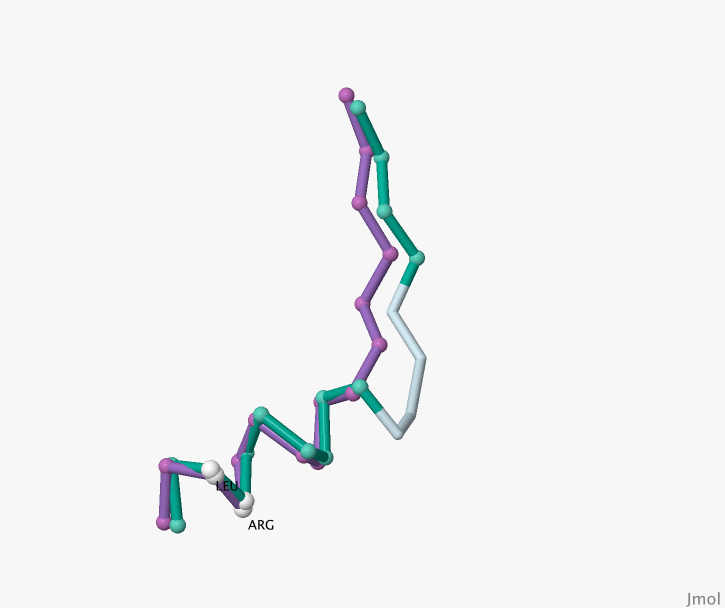
Рисунок 1. Участок сравнения полипептидной последовательности белка дегидратазы цепочка А (PDB-идентификатор 4E3E) и белка rv0216 цепочка А (PDB-идентификатор 2BI0).
Фиолетовым цветом обозначена цепочка, соответствующая дегидратазе, зелёным — белка rv0216. Голубым отмечен участок расхождения, белым цветом выделены Cα-атомы совпадающих одинаковых аминокислотных остатков.
Изображение получено с помощью сервиса PDBeFold и программы J-mol.
Исходя из Рисунка 1, "геометрическое ядро" (т.е. наиболее хорошо совмещаемые Cα-атомы) состоит из 14 атомов. Наиболее хорошо совмещаемыми атомами считатались те Cα-атомы, расстояние между которыми не превышало 1,75 Å.
Структуры других белков, которые похожи на структуру белка дегидратазы
На сайте PDB существует огромное количество различных структур белков. Для того, чтобы найти схожие с данным белком структуры, можно воспользоваться "Advanced search". Критерии поиска, использованные мной, приведены ниже в Таблице 2.
В результате было найдено 8 структур, соответствующих критериям, которые приведены ниже. Таблицу в формате Excel с характеристикой данных структур можно скачать отсюда.
| Наименование графы | Выбранное значение |
| Structure Title | homing endonuclease |
| Protein Symmetry | C3 and RMSD is between the default min 0.0 and the default max 7.0 |
| Chain Type | there is a Protein but not any DNA or RNA or Hybrid |
| Experimental Method | X-RAY and has Experimental Data |
| Resolution | between 1.5 and 2.5 |
| Ligand Search | Has free ligands=yes |
[1] Rv0216, a conserved hypothetical protein from Mycobacterium tuberculosis that is essential for bacterial survival during infection, has a double hotdog fold.
Protein Sci. 2005 Jul ;14(7):1850-62.