A-, В-, Z-формы ДНК. Структура РНК
С помощью программы fiber пакета 3DNA были построены модели структур A-, B- и Z-формы ДНК. Последовательность каждой нити дуплекса — это 5 раз повторенная последовательность "GATC" (для Z-формы — 10 раз "GC"). Скачать структуры A-формы, B-формы, Z-формы можно по соответствующим ссылкам. Полученные структуры можно увидеть на Рисунках 5а, b, c с уже сделанными измерениями.
Для работы в Jmol по выделению различных атомов и химических групп был выбран файл с А-формой ДНК. Полученный результат представлен на Рисунке 1. Скрипт, с помощью которого было получено следующее изображение можно загрузить по ссылке.
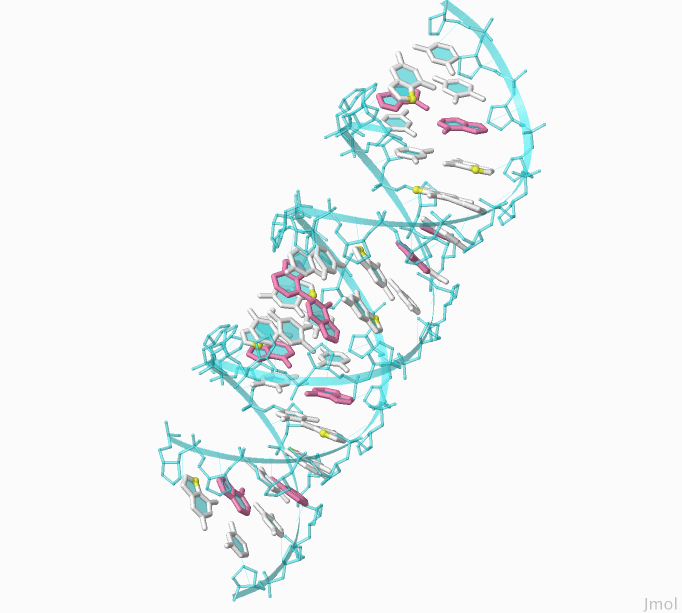
Рисунок 1. А-форма ДНК.
Сахарофосфатный остов ДНК окрашен голубым цветом, белым цветом выделены все нуклеотиды. Аденины представлены в розовом цвете.
Жёлтым цветом отмечен атом N7 во всех гуанинах.
Рисунок получен с помощью программы Jmol. Скрипт можно скачать по следующей ссылке.
Далее было предложено рассмотреть файлы 1H4S (РНК и связанный с ней белок) и 1MHD (ДНК и белок) из структурного банка PDB. Для того, чтобы проверить заданные структуры ДНК и РНК на разрывы, сначала были получены изображения только нуклеиновой кислоты в проволочной модели (скрипт для 1H4S, скрипт для 1MHD). Взглянув на Рисунок 2 и 3, можно сделать вывод, что разрывов нет, структура цельная и всё, в общем, хорошо. Отдельно были созданы файлы с координатами ДНК и РНК.
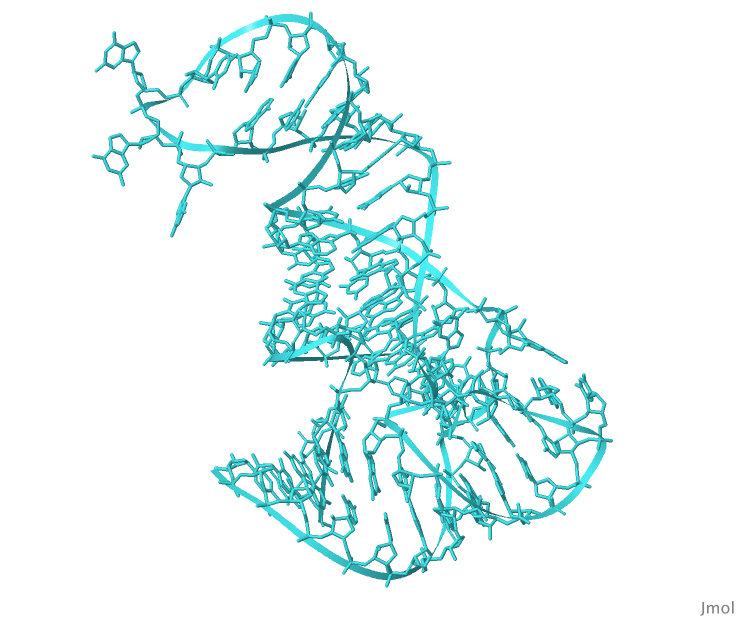
Рисунок 2. Графическое изображение структуры РНК из 1H4S.
Сахарофосфатный остов изображён в проволочной модели.
Рисунок получен с помощью программы Jmol.
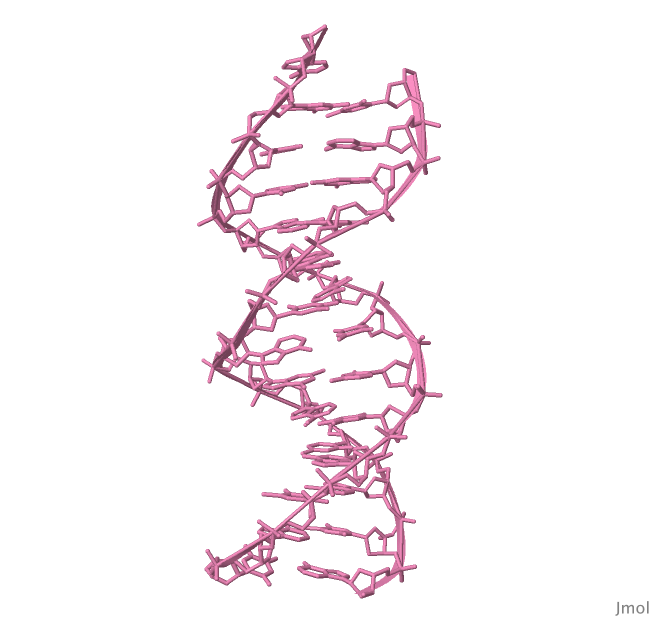
Рисунок 3. Графическое изображение структуры ДНК из 1MHD.
Сахарофосфатный остов изображён в проволочной модели.
Рисунок получен с помощью программы Jmol.
Сравнение структур трёх форм ДНК
Во вторичной структуре ДНК можно визуально выделить большую и малую бороздки, которые, исходя из своего названия, различаются по ширине. На Рисунке 4 приведено изображение участка молекулы ДНК В-формы. По рисунку можно явно определить, где большая, а где малая бороздка.
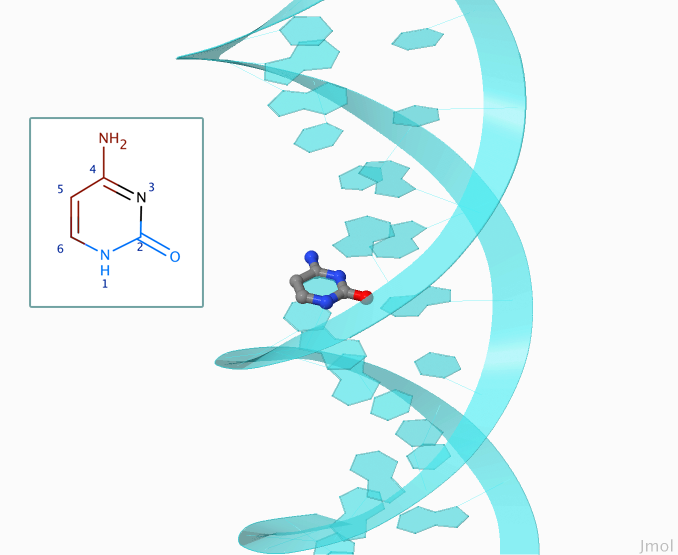
Рисунок 4. Расположение цитозина 14 в ДНК В-формы.
Красным цветом отмечены атомы, обращённые в сторону большой бороздки, синим — малой. Чёрным выделены остальные атомы.
Изображения получены с помощью программы Jmol и MarvinSketch.
Рассмотрим ориентацию атомов цитозина 14 в различных формах ДНК.
- Для цитозина 14 в B-форме ДНК:
- В сторону большой бороздки обращены атомы: с16.n4, с16.с4, c14.c5, c14.c6
- В сторону малой бороздки обращены атомы:c14.o2, c14.n1, c14.c2
- Остальные атомы основания: c14.n3
- Для A-формы ДНК:
- В сторону большой бороздки обращены атомы: с16.o2, с16.n1, c14.n3, c14.c2
- В сторону малой бороздки обращены атомы: c14.n4, c14.c4, c14.c5
- Остальные атомы основания: c14.c6
- Для Z-формы ДНК:
- В сторону большой бороздки обращены атомы: с16.n4, с16.с4, c14.c5
- В сторону малой бороздки обращены атомы: c14.o2, c14.n1, c14.c2
- Остальные атомы основания: c14.n3, c14.c6
Далее было предложено сравнить основные спиральные параметры разных форм ДНК. Изображение A-, B- и Z-формы ДНК с измерениями представлено на Рисунке 5. В таблице 1 приведены полученные параметры.
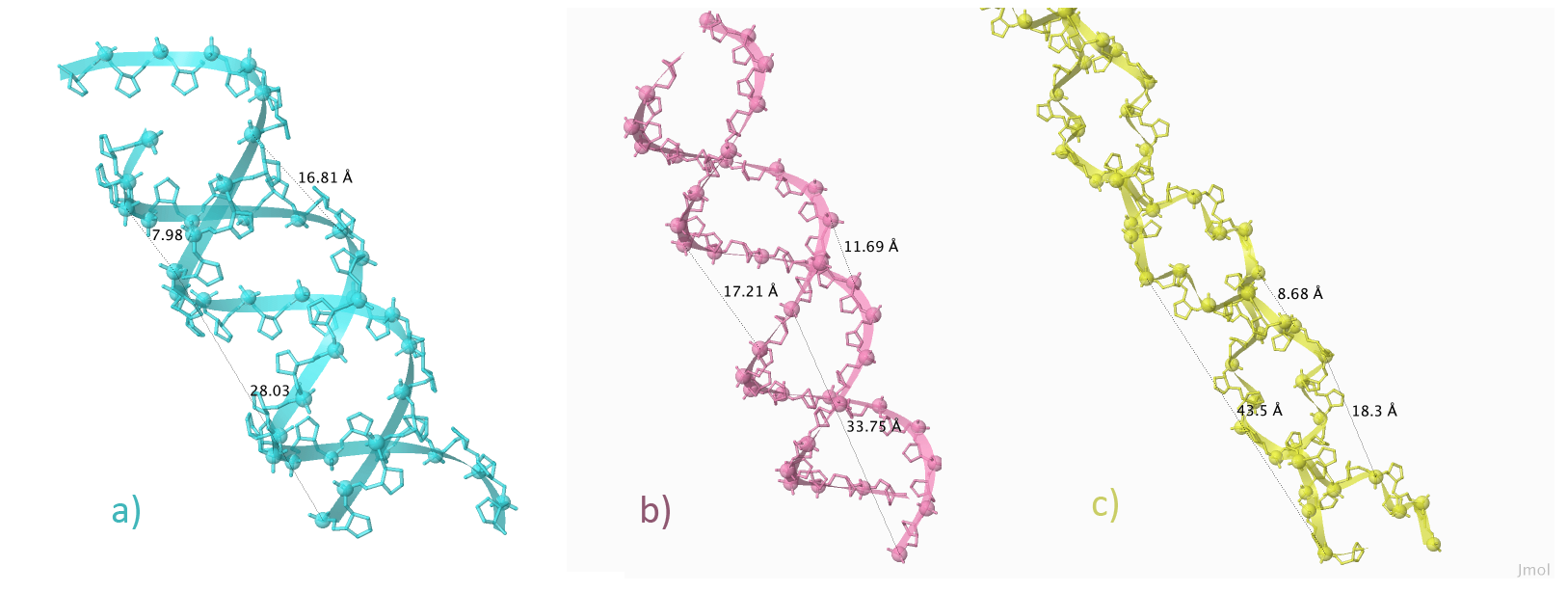
Рисунок 5. A-, B- и Z-форма ДНК.
Под буквой а) представлена A-форма, b) — B-форма, c) — Z-форма. Сахарофосфатный остов изображён в шарнирной модели. Пунктиром отмечены расстояния.
Изображения получены с помощью программы Jmol.
| A-форма | В-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16.81 (T25:B-C8:A) | 17.21 (C10:A-C30:B) | 18.3 (C34:B-C24:A) |
| Ширина малой бороздки | 7.98 (T3:A-C32:B) | 11.69 (G33:B-C12:A) | 8.68 (G23:A-G43:B) |
Торсионные углы
С помощью команды Jmol Settings->Torsion были измерены торсионные углы цитозина 14 в A и В-формах ДНК. Также были использованы данные о торсионных углах из презентации.
В таблице 2 представлены полученные результаты.
| Форма | α | β | γ | δ | ε | ζ | χ |
| А-форма (измеренные) | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| А-форма (презентация) | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| В-форма (измеренные) | -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98 |
| В-форма (презентация) | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
Из таблицы видно, что данные, посчитанные вручную, и данные, взятые из презентации, сильно различаются. Вероятно, это связано с тем, что значения углов из презентации — средние по всем основаниям, а наши посчитаны только для цитозина.
Посчитать торсионные углы можно также с помощью программ из пакета 3DNA. Этот пакет работает только со старым форматом PDB, поэтому с помощью программы remediator переведём файлы с нашими ДНК и РНК в старый формат (1H4S и 1MHD в старом формате). Далее используется конвейр из команд find_pair -t и analyze. Из всех полученных файлов нам нужны те, у которых формат .out. Результаты представлены в Таблице 3.
| Форма | α | β | γ | δ | ε | ζ | χ |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.1 | 143.3 | -140.8 | -160.5 | --97.9 |
| Z-форма (цитозин) | -139.5 | -136.7 | 31.1 | 143.4 | -140.8 | -160.5 | -98 |
| Z-форма (гуанин) | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
Мы видим, что очень сильно отличаются торсионные углы цитозина и гуанина в ДНК Z-формы.
То же самое было сделано определения торсионных углов в заданных ДНК и РНК. Была составлена таблица Excel, где цветом были выделены самые отклоняющиеся (при подсчёте были исключены крайние углы). Это G17 и U15 для РНК и A7 и A6 для ДНК.
Структура водородных связей
Из тех же файлов в формате .out можно получить информацию о вторичной структуре РНК. Она представлена ниже. Названия, обозначения и типы стеблей у тРНК представлены на Рисунке 6.
Strand I Strand II Helix
1 (0.008) T:...4_:[..G]G-----C[..C]:..69_:T (0.008) |
2 (0.009) T:...5_:[..G]G-----C[..C]:..68_:T (0.006) |
3 (0.012) T:...6_:[..A]A-----U[..U]:..67_:T (0.003) |
4 (0.005) T:...7_:[..G]Gx----C[..C]:..66_:T (0.010) |
5 (0.005) T:..49_:[..G]G-*---U[..U]:..65_:T (0.010) |
6 (0.004) T:..50_:[..C]C-----G[..G]:..64_:T (0.013) |
7 (0.002) T:..51_:[..U]U-----A[..A]:..63_:T (0.004) |
8 (0.006) T:..52_:[..G]G-----C[..C]:..62_:T (0.002) |
9 (0.005) T:..53_:[..G]G----xC[..C]:..61_:T (0.005) |
10 (0.012) T:..54_:[5MU]u-**-xG[..G]:..58_:T (0.006) |
11 (0.026) T:..55_:[PSU]Px**+xG[..G]:..18_:T (0.012) x
12 (0.011) T:..38_:[..A]A-----U[..U]:..32_:T (0.009) |
13 (0.003) T:..39_:[..C]C-----G[..G]:..31_:T (0.006) |
14 (0.006) T:..40_:[..G]G-----C[..C]:..30_:T (0.007) |
15 (0.008) T:..41_:[..A]A-----U[..U]:..29_:T (0.009) |
16 (0.005) T:..42_:[..G]G-----C[..C]:..28_:T (0.005) |
17 (0.008) T:..43_:[..G]G-----C[..C]:..27_:T (0.007) |
18 (0.007) T:..44_:[..G]Gx*---A[..A]:..26_:T (0.008) |
19 (0.004) T:..10_:[..G]G-----C[..C]:..25_:T (0.006) |
20 (0.003) T:..11_:[..C]C-----G[..G]:..24_:T (0.004) |
21 (0.004) T:..12_:[..G]G-----C[..C]:..23_:T (0.003) |
22 (0.004) T:..13_:[..C]C----xG[..G]:..22_:T (0.005) |
23 (0.003) T:..14_:[..A]A-**-xU[..U]:...8_:T (0.004) |
24 (0.004) T:..15_:[..G]Gx**+xC[..C]:..48_:T (0.007) x
25 (0.011) T:..19_:[..G]G-----C[..C]:..56_:T (0.005) +
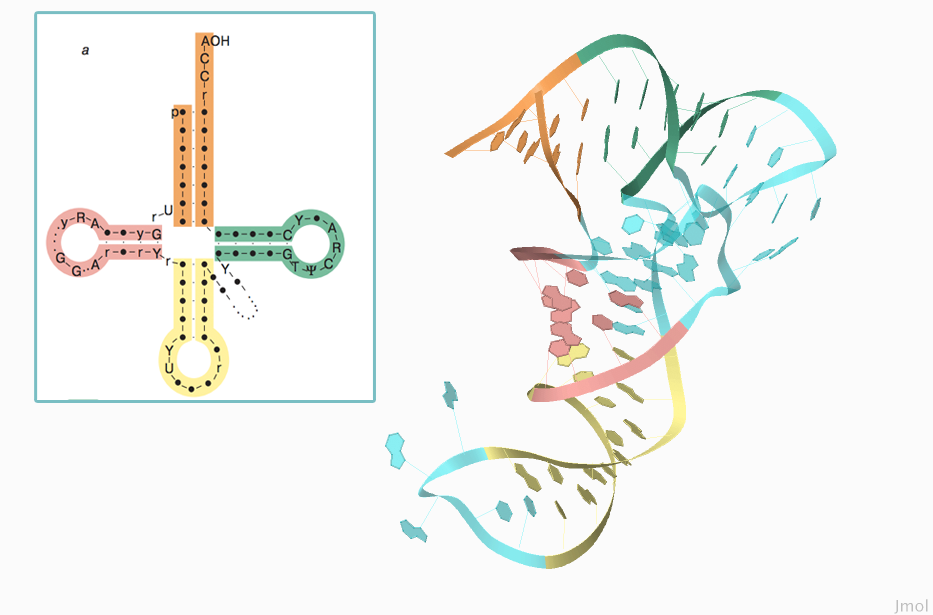
Рисунок 6. Стебли РНК.
Оранжевым цветом обозначен акцепторный стебель, зелёным — T-стебель, жёлтым — антикодоновый, розовым — D-стебель. Всё остальное, что не вошло в стебли, окрашено голубым цветом.
Изображения получены с помощью программы Jmol и MarvinSketch.
Неканонические пары — пары 7 и 66, 49 и 65, 53 и 61, 54 и 58, 55 и 18, 13 и 22, 14 и 8, 14 и 48. Дополнительные водородные связи — 54 и 58, 55 и 18, 44 и 26, 14 и 8, 15 и 48, 19 и 56. Цифры — номера нуклеотидов.
Стекинг-взаимодействия
Из файла в формате .out можно также почерпнуть информацию о стекинг-взаимодействии. Ниже представлена информация о стекинг-взаимодействиях для тРНК 1H4S. Стекинг-взаимодействиях с наибольшей площадью перекрывания окрашены красным, с меньшей — синим:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GG/CC 3.88( 2.64) 0.00( 0.00) 0.05( 0.00) 0.57( 0.02) 4.50( 2.66) 2 GA/UC 5.04( 3.03) 0.00( 0.00) 0.00( 0.00) 0.08( 0.00) 5.12( 3.03) 3 AG/CU 1.42( 1.41) 0.00( 0.00) 1.21( 0.00) 0.00( 0.00) 2.63( 1.41) 4 GG/UC 1.59( 0.26) 0.00( 0.00) 1.10( 0.00) 0.00( 0.00) 2.69( 0.26) 5 GC/GU 6.56( 3.49) 0.00( 0.00) 0.00( 0.00) 6.55( 3.64) 13.11( 7.12) 6 CU/AG 0.00( 0.00) 0.00( 0.00) 0.38( 0.00) 3.82( 3.26) 4.20( 3.26) 7 UG/CA 0.00( 0.00) 0.00( 0.00) 4.80( 3.05) 0.00( 0.00) 4.80( 3.05) 8 GG/CC 5.09( 2.72) 0.00( 0.00) 0.74( 0.00) 0.00( 0.00) 5.83( 2.72) 9 Gu/GC 7.60( 2.39) 0.00( 0.00) 0.00( 0.00) 5.65( 2.63) 13.24( 5.01) 10 uP/GG 7.22( 2.93) 0.00( 0.00) 0.00( 0.00) 6.02( 3.11) 13.24( 6.04) 11 PA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 12 AC/GU 3.07( 1.61) 0.00( 0.00) 0.00( 0.00) 7.18( 4.20) 10.25( 5.81) 13 CG/CG 0.52( 0.00) 0.00( 0.00) 2.89( 0.58) 1.19( 0.19) 4.59( 0.77) 14 GA/UC 4.54( 3.06) 0.00( 0.00) 0.00( 0.00) 1.33( 0.11) 5.87( 3.17) 15 AG/CU 2.91( 2.55) 0.00( 0.00) 0.58( 0.00) 0.00( 0.00) 3.50( 2.55) 16 GG/CC 3.57( 1.78) 0.00( 0.00) 1.11( 0.00) 0.00( 0.00) 4.68( 1.78) 17 GG/AC 1.05( 0.00) 0.00( 0.00) 0.00( 0.00) 2.78( 1.53) 3.82( 1.53) 18 GG/CA 0.00( 0.00) 0.00( 0.00) 0.05( 0.00) 0.75( 0.24) 0.80( 0.24) 19 GC/GC 4.70( 1.85) 0.00( 0.00) 0.00( 0.00) 7.21( 4.46) 11.91( 6.31) 20 CG/CG 0.38( 0.00) 0.00( 0.00) 4.44( 1.72) 0.00( 0.00) 4.83( 1.72) 21 GC/GC 6.58( 2.45) 0.00( 0.00) 0.00( 0.00) 3.40( 0.98) 9.98( 3.44) 22 CA/UG 0.00( 0.00) 1.35( 0.00) 4.78( 1.78) 0.00( 0.00) 6.13( 1.78) 23 AG/CU 3.84( 1.22) 0.00( 0.00) 0.02( 0.00) 0.00( 0.00) 3.86( 1.22) 24 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
На Рисунках 6-7 представлены графические изображения выделенных стекинг-взаимодействий, полученные с помощью команд (* номер взаимодействия):
ex_str -* stacking.pdb step*.pdb stack2img -cdolt step*.pdb step*.ps
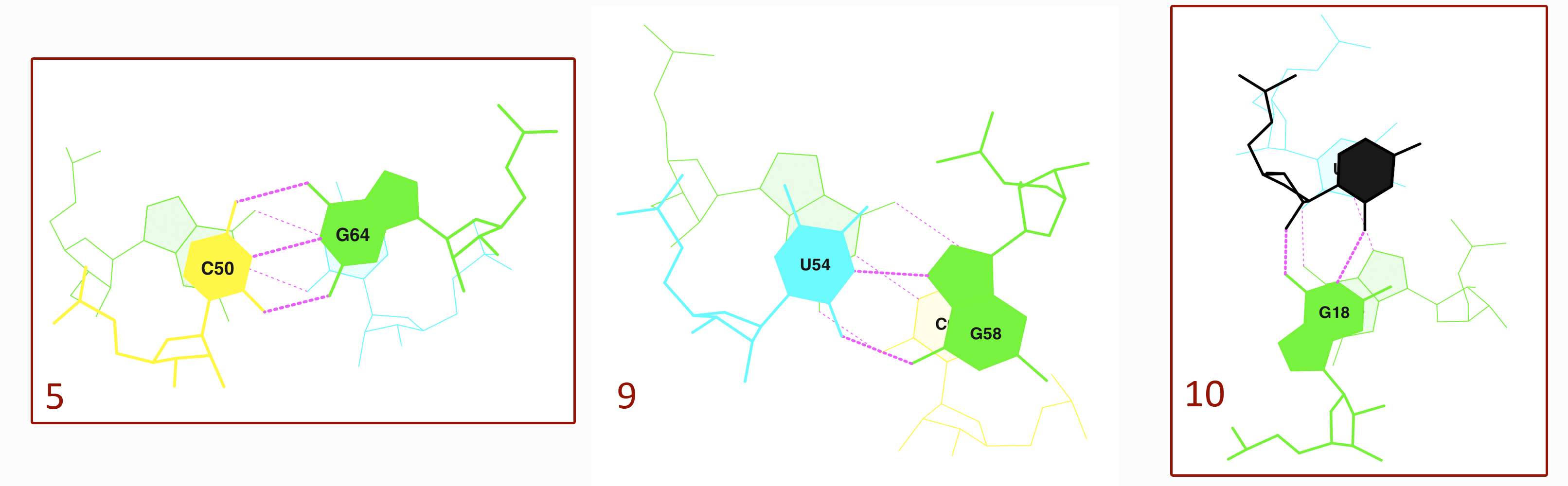
Рисунок 6. Графическое изображение структуры ДНК из 1MHD.
Сахарофосфатный остов изображён в проволочной модели.
Изображения получены с помощью программы Jmol и MarvinSketch.
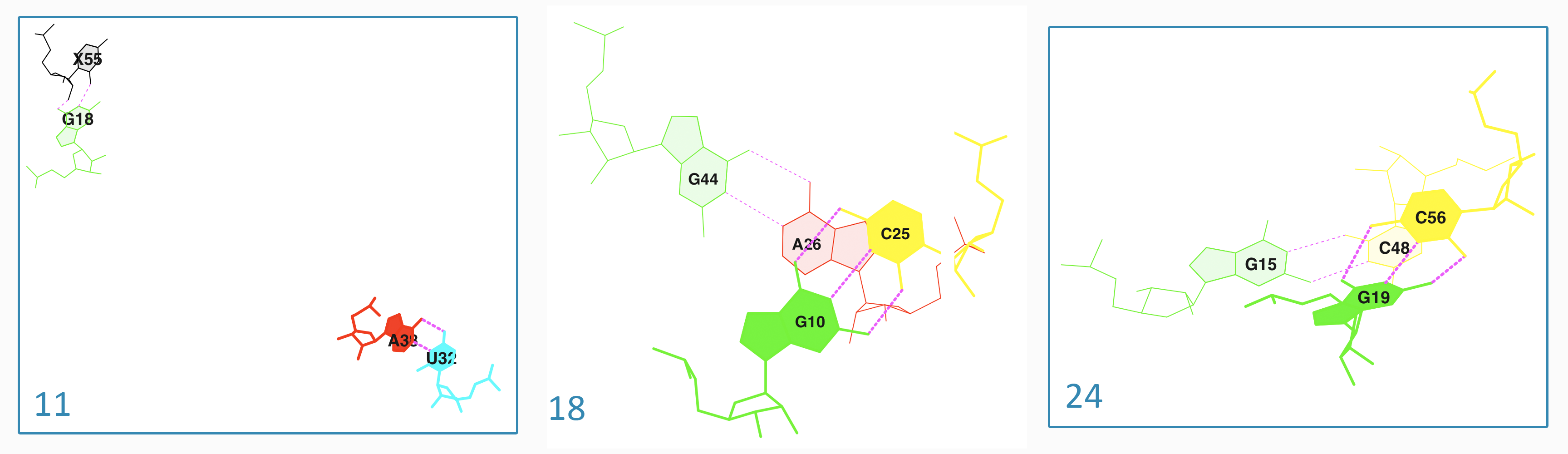
Рисунок 7. Графическое изображение структуры ДНК из 1MHD.
Сахарофосфатный остов изображён в проволочной модели.
Изображения получены с помощью программы Jmol и MarvinSketch.