Комплексы ДНК-белок
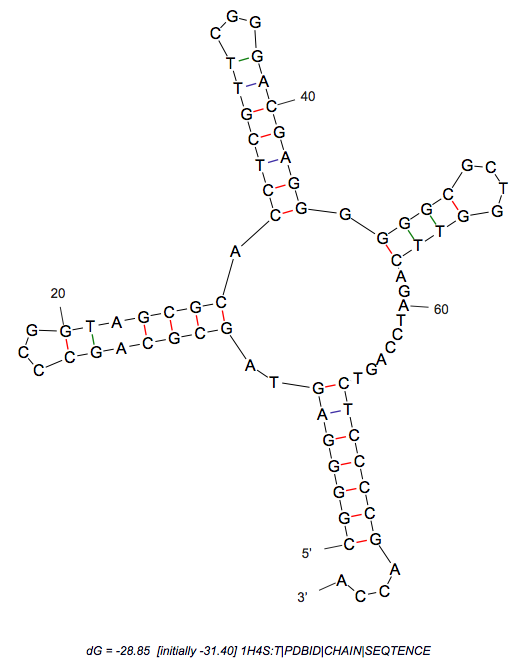
Вторичная структура тРНК
Программа запускалась несколько раз с различными параметрами. При стандартных (gap penalty 12, minimum score threshold 50, match score 3, mismatch score -4) программа выдаёт пустой файл. То же самое происходит в любом случае, когда второй параметр равен 50. При mismatch score меньше -2, программа выдаёт только один стебель:
SEQTENCE: Score 21: 7/7 (100%) matches, 0 gaps
1 cggggag 7
|||||||
73 gcccctc 67
В дальнейшем я использовала результ полученный при gap penalty 12, minimum score threshold 30, match score 3, mismatch score -2:
SEQTENCE: Score 27: 19/34 ( 55%) matches, 0 gaps
2 ggggagtagcgcagcccggtagcgcacctcgttc 35
|| || | | | | | || | |||||| |
70 cctctgacctagacttggtcgcggggggagcagg 37
Посмотрев на результат, замечаем, что он довольно-таки сильно отличается от того, что мы видели после применения find_pair в предыдущем практикуме.
Вторичную структуру РНК можно также предсказать с помощью алгоритма Зукера. Для этого можно использовать программу mfold из пакет EMBOSS. Я использовала web-вариант. Результат представлен на Рисунке 1.
Рисунок 1. Предсказанная вторичная структура тРНК по алгоритму Зукера.
В таблице 1 представлен анализ полученных результатов.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-4-7-3' 5'-66-69-3' всего 4 пары |
предсказано 0 пар из реальных | предсказано 0 пар (смещение на 1 основание) |
| D-стебель | 5'-10-13-3' 5'-22-25-3' всего 4 пары |
предсказано 0 пар из реальных | предсказано 0 пар (смещение на 1 основание) |
| T-стебель | 5'-49-53-3' 5'-61-65-3' всего 5 пар |
предсказано 0 пар из реальных | предсказано 0 пар |
| Антикодоновый стебель | 5'-38-44-3' 5'-26-32-3' всего 7 пар |
предсказано 0 пар из реальных | предсказано 7 пар |
| Общее число канонических пар нуклеотидов | 23 | 19 | 21 |
Комплексы ДНК-белок
Для начала был создан скрипт, который определите множество атомов кислорода 2'-дезоксирибозы (RibO), множество атомов кислорода в остатке фосфорной кислоты (PhosphO), множество атомов азота в азотистых основаниях (NucN), а также даёт последовательное изображение всей структуры (только ДНК в проволочной модели, ДНК той же модели, но с выделенными шариками множеством атомов RibO, затем PhosphO и NucN). Скрипт можно скачать отсюда. PDB-файл с ДНК отсюда.
Теперь опишем ДНК-белковые контакты в структуре 1MHD. Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5 А. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии 4.5 А. Данные о контактах представлены в таблице 2. Заметим, что меньше всего контактов со стороны малой бороздки.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 9 | 10 |
| остатками фосфорной кислоты | 8 | 0 | 10 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 8 | 10 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 3 | 3 |
Оценим ДНК-белковые контакты с помощью программы nucplot на kodomo. Использованная команда: nucplot 1MHD_old.pdb. Скачать файл в старом формате можно по ссылке
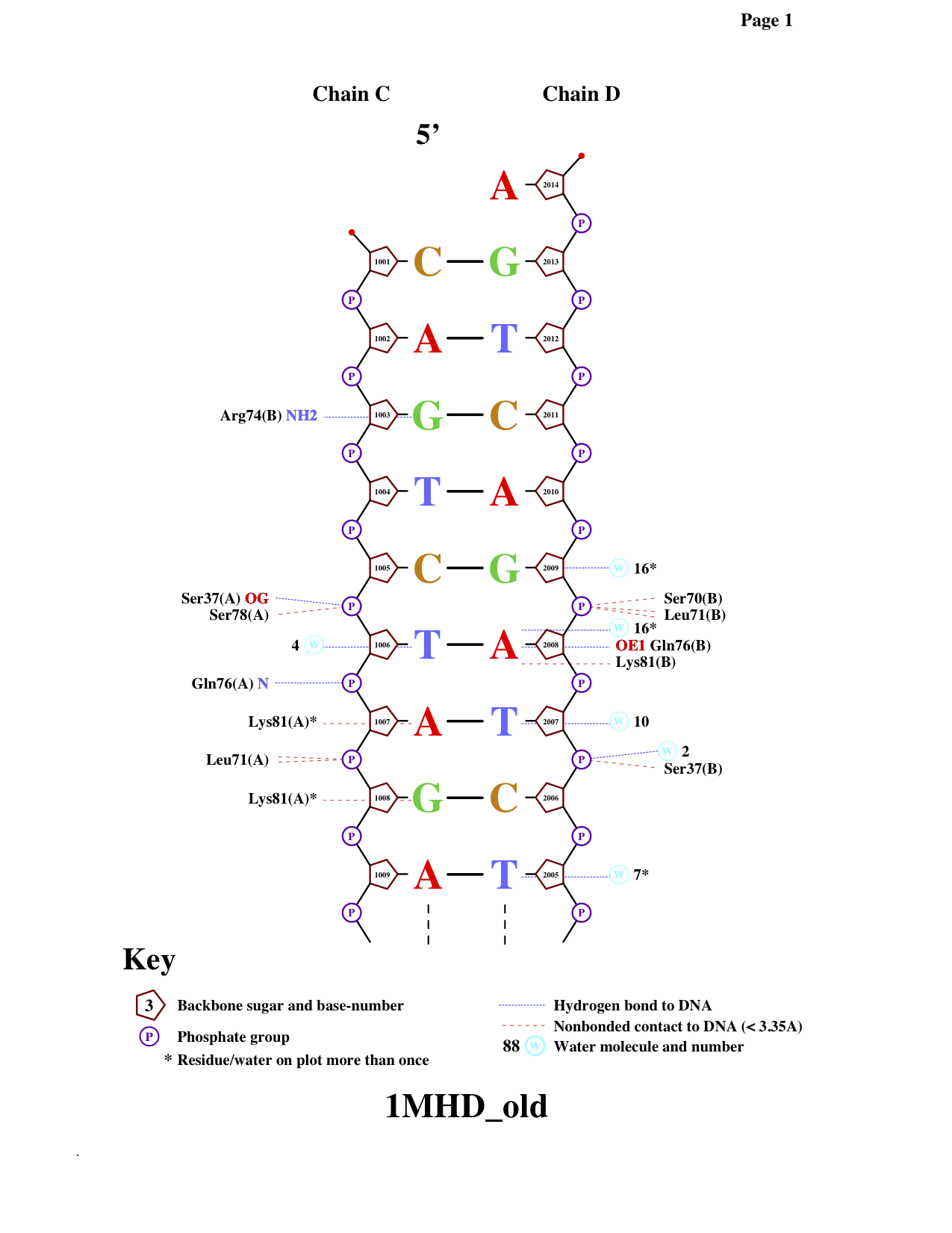
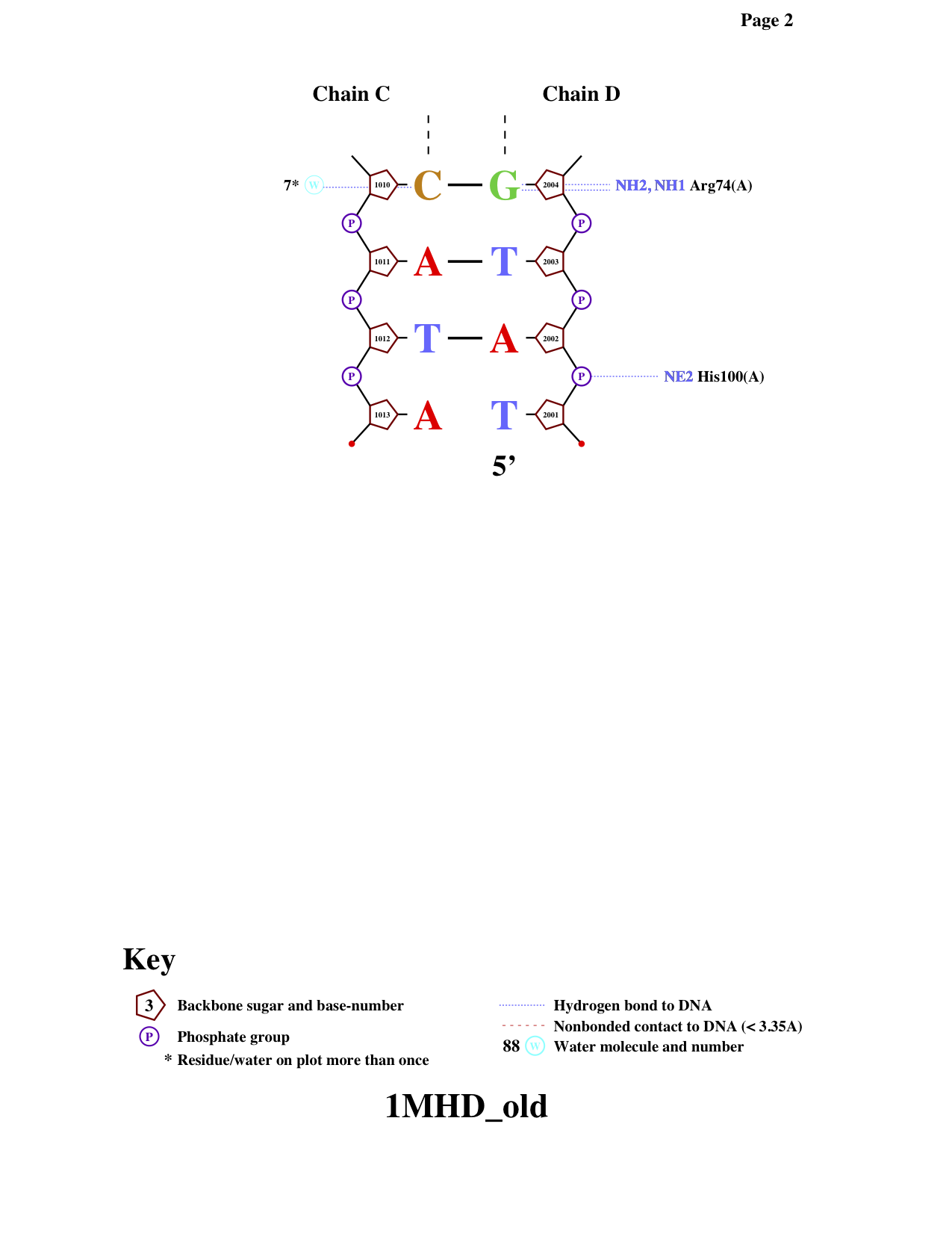
Рисунок 2. Результат работы программы nucplot.
- Дальше рассмотрим:
- аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК: Arg74
- аминокислотный остаток наиболее важный для распознавания последовательности ДНК: Gln76 (важен, т.к. связан не с сахарофосфатным остовом, а с самим азотистым основанием (см. Рисунок 2))
Ниже представлены изображения соответствующих аминокислотных остатком, сделанных с помощью JMol (Рисунок 3, 4).
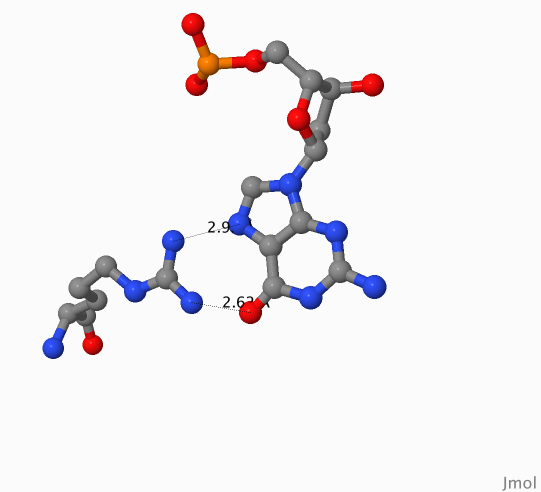
Рисунок 3. Контакты Arg74 и G2004
Изображения получены с помощью программы Jmol.
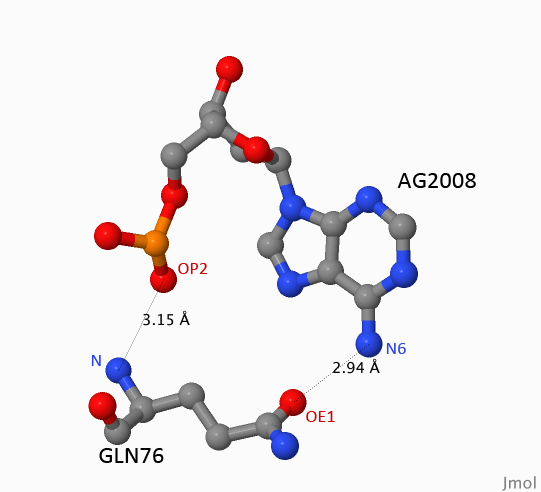
Рисунок 4. Контакты Gln76 с A2008.
Изображения получены с помощью программы Jmol.