Поиск сигналов. Последовательность Шайн-Дальгарно
Поиск последовательности Шайн-Дальгарно осуществлялся в геноме бактерии Chloroflexus aurantiacus. В качестве участков поиска выступали участки [-35,0] относительно начала трансляции.
Для этого сначала был получен полный геном бактерии, а также хромосомная таблица. С помощью скрипта были получены координаты участков поиска. Всего было получено 3944 последовательностей.
Для создания PWM использовалась программа MEME. На вход были подана выборка из 100 последовательностей (выбирались такие, у кого в графе был описан продукт, а не предполагаемый белок). Использовалась команда ememe col.fasta -mod zoops -minw 6 -maxw 7
В результате было найдено 25 семинуклеотидных сайтов, получено лого сигнала (рисунок 1), а также html-файл о проделанной работе.
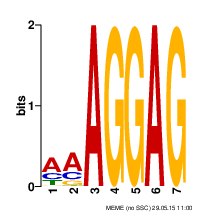
Рисунок 1. LOGO сигнала предполагаемой последовательность Шайн-Дальгарно.
Изображение получено с помощью программы MEME.
Далее, основываясь на результатах MEME, была запущена программа MAST уже по всем 3944 последовательностям (команда: emast meme.txt -dfile find2.fasta mast.html). В итоге было найдено 85 сигналов, чей e-value равнялся 15. Здесь можно посмотреть html-выдачу и следовательно номера тех последовательностей, перед которыми по идее находится наш сигнал.