Восстановление предкового состояния доменной архитектуры
Выбор объекта изучения
В качестве объекта изучения было выбрано семейство доменов MaoC_dehydrat_N (PFAM ID PF13452). Этот домен появился в результате расхождения в высших эукариот N- и C-мономеров дегидратазы MaoC. В то же время возник и второй домен — MaoC_dehydratas, который рассматривался в одном из предыдущих практикумов. Известно, что этот домен связан с дегидратазной активностью.
MaoC_dehydrat_N входит в состав 31 архитектуры. На рисунке 1 представлены архитектуры, с которыми будет осуществляться последующая работа. Первая из них — однодоменная, представлена 1182 последовательности. Вторая включает в себя ещё домен MaoC_dehydratas, и её содержит 818 последовательностей.
Выравнивание выбранного семейства доменов можно загрузить в формате JalView и fasta.

Рисунок 1. Выбранные архитектуры с доменом MaoC_dehydrat_N.
Рисунок получен с сайта Pfam.
Далее с помощью скрипта swisspfam_to_xls.py была получена таблица Excel с информацией об архитектуре всех последовательностей с доменом MaoC_dehydrat_N. Полученные данные находятся на листе с названием "domain list". Далее была составлена сводная таблица (лист "domain architecture"), в которой показана встречаемость каждого домена в последовательности. Цветом выделены колонки, соответствующие доменам из двух выбранных архитектур. Также отдельно с помощью ранее полученных данных была посчитана длина доменов.
Следующий скрипт (uniprot-to-taxonomy.py) позволил получить данные о таксономии последовательностей. Их можно найти на листе "domain architecture" и отдельно на странице "taxonomy".
В качестве основного таксона были выбраны все "cellular organisms", а подтаксонов — царства Bacteria и Eukaryota.
Построение филогенетического дерева последовательностей домена
Для построения дерева было отобрано 46 последовательностей (на каждую архитектуру по 23) длины от 126 нуклеотидов до 136. Отобранные представители архитектур отмечены на листе "domain architecture" в отдельной колонке знаком "+".
Далее с помощью скрипта filter-alignment.py были все выбранные последовательности получены и потом обработаны в JalView (были удалены те, которые хуже всего выравнивались, а также плохо выровненные N- и C-концевые участки). Проект JalView можно загрузить здесь.
На основе полученного выравнивания было построено дерево с помощью метода Neighbour-Joining (файл в формате .nwk). Полученный результат можно увидеть на рисунке 2. Выбранные токсоны и доменные архитектуры закодированы следующим образом: E — эукариоты, B — бактерии, 1 — выбранная однодоменная архитектура, 2 — двудоменная.
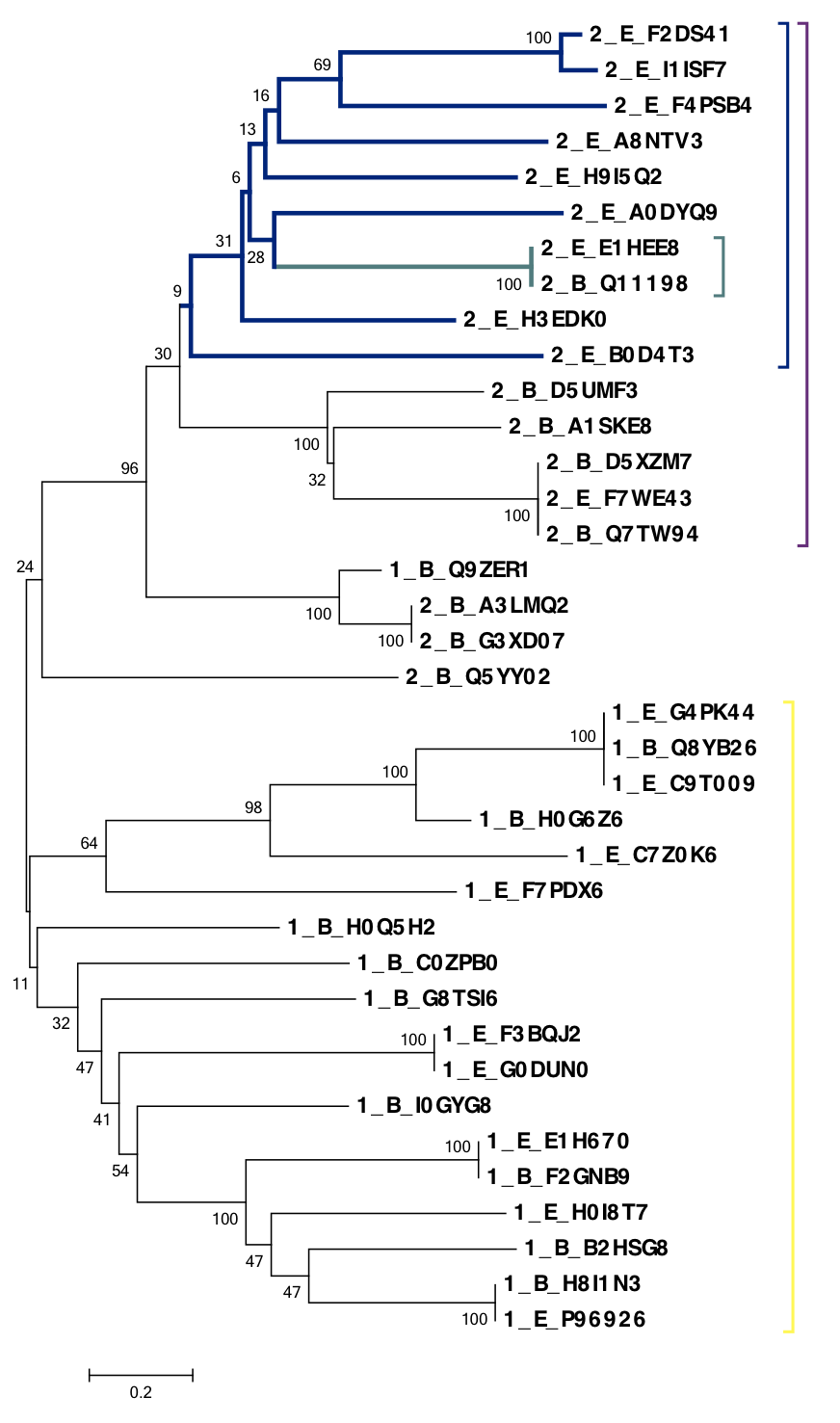
Рисунок 2. Дерево, построенное по последовательностям с выбранными доменными архитектурами.
Рисунок получен с помощью программы Mega5.1. Дерево построено по алгоритму Neighbour-Joining. Также был проведён бутстреп-анализ.
По полученному дереву видно, что последовательности разбиваются на 2 клады, которые почти соответствуют доменной архитектуре (фиолетовая и жёлтая скобки). Также видно, что отдельно выделяются почти все двудоменные эукариотические последовательности (синяя ветвь и скобочка).
Можно предположить, что на каком-то этапе исходная архитектура разделилась на двудоменную и однодоменную ( на каком этапе не понятно). Двудоменная появилась у бактерий, а потом распространилась у эукариот.
Построение профиля подсемейства
В качестве подсемейства последовательностей были выбраны эукариотические с двудоменной архитектурой, так они все входят в одну кладу (см. рисунок 2, фиолетовая скобка). Выравнивание этих последовательностей, полученное из общего выравнивания, можно загрузить отсюда. Для построения профиля по выбранному подсемейству использовалась программы пакета HMMER. Сначала профиль был построен программой hmm2build, потом откалиброван hmm2calibrate (результат). С помощью профиля был осуществлён поиск по всем белкам Uniprot по всем белкам с доменом MaoC_dehydrat_N (fasta-файл). Полученную информацию можно увидеть в txt-файле, а также в упоминавшемся ранее Excel-файле на листе "HMM". На рисунке 3 представлена roc-кривая, построенная с помощью полученных данных.
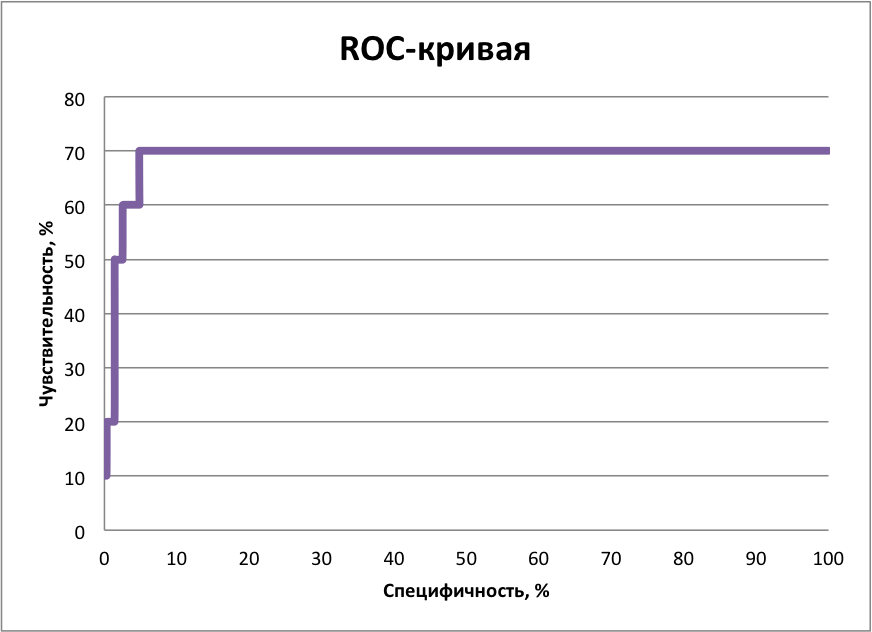
Рисунок 3. Roc-кривая.
Рисунок получен с помощью программы Excel.
Всего было найдено 1388 последовательностей, 1275 из них удовлетворяли порогу на e-value (<10):от 1,5x10-74 до 9,9. При этом нашлось лишь 7 из 10 последовательностей выбранного подсемейства, что отразилось на чувствительности (она не больше 70%). В Excel-файле эти последовательности выделены зелёным цветом.
При пороге e-value 10-7 чувствительность составляет 70%, а специфичность — 43,3%. Остальные данные представлены в таблице 1.
| На самом деле | принадлежит семейству | не принадлежит | сумма |
| Выше порога по профилю | 7 | 549 | 556 |
| Ниже порога | 0 | 720 | 720 |
| сумма | 7 | 1269 | 1276 |
Таким образом, можно сказать, что профиль не пригоден для выделения данного подсемейства, так как примерно 1/3 исходных последовательностей была не найдена. Также можно сделать вывод и о неудачном выборе самого подсемейства.