» Семестры » Восьмой семестр » Докинг низкомолекулярных лигандов в структуру белка
Докинг низкомолекулярных лигандов¶
Цель данного занятия является знакомство с возможностями докинга низкомолекулярного лиганда в структуру белка. В работе использовался пакет Autodock Vina и oddt. Объектом исследования был белок лизоцим, структура которого была построена на прошлом практикуме.
Для начала импортируем необходимые для работы модули:
import numpy as np
import copy
# Отображение структур
import IPython.display
import ipywidgets
from IPython.display import display,display_svg,SVG,Image
# Open Drug Discovery Toolkit
import oddt
import oddt.docking
import oddt.interactions
# Органика
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
import pmx # Модуль для манипулирования pdb
Посмотрим, где в нашей структуре лизоцима находятся лиганды:
pdb=pmx.Model('hyace_lig.B99990001.pdb')
for r in pdb.residues[139:]:
print r #посмотрим остатки
Удалим все лиганды (NAG, NAG и NDG):
newpdb = pdb.copy()
for r in newpdb.residues[-3:]:
print r
newpdb.remove_residue(r)
lig = pdb.copy()
del lig.residues[:-3]
newpdb.writePDB(fname="hyace_prot_only.pdb")
lig.writePDB(fname="hyace_ligs_only.pdb")
Найдём айдите геометрический центр лиганда (место для докинга):
a_lig_x = [a.x[0] for a in lig.atoms]
a_lig_y = [a.x[1] for a in lig.atoms]
a_lig_z = [a.x[2] for a in lig.atoms]
x_average = sum(a_lig_x)/len(a_lig_x)
y_average = sum(a_lig_y)/len(a_lig_y)
z_average = sum(a_lig_z)/len(a_lig_z)
Подготовим белок для докинга:
prot = oddt.toolkit.readfile('pdb','hyace_prot_only.pdb').next()
prot.OBMol.AddPolarHydrogens()
prot.OBMol.AutomaticPartialCharge()
print 'is it the first mol in 1lmp is protein?',prot.protein,':) and MW of this mol is:', prot.molwt
Хотя в выдаче написано, что структура не является белком, это не так. В общем, работаем с тем, что имеем.
Теперь подготовим лиганды для докинга. NAG содержит в себе СH3C(=O)NH группу.
Создадим лиганды, где метильный радикал этой группы будет заменён на:
- OH
- NH3+
- H
- Ph
- COO-
smiles = ["CC(=O)NC1C(C(C(OC1O)CO)O)O","OC(=O)NC1C(C(C(OC1O)CO)O)O","[NH3+]C(=O)NC1C(C(C(OC1O)CO)O)O","C(=O)NC1C(C(C(OC1O)CO)O)O","C1=CC=CC=C1C(=O)NC1C(C(C(OC1O)CO)O)O","[O-]C(=O)C(=O)NC1C(C(C(OC1O)CO)O)O"]
mols= []
images =[]
for s in smiles:
m = oddt.toolkit.readstring('smi', s)
if not m.OBMol.Has3D():
m.make3D(forcefield='mmff94', steps=150)
m.removeh()
m.OBMol.AddPolarHydrogens()
mols.append(m)
###with print m.OBMol.Has3D() was found that:
### deep copy needed to keep 3D , write svg make mols flat
images.append((SVG(copy.deepcopy(m).write('svg'))))
display_svg(*images)
Докинг¶
Теперь займёмся непосредственно докингом:
#create docking object
dock_obj= oddt.docking.AutodockVina.autodock_vina(
protein=prot,size=(20,20,20),center=[x_average,y_average,z_average],
executable='/usr/bin/vina',autocleanup=True, num_modes=20)
print dock_obj.tmp_dir
print " ".join(dock_obj.params)
center_x, center_y, center_z- центр куба, куда будет помещена молекула;size_x, size_y, size_z- размер куба;exhaustiveness- параметр Autodock Vina;num_modes- число конформаций, генерируемых Autodock Vina;energy_range- cut-off по энергии.
# do it
res = dock_obj.dock(mols,prot)
Анализ результатов¶
from operator import itemgetter
res_list = [[r.formula, r.data['vina_affinity'], r.data['vina_rmsd_ub'], r.residues[0].name, i] for i,r in enumerate(res)]
final_res = sorted(res_list, key=itemgetter(1),reverse=True)
for el in final_res:
print "%4d%10s%8s%8s%8s" %(el[-1],el[0],el[1],el[2],el[3])
Наилучшей оказалась структура под номером 28. Попробуем получить её трёхмерную структуру:
for i,r in enumerate(res):
hbs = oddt.interactions.hbonds(prot,r)
stack= oddt.interactions.pi_stacking(prot,r)
phob = oddt.interactions.hydrophobic_contacts(prot,r)
for i,r in enumerate(res):
r.write(filename='r%s.pdb' % i, format='pdb')
Ниже представлено графическое представление результатов докинга. Белым цветом окрашена структура с худшим заместителем (C7H13NO6), малиновым - наилучшим (C13H17NO6, 28). Тонкими синими линиями показано положение исходных лигандов.
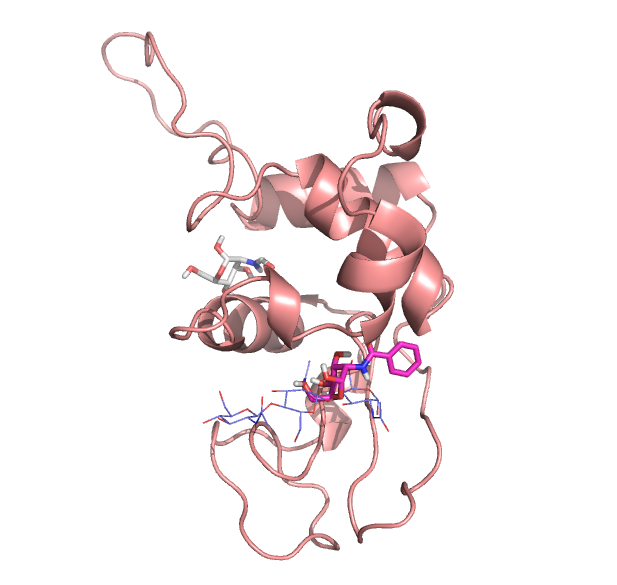
Видно, что хорошая структура находится в том же кармане, что и исходные лиганды.
Дата последнего обновления: 19.05.17
© Светлана Яровенко