Сигналы и мотивы - 2
7.1 Последовательности Козак Danio rerio
Для выполнения был выбран вариант 9.
Таблица с расчётами и результатами.
Суммарное информационное содержание сигнала 10.6, сигнал достаточно сильный.

7.2 Сайты разрывной транскрипции sgmRNA
Результат выполнения пункта 6.2 (а именно короткий вариант AACGAA из шести позиций) был передан на вход программе FIMO вместе с геномами трёх бета-коронавирусов вирусов: человеческих EMC/2012 (того же, что в прошлом практикуме), Jordan-N3/2012 (другого штамма того же вида) и летучей мыши BetaCoV/SC2013 (близкого вида).
Выдача FIMO для них: HCoV-EMC, HCoV-Jordan-N3, BCoV-SC.
Для удобства находки FIMO были сопоставлены с разметкой генов в feature table (находка добавлялась до гена, если целиком лежит до его начала и после в противном случае, в т.ч. при нахождении внутри гена): HCoV-EMC, HCoV-Jordan-N3, BCoV-SC.
В HCoV-EMC мотив нашёлся перед каждым из генов кроме последнего (orf8b), а также несколько раз внутри генов.
Для HCoV-Jordan-N3 и BCoV-SC находки совершенно аналогичны: перед каждым геном, кроме orf8b и несколько раз внутри.
Стоит заметить, что "расширенный", но менее строгий вариант TTAACGAAC мотива из первой выдачи MEME для HCoV-EMC находился и в upstream области orf8b, однако именно более константная для других генов средняя часть, которая была подана на вход FIMO у него отличается (он выглядит как TTACCCAAC, т.е. ACCCAA вместо AACGAA), поэтому он и отсутствует среди находок FIMO.
Интересно, что между найденным мотивом и началом трансляции гена orf1ab HCoV-EMC находится ещё один ATG (см. фрагмент файла с upstream регионами из шестого практикума, приведённый ниже; находки FIMO и ATG выделены заглавными буквами). Вероятно он не работает как инициаторный из-за несоотвествия окружения последовательности Козак.
>"orf1ab" upstream region gatttaagtgaatagcttggctatctcacttcccctcgttctcttgcagaactttgattt tAACGAActtaaataaaagccctgttgtttagcgtatcgttgcacttgtctggtgggatt gtggcattaatttgcctgctcatctaggcagtggacatATGctcaacactgggtataatt ctaattgaatactatttttcagttagagcgtcgtgtctcttgtacgtctcggtcacaata cacggtttcgtccggtgcgtggcaattcggggcacatc
Проверить эту гипотезу можно, построив консенсус последовательностей Козак данного коронавируса. Для лого были взяты участки -6, 7 (как наиболее информационно нагруженная, судя по лого для человеческих последовательностей) для каждого из генов HCoV-EMC.

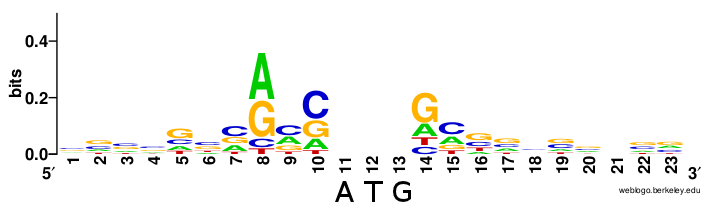
Сходство невысокое, схожа только позиция +5 (на рисунке обозначена как 4 и 15 соответственно из-за особенностей нумерации).
Тем не менее, можно видеть, что последовательность ggacatATGctca из upstream региона гена orf1ab не похожа на последовательности Козак вокруг реальных инициаторных кодонов этого же вируса, гипотеза подтвердилась.
Таким образом, можно сделать вывод, что найденный мотив дейстивтельно является сигналом транскрипции (по крайней мере его частью, CS) и он достаточно специфичен для вида.