MEME и MAST
1. Поиск мотивов программой MEME.
Лист-файл с невыравненными последовательностями белком мы уже делали в одном из предыдущих практикумов, оттуда его и возьмем. Запустим программу ememetext:
ememetext myproteins.fasta myproteins_meme.txt tmp.fasta -nmotifs 3
Итак, было найдено всего три мотива.
| № мотива | Длина | E-value | Во вcех ли последовательностях нашелся | Координаты в RIDA_BACSU | P-value в RIDA_BACSU | Мотив |
| 1 | 21 | 2.4e-047 | + | 99-119 | 2.57e-23 | [FK]PAR[SA][CAT]V[EGQ]V[AK][AR]LPK[NDG][AV][KL][VI]EIE |
| 2 | 23 | 3.4e-038 | + | 2-24 | 3.61e-23 | [MT]K[AIV][IV][HS]T[DK]DAP[AK][AP][IV]GPY[SV]Q[AG][IV][IK]V[DNG] |
| 3 | 29 | 1.9e-041 | + | 67-95 | 2.78e-25 | [DQS][FRV][ES]N[IV][VI]K[TA]T[VI]F[LI][TK]D[LI][EGK][DK]FA[ER][VI]N[EA][IV]Y[EG][KQR][FY]F |
2. Сравнение блоков, найденных MEME, с полным выравниванием, выданным muscle.
Сначала получим выравнивание muscle:
muscle -in myproteins.fasta -out myproteins_muscle.fasta
(результат в файле myproteins_muscle.fasta.)
Результаты выравнивания в JalView - в файле myproteins11.jar и на следующем изображении (все блоки полностью совпадают с выравниваниями и выделены разными цветами):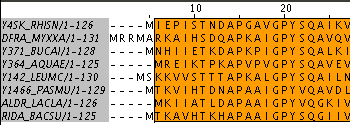
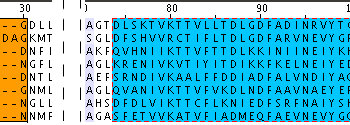

3. Поиск найденных мотивов в других последовательностях.
Файл с последовательностями из предыдущего практикума - vyr.msf.
Сначала удалим гэпы и и переведем файл в fasta-формат:
degapseq vyr.msf vyr.fasta
Затем запустим emast:
emast -dfile vyr.fasta myproteins_meme.txt mastout.html
Результат находится в файле mastout.html.
Второй мотив вообще не попал в выравнивание (просто не вошел в нужный фрагмент, к примеру, в RIDA_BACSU выравнивание начинается с 7го остатка, а мотив - со 2го), поэтому он не был найден ни в одной последовательности. Первый мотив был найден в 23 из 30 последовательностей, третий - в 25. Оба мотива были найдены в 22 последовательностях. Выравнивание соответствует мотивам.