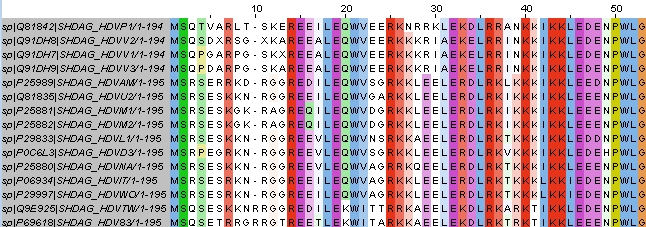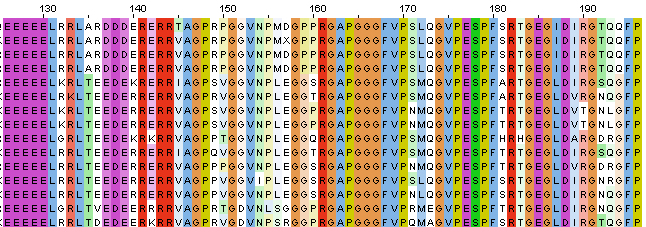Множественные выравнивания
1. Выравнивание набора гомологов своего белка
a) С помощью BLAST я получила список гомологов своего белка (P37552). Так как их количество даже с ограничениями в e-value было слишком большим, пришлось провести отбор. Прежде всего, я оставила только те, где идентичность была > 40% и выкинула чересчур похожие последовательности, так как их выравнивание смотреть неинтересно и во многом бессмысленно. Остались следующие 7 (+1 исходная) последовательностей:
sw:rida_bacsu sw:aldr_lacla sw:y1466_pasmu sw:dfra_myxxa sw:y142_leumc sw:Y4SK_RHISN sw:Y364_AQUAE sw:Y371_BUCAI
Этот список я внесла в файл myproteins.list.
Затем командой seqret @myproteins.list myproteins.fasta я получила, собственно, последовательности указанных белков в fasta-формате (myproteins.fasta).
b) С помощью JalView и программы Probcons я выровняла полученные последовательности. Сохраненное выравнивание можно посмотреть в файлах myproteins.msf и myproteins.jar.
c) По строке "Conservation" в результатах выравнивания можно увидеть, какие участки оставались консервативными во всех найденных последовательностях.
- 14-23 остатки выравнивания (10-19 моего белка) можно целиком рассматривать как достаточно консервативный участок, с наиболее консервативными позициями 14-15, 19-21 и 23. Можно заметить, что чаще всего прочие остатки заменялись на в целом похожие.
- 26-40 остатки выравнивания (30-34 моего белка): участок 26-29 наиболее полно сохранялся, дальше тенденция уже хуже, но остатки изменялимь в 1-2 случаях из семи и на одни и те же остатки.
- 111-113 (100-102) - участок всем хорош, кроме того, что он, наверное, слишком короткий. Но вряд ли это случайность, так что приведу этот участок здесь.
- 127-133 (116-122) - с одной стороны, включает в себя совсем консервативный участок 128-129, прочие остатки заменялись всего в 1-2 случаях и всегда на одни и те же, что показывает консервативность всего участка; с другой стороны, его концы не консервативны.
Белки ALDR_LACLA, Y1466_PASMU и DFRA_MYXXA отличаются от прочих тем, что в них встречаются "вставленные" участки последовательности, которые соответствуют гэпам у всех остальных белков, к примеру:
ALDR_LACLA: 105-108;
Y1466_PASMU: 3-7, 29-30;
DFRA_MYXXA: 105-108.
Это явление соответствует вставочной мутации, так что не противоречит биологическому смыслу.
d) Используя раскраску BLOSUM62 и строку "Consensus", я определила функцонально консервативные группы этих белков. Скобками обозначены остатки, которые в 1-2 случаях заменяются на схожие, так что можно сказать, что они тоже входят в группу:
[AP] [(I)GPY(S)Q(A)] [SGQ(I)] [YF] ~ [FF] [PAR] [LP] [EI(E)]
Ну и, разумеется, консервативен метионин в начале последовательностей.
Трудно выделить наиболее часто встречающуюся группу, но можно заметить, что сочетание PA (AP) встречается часто, на соответствуюзих позициях и во всех белках.
С помощью JalView я построила филогенетическое дерево гомологичных моему белку последовательностей. Как мы видим, наиболее близкий к моему белку - ALDR_LACLA (тот самый со вставочной мутацией), самый дальний от всех прочих последовательностей - Y142_LEUMC (кликабельно).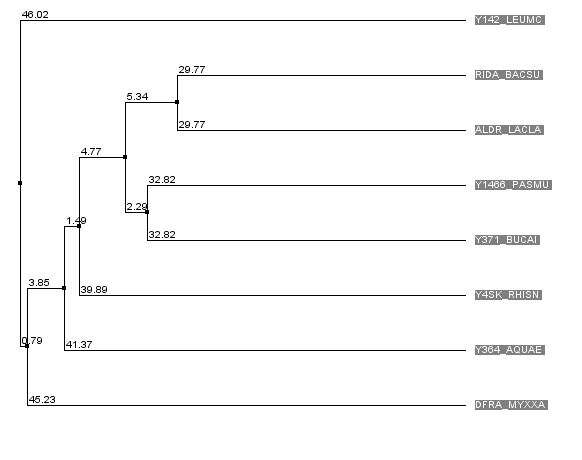
2. Программа Muscle
В SRS c помощью запроса (([swissprot-Organism:Deltavirus*] & [swissprot-Description:delta*]) & [swissprot-Description:small*]) я нашла "малые дельта-антигены", которые я надо выравнять в задании. Нашлось их 17, последовательности находок были сохранены в формате fasta.
С помощью команды muscle -in delta.fasta -out delta_aligned.fasta я выравняла последовательности белков. Полученный файл открыла в JalView (последовательности были выравнены на предыдущем шаге, на глаз лучшего выравнивания я не нашла, зачем там что-то менять?) и получила нужное выравнивание (из-за большой длины разбито на несколько частей, кликабельно).