A- и B- формы ДНК. Структура РНК
Задание 1
С помощью программы 3DNA я создала pdb-файлы, описывающие структуры A- и B-формы ДНК, состоящей из 5 повторов последовательности "gatc", и Z-формы, состоящей из 5 повторов пары GC (3DNA позволяет только так).
Получившиеся файлы - gatc-a.pdb, gatc-b.pdb, gatc-z.pdb.
Задание 2
1. Для получения изображений ниже был использован Pymol, а не Rasmol, потому что так правда гораздо легче, удобнее и красивее. Для примера того, что в Rasmol тоже можно выделять различные части ДНК, была создана эта картинка (скрипт). Некоторые картинки кликабельны.
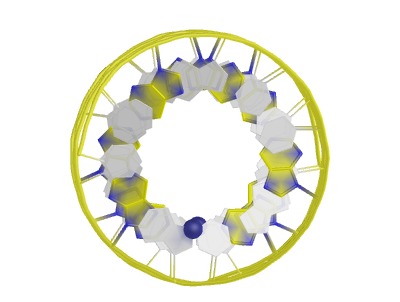
А-форма
Скрипт
Pse-файл
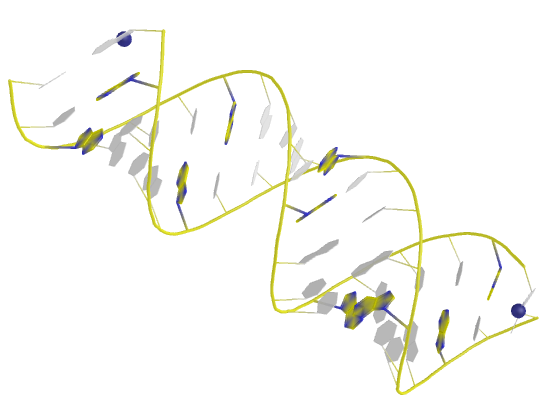
B-форма
Скрипт
Pse-файл

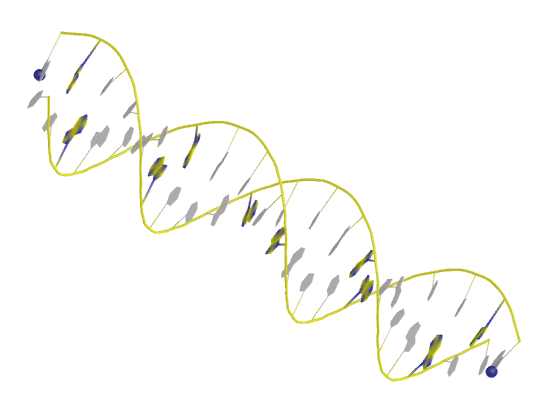
Z-форма
Скрипт
Pse-файл

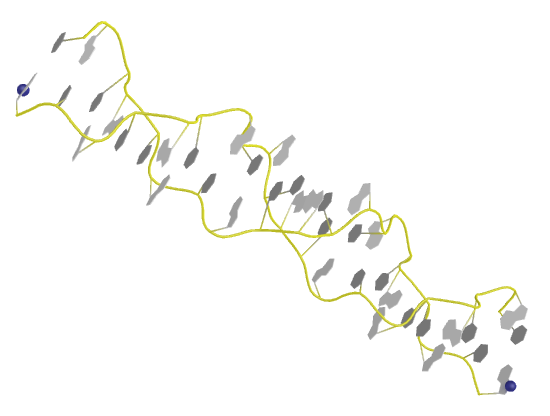
2. Исходные файлы PDB:
1P47.pdb - ДНК;
1O0B.pdb - тРНК.
3.
ДНК (pdb-файл) | тРНК (pdb-файл) |
 |  |
Задание 3
1. Заданное основание - тимин
А-форма
В сторону большой бороздки обращены атомы C4, O4, C5, C5M
В сторону малой бороздки обращены атомы C6, N1, C2, O2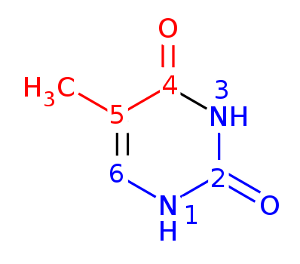
B-форма
В сторону большой бороздки обращены атомы C6, C5, C4, O4, C5M
В сторону малой бороздки обращены атомы O2, C2, N1
Остальные атомы - N3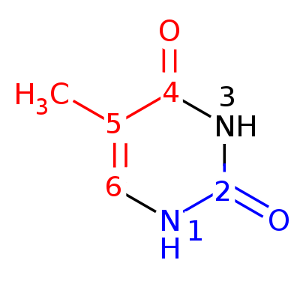
Z-форма (выбран цитозин)
В сторону большой бороздки обращены атомы N3, С4, N4, C5, C6
В сторону малой бороздки обращены атомы N1, C2, O2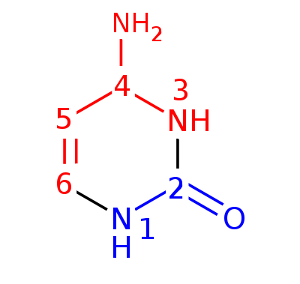
2.
| А-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (Å) | 28.0 | 33.8 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 8.0 (T'27 - C'8) | 17.2 (A'14 - C'24) | 18.3 (C'24 - C'14) или 22.5 (G'7 - G'31) |
| Ширина малой бороздки | 16.8 (C'16 - T'27) | 11.7 (T'31 - A'14) | 9.9 (C'14 - G'31) |
Изображения A-, B- и Z-форм с измеренными бороздками. Как выяснилось потом, значения ширины бороздок форм B и Z совпадают с полученными в 4м задании; значения же А-формы не совпадают совсем. Видимо, наш способ подсчета ширины как расстояния между ближайшими одноименными атомами комплементарных цепей все-таки слишком далек от реальности.
3. Скрипт для получения изображения и значений углов, вид подобран для А-формы.
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| A-форма (изм.) | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| A-форма (презент.) | -62 | 173 | 52 | 88 | 178 | -50 | -160 |
| В-форма (изм.) | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98 |
| В-форма (презент.) | -63 | 171 | 54 | 123, 131 | 155 | -90 | -117 |
Как видно, торсионные углы созданных структур и их значения из презентации отличаются, причем иногда немного (альфа-углы A-формы), а иногда просто кардинально.
Задание 4
1. С помощью команд remediator, find_pair и analyze была проанализирована структура имеющихся нуклеиновых кислот. В том числе, были получены файлы gatc-a.out, gatc-b.out, gatc-z.out, dna_old.out, rna_old.out с необходимой информацией.
Значения углов для A- и B-форм, во-первых, одинаковы для всех нуклеотидов, а, во-вторых, полностью совпали с измеренными в задании 3. Полученные значения для Z-формы:
base alpha beta gamma delta epsilon zeta chi C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7
Как видно, они различны для цитозина и гуанина, что и придает всей молекуле такую странную зигзагообразную форму.
Были определены углы в тРНК (файл). Больше всего эта тРНК похожа на А-форму ДНК, но углы краевых нуклеотидов стеблей сильно отличаются от "обычных".
Были определены углы в ДНК (файл). Все подсчеты и сравнения были сделаны в torsion.ods, здесь приведу конечные результаты.
Значения "средних" углов:
alpha beta gamma delta epsilon zeta chi -38,0 56,7 20,5 133,9 -110,2 -101,4 -118,8
Нуклеотид с наиболее отклоняющимися значениями - C'18 второй цепи (настоящий номер 929).
98,8 -141,2 -161,2 111,3 -142,9 -91,4 -154,3
Самые большие расхождения - в углах альфа, гамма, эпсилон и хи.
3. Номера нуклеотидов, образующих водородные связи, приведены в файле rna_old.out. Справа соответствующими буквами помечены пары, входящие в стебли (раскрашены на рисунке разными цветами: g, b, y и r):
Strand I Strand II Helix 1 (0.014) B:.902_:[..G]G-----C[..C]:.971_:B (0.007) | g 2 (0.007) B:.903_:[..G]G-----C[..C]:.970_:B (0.007) | g 3 (0.005) B:.904_:[..G]G-----C[..C]:.969_:B (0.005) | g 4 (0.009) B:.905_:[..G]G-----C[..C]:.968_:B (0.007) | g 5 (0.012) B:.906_:[..U]U-----A[..A]:.967_:B (0.010) | g 6 (0.012) B:.907_:[..A]Ax----U[..U]:.966_:B (0.009) | g 7 (0.005) B:.949_:[..C]C-----G[..G]:.965_:B (0.009) | b 8 (0.004) B:.950_:[..G]G-----C[..C]:.964_:B (0.003) | b 9 (0.003) B:.951_:[..A]A-----U[..U]:.963_:B (0.002) | b 10 (0.007) B:.952_:[..G]G-----C[..C]:.962_:B (0.005) | b 11 (0.005) B:.953_:[..G]G----xC[..C]:.961_:B (0.006) | b 12 (0.002) B:.954_:[..U]U-*--xA[..A]:.958_:B (0.010) | b 13 (0.003) B:.955_:[..U]Ux**+xG[..G]:.918_:B (0.005) x 14 (0.009) B:.937_:[..A]A-*---U[..U]:.933_:B (0.006) | r 15 (0.004) B:.938_:[..U]U-*---U[..U]:.932_:B (0.005) | r 16 (0.004) B:.939_:[..U]U-----A[..A]:.931_:B (0.003) | r 17 (0.003) B:.940_:[..C]C-----G[..G]:.930_:B (0.009) | r 18 (0.005) B:.941_:[..C]C-----G[..G]:.929_:B (0.010) | r 19 (0.006) B:.942_:[..G]G-----C[..C]:.928_:B (0.005) | r 20 (0.009) B:.943_:[..G]G-----C[..C]:.927_:B (0.008) | r 21 (0.005) B:.944_:[..C]Cx*---A[..A]:.926_:B (0.009) | r 22 (0.011) B:.910_:[..G]G-----C[..C]:.925_:B (0.005) | y 23 (0.002) B:.911_:[..C]C-----G[..G]:.924_:B (0.009) | y 24 (0.002) B:.912_:[..C]C----xG[..G]:.923_:B (0.011) | y 25 (0.004) B:.913_:[..A]A-**+xA[..A]:.945_:B (0.005) | 26 (0.005) B:.914_:[..A]A-**-xU[..U]:.908_:B (0.009) | 27 (0.007) B:.915_:[..G]Gx**+xC[..C]:.948_:B (0.019) x 28 (0.026) B:.919_:[..G]G-----C[..C]:.956_:B (0.003) +
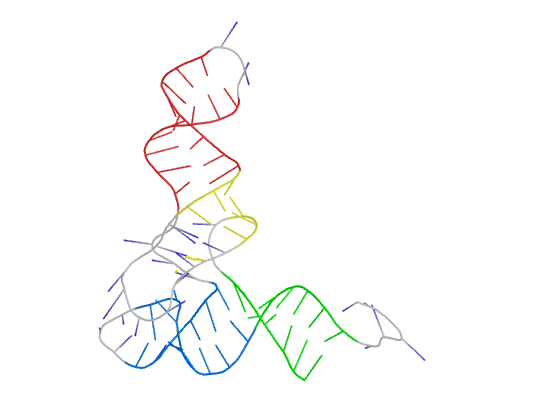
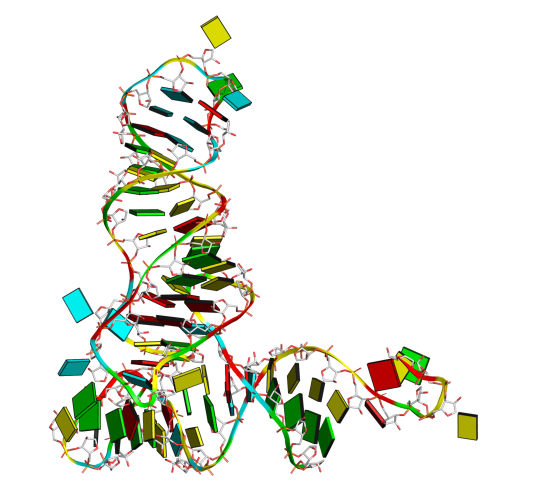
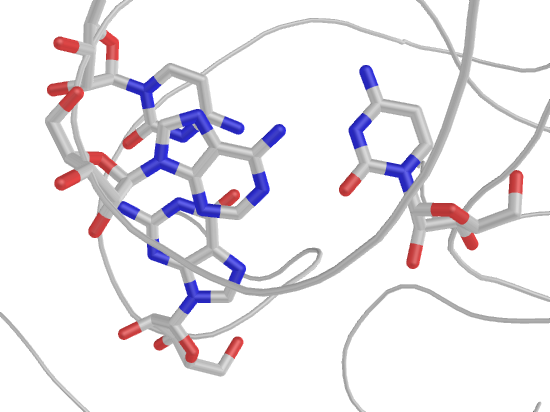
В структуре есть 4 неканонические пары: 13 U-G, 15 U-U, 21 C-A и 25 A-A. Изображения двух из них, с урацилом:
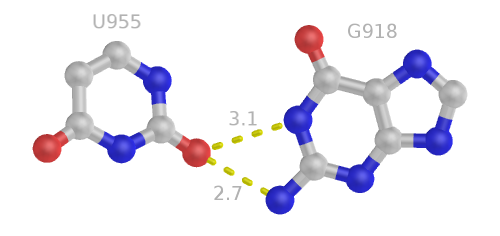
Пары, не задействованные в стеблях, формируют третичную структуру тРНК. Так, пары 13 (U955 - G918, нарисованная выше неканоническая) и 28 (G218 - C956) образованы между основаниями одних и тех же двух петель: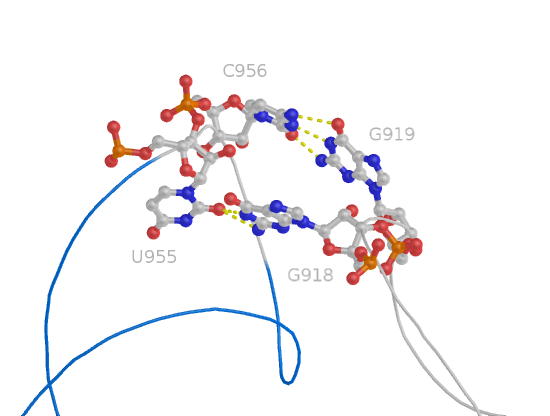
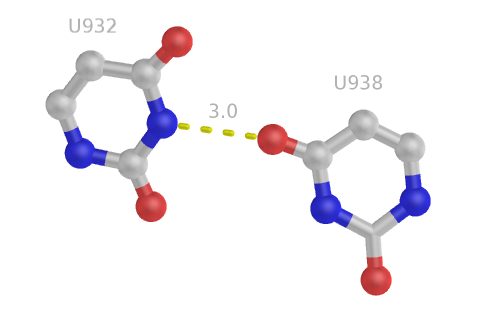
26 (A914 - U908) и 27 (G915 - C948) пары образуют "завиток" одной цепи и соединяют ее с другой, как видно на рисунке: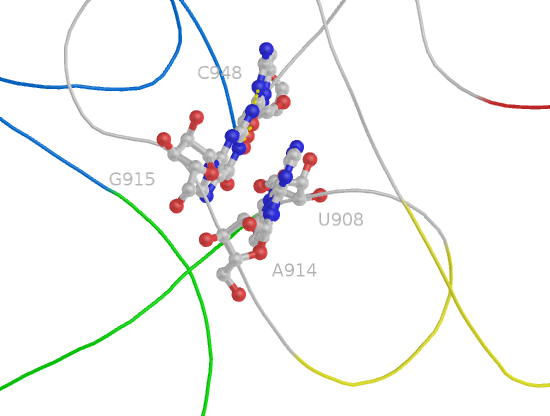
4. Пары оснований с наибольшими значениями перекрываний:
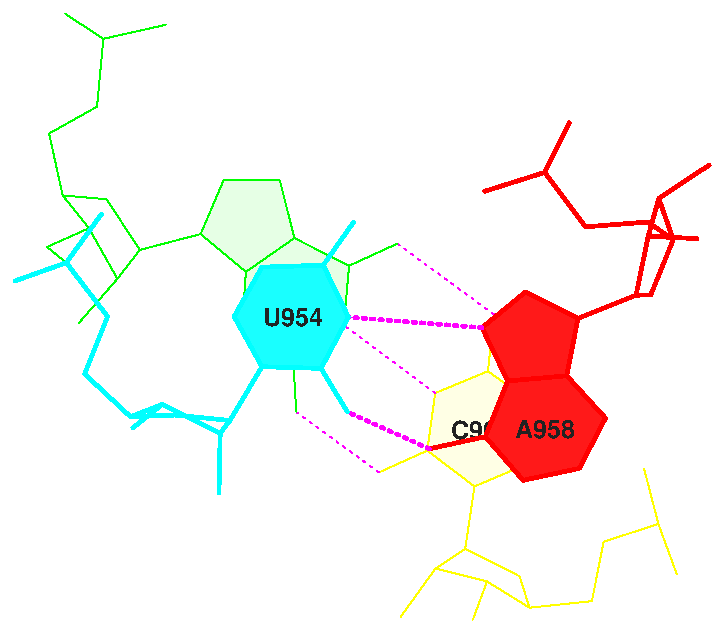
4 GU/AC 6.22( 3.44) 0.00( 0.00) 0.00( 0.00) 4.03( 2.48) 10.26( 5.92) 11 GU/AC 6.53( 3.69) 0.00( 0.00) 0.00( 0.00) 3.76( 1.46) 10.29( 5.15) 20 GC/AC 5.97( 3.57) 0.00( 0.00) 0.00( 0.00) 5.55( 3.05) 11.52( 6.62) 22 GC/GC 4.56( 1.74) 0.00( 0.00) 0.00( 0.00) 6.96( 3.74) 11.52( 5.47)
И равными нулю или наименьшими:
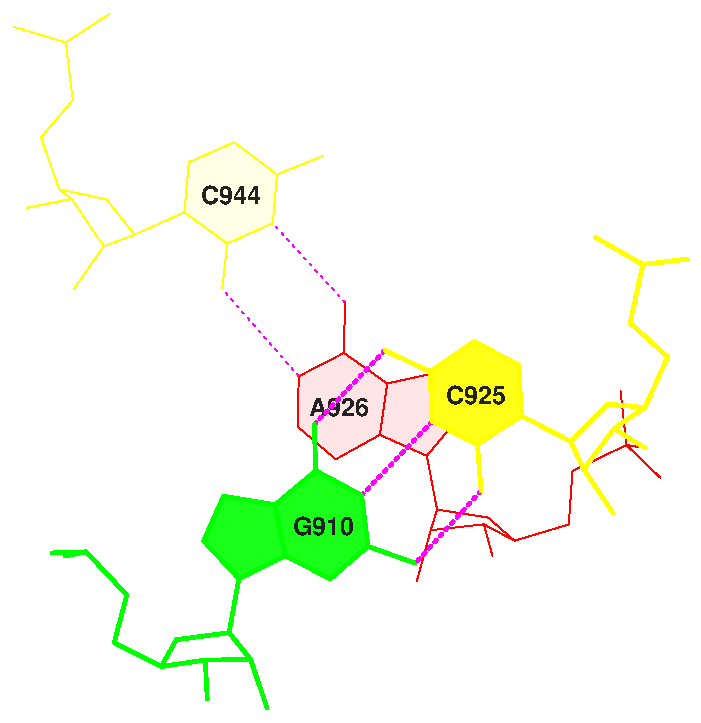
13 UA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 21 CG/CA 0.00( 0.00) 0.00( 0.00) 0.18( 0.00) 1.35( 0.72) 1.53( 0.72) 27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Изображения их стекинг-взаимодействий:
| Step 21 | Step 11 |
 |  |
| Step 20 | Step 22 |
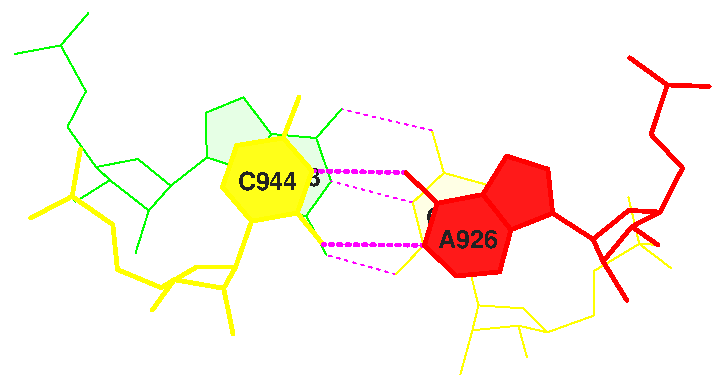 | 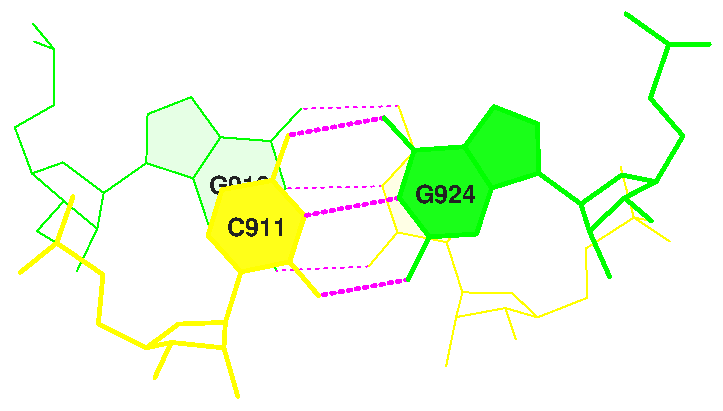 |
| Step 4 | Step 13 |
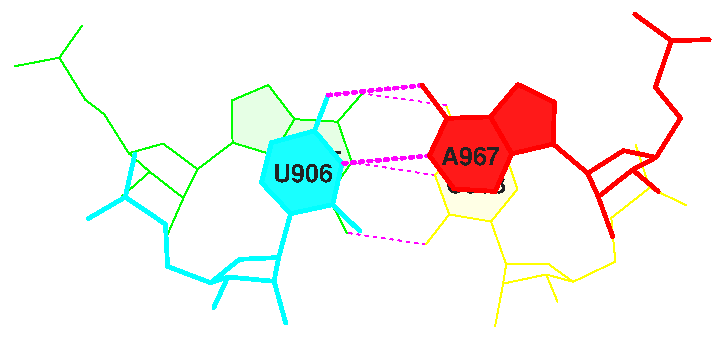 |  |
| Step 27 | |
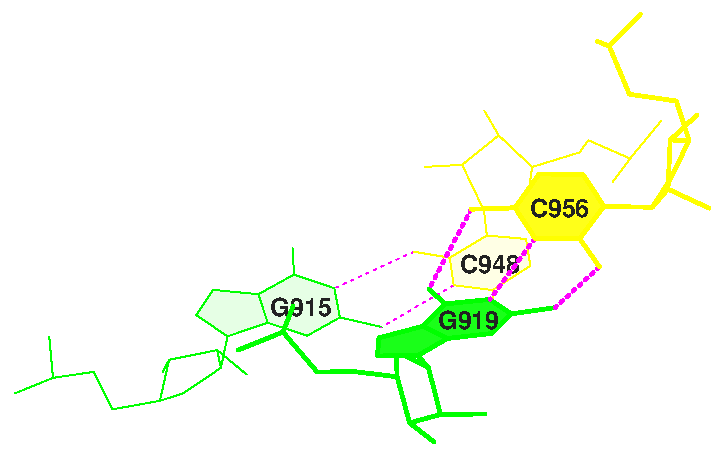 |
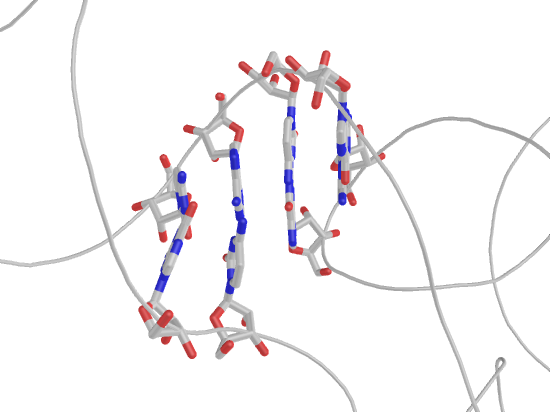
Взаимная ориентация 20 и 21 пар (step20, большое перекрывание):
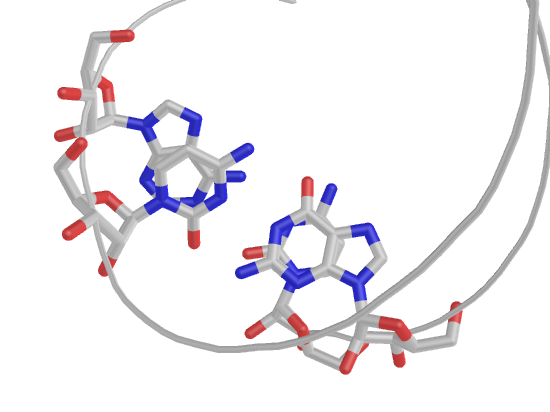
Взаимная ориентация 21 и 22 пар (step21, маленькое перекрывание):