Комплексы ДНК-белок
Задание 1: поиск ДНК-белковых контактов в заданной структуре
1. К сожалению, аналога команды pause в Pymol нет, а способа отображать разные изображения молекулы с паузами при вызове программы не из консоли я пока не нашла. Поэтому скрипт - для Rasmol (cкрипт).
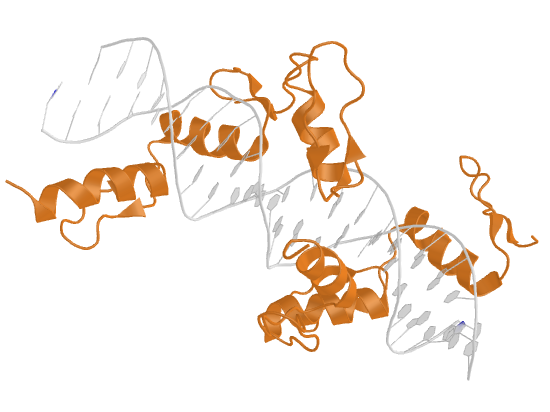


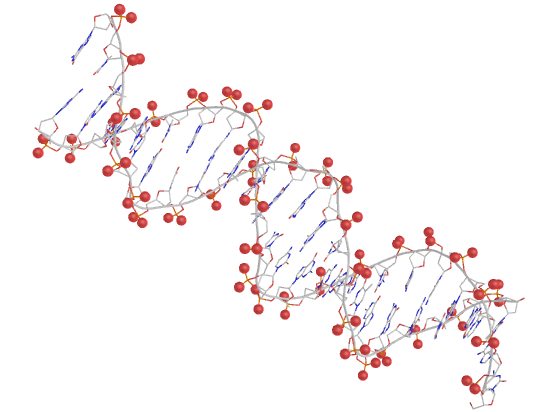
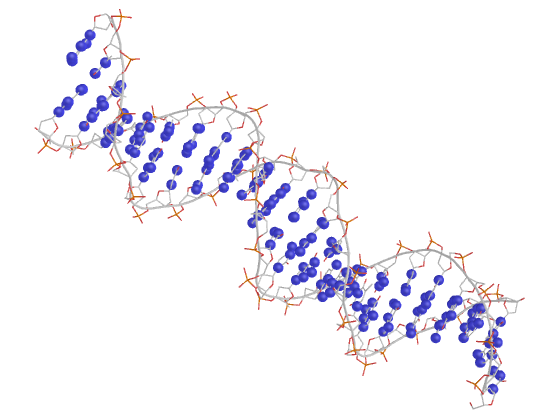
2. Для удобства были созданы множества атомов ДНК, белка, дезоксирибозы, остатков фосфорной кислоты, большой и малой бороздки, полярных и неполярных атомов. Взаимодействия с водой мы не учитываем. Возник вопрос, как считать количество ДНК-белковых контактов: если искать атомы белка около ДНК, то найдется одно число атомов, а если наоборот - другое. Это логично, так как атом может образовывать контакты не с одним, а несколькими другими атомами, а в некоторых "скоплениях" подходящих атомов определить настоящее число контактов сложно. Поэтому я привожу как число атомов белка, участвующих в контактах, так и число атомов ДНК. Pse-файл со всеми выделенными множествами и прочим: all.pse.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | Белок: 2 (азоты аргинина) ДНК: 2 (O3') | Белок: 24 ДНК: 22 | Белок: 26 ДНК: 24 |
| остатками фосфорной кислоты | Белок: 17 (N и O) ДНК: 16 (OP1 и OP2) | Белок: 21 ДНК: 13 (фосфоры) | Белок: 38 ДНК: 29 |
| остатками азотистых оснований со стороны большой бороздки | Белок: 25 ДНК: 31 | Белок: 32 ДНК: 34 | Белок: 57 ДНК: 65 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Множества атомов оснований в большой и малой бороздках были взяты из презентации (кстати, там опечатка - у тимина в малой бороздке написан C6). Как видно, атомы малой бороздки никаких взаимодействий с белком не образуют.
Понятно, что полярных контактов меньше, чем неполярных, потому что для них мы берем расстояние взаимодействия 3.5 А против 4.5 А (если брать 4.5, атомов находится примерно столько же, но это уже не контакт). Также всего два полярных взаимодействия с дезоксирибозой, видимо потому, что она обращена вглубь ДНК и защищена атомов белка. Относительно большая разница контактирующих неполярных атомов белка и остатков фосфорной кислоты ДНК (21/13) показывает, что каждый атом фосфора взаимодействует не с одним атомом, а сразу с несколькими.
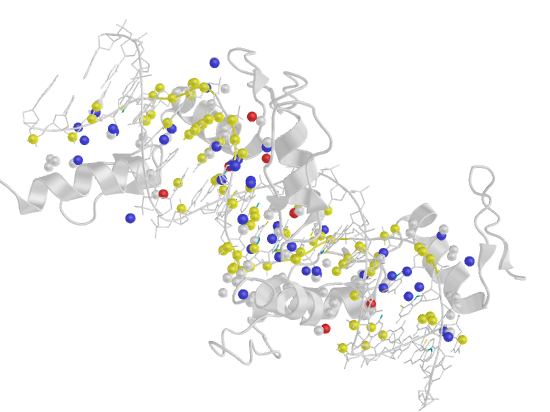
3. Схема ДНК-белковых контактов (nucplot):
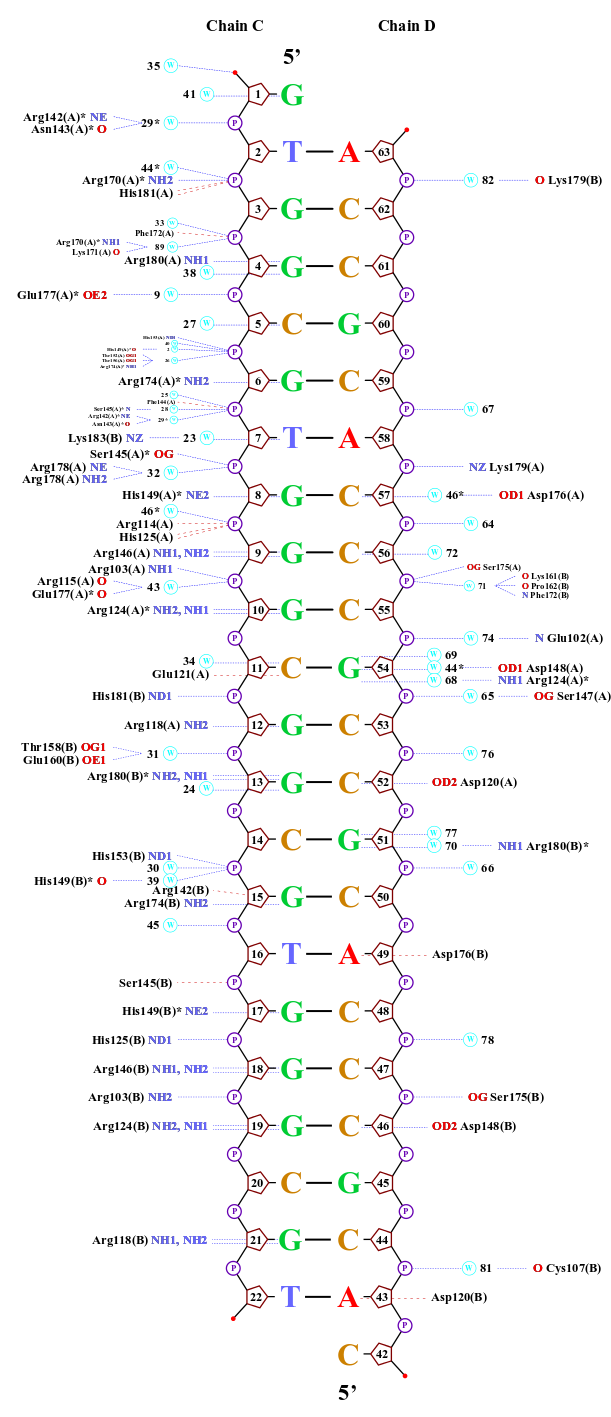
4. В структуре очень много аминокислотных остатков белка участвуют в двух контактах (помечены звездочкой на схеме), и ни один (!) - в трех или больше. Однако надо учесть, что в схеме показаны в том числе и взаимодействия белка с водой, а нам нужны только с ДНК; к тому же, взаимодействие остатка с одним элементом ДНК сразу по двум атомам засчитывается как одно. В итоге для визуализации были выбраны остатки Arg180 и His181 цепи А, так как они находятся рядом образуют в сумме две водородные и одну неполярную связи с ДНК (то же самое в цепи В). И вообще как-то этот участок белка обращает на себя внимание, если честно.
На расстоянии меньше 3.35 от 181-го гистидина находится только фосфатная группа ДНК, причем взаимодействовать по этим параметрам она может только с ND1-атомом гистидина. Поэтому, видимо, контакт существует у OP1 c ND1 и OP2 с ND1 (кислороды выделены красными шариками, азот - синим).
В схеме указано, что водородная связь образуется с участием NH1-атома 180-го аргинина и 4-м гуанином цепи С ДНК. Подходящий атом ДНК - О6 гуанина (расстояние 3.0 А). На самом деле, точно на таком же расстоянии находятся атомы NH2 аргинина (может быть донором водородной связи) и N7 гуанина (может быть акцептором), и я не очень понимаю, почему в схеме никакого взаимодействия между ними не указывается. На рисунке выделены шариками все упомянутые атомы, а также показаны расстояния между ними.
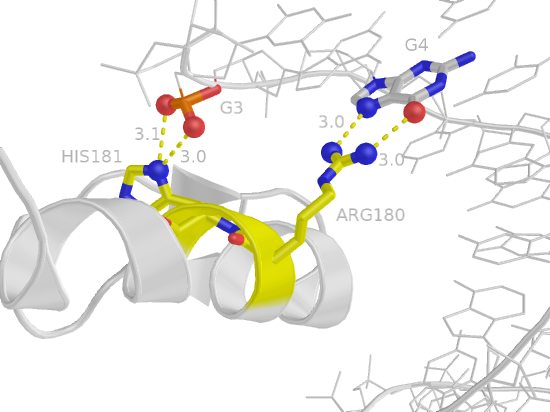
Гугл подсказал, какие именно аминокислотные остатки принимают участие в распознавании последовательности ДНК. Вообще структура белка представляет собой комплекс трех "цинковых пальцев" (каждый из альфа-спирали и бета-тяжа, связанных ионом цинка). Каждый "палец" связывает три основания ДНК: первый - GCG, второй - TGG, третий - GCG. Седьмой остаток каждого "пальца" связывает первое основание триплета, третий - второе, "минус первый" - третье (как ни странно, хорошая табличка соответствия остатков и номеров есть в соответствующей статье Википедии). Взаимодействует каждый третий остаток, так как они расположены с одной стороны от альфа-спирали.
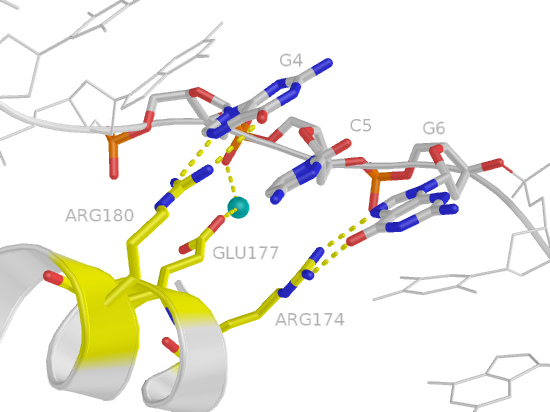
Связывающие остатки вроде бы равноценны, и наиболее важный среди них выделить нельзя. Сначала я хотела показать изображение первого из них, и тут выяснилось, что он как раз и есть случайно выбранный в предыдущем пункте 180-й аргинин. Это приятно) В итоге на изображении представлены остатки, соединенные с первым триплетом GCG: это 180-й и 174-й аргинины и 177-я глутаминовая кислота.
Глутаминовая кислота непосредственно с ДНК не взаимодействует, зато соединена водородной связью с водой и через нее с нуклеиновой кислотой (атом OE2 - HOH9 - OP2). На рисунке не показаны вторая цепь ДНК и все части белка, кроме участвующей альфа-спирали.
Задание 2: предсказание вторичной структуры заданной тРНК
1. einverted
einverted пытается найти инвертированные участки последовательности нуклеиновых кислот, в первую очередь в ДНК (пруф). Поэтому, во-первых, он переводит все урацилы в тимины (на результат, понятно, не влияет), а во-вторых, находит две цепи и линейно их выравнивает. То есть в нелинейной, с "боковыми" D- и T-стеблями тРНК восстановить реальную структуру заведомо не получится. В лучшем случае можно попробовать подобрать такие параметры, чтобы по максимуму совпали акцепторный и антикодоновый стебли, а на месте остальных стеблей будут гэпы или просто некомплементарные последовательности.
Сразу заметно, что штрафы за гэпы слишком большие, а баллы за найденные пары наоборот, слишком маленькие. При параметрах gap penalty = 6, minimum score threshold = 50, match score = 10 и mismatch score = -4 был найден более или менее оптимальный вариант:
:Score 162: 25/29 ( 86%) matches, 12 gaps
1 tggggtatc---g---ccaa--gcggtaaggcaccggattc 33
||||||| | | | || ||| || | ||||| ||
71 accccat-gctcctaagcttggagcc-ttac--ggccttag 35
Полностью (и даже с избытком, как потом в msfold) был определен акцепторный стебель. Антикодоновый стебель определился частично, чего и следовало ожидать, так как он содержит две неканонические пары. T- и D-стебли не видны вообще, что тоже понятно. Между акцепторным и антикодоновым стеблями встречаются какие-то пары, но это просто случайные совпадения, не отражающие реальную структуру РНК. Совпадшие с реальностью участки выделены цветом.
2. msfold
С первой попытки (P=5) была получена только одна структура, причем вообще не с четыремя, я с тремя стеблями. С P=15 четвертый наконец появился, но антикодоновый стебель был слишком коротким (на самом деле, просто не были учтены неканонические пары, из-за которых стебель так уменьшился). При повышении P дальше начали появляться какие-то очень странные структуры с кучей петель, мультипетель, пересекающихся стеблей и прочим адом. В итоге, та самая структура с обрезанным антикодоновым стеблем (третья, Р=15) получилась самой оптимальной.
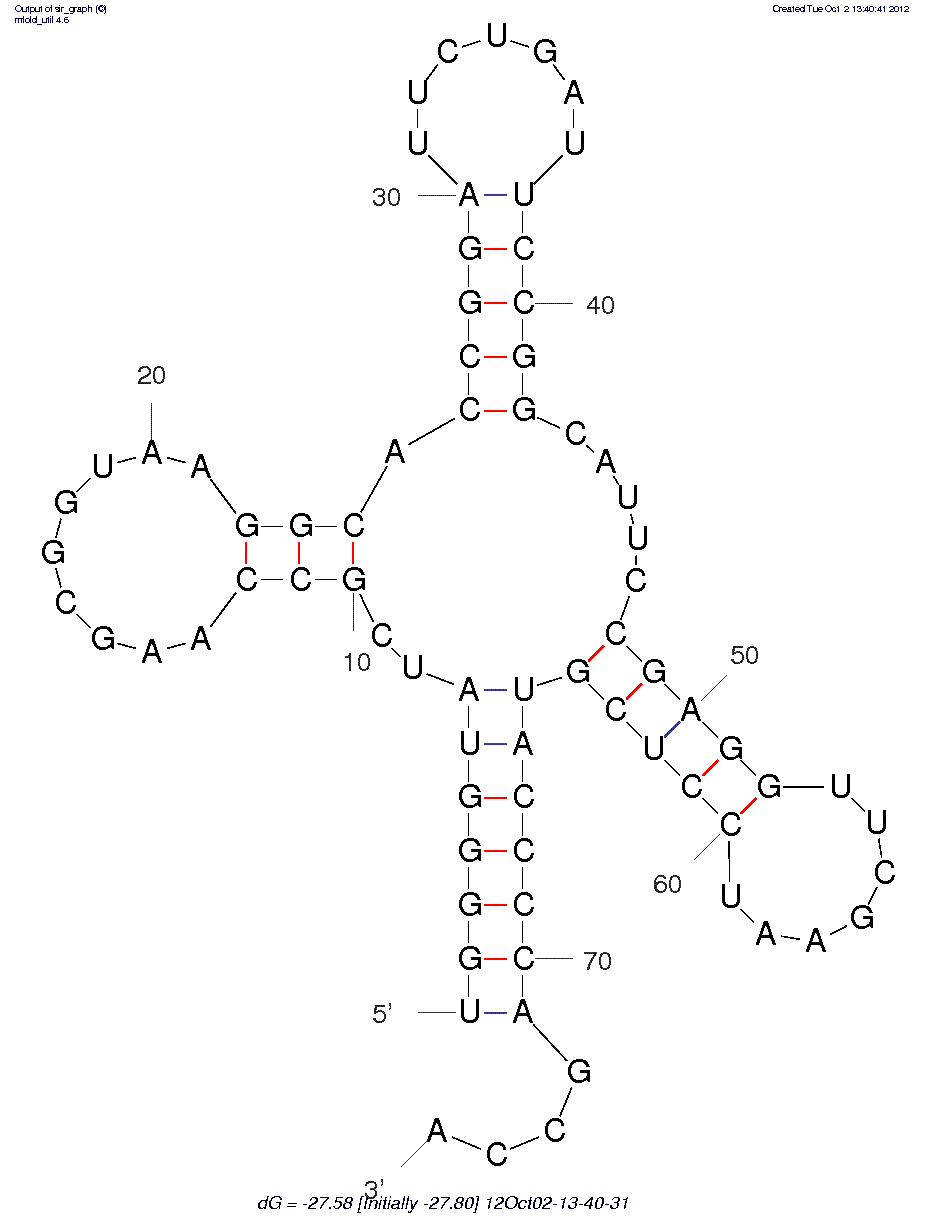
В акцепторном стебле по версии einverted и msfold на одну пару оснований больше, чем в find_pair, что как минимум странно. Выяснилось, что в pdb-файле тРНК в общей последовательности (SEQRES) терминальный урацил есть, а в координатах атомов (ATOM) о нем ни слова, соответственно, ни find_pair, ни Pymol его не видят. Ну а как поясняет поле REMARK, этот урацил просто "was not located in the experiment", как еще и группа аминокислотных остатков белка и заодно фосфор следующего за потеряным урацилом гуанина.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказаний по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-902-907-3' 5'-966-971'3' 6 пар оснований | предсказаны все пары + 1 | предсказаны все пары + 1 |
| D-стебель | 5'-910-912-3' 5'-923-925-3' 3 пары оснований | ничего | предсказаны все пары |
| T-стебель | 5'-949-954-3' 5'-961-965-3' 5 пар оснований | ничего | предсказаны все пары |
| Антикодоновый стебель | 5'-937-944-3' 5'-926-933-3' 8 пар оснований (2 неканонических) | предсказаны 5 пар из 8 реальных (первая и предпоследняя пары - неканонические и вообще не были учтены, а вместе с ними и последняя каноническая пара) | предсказаны 5 пар из 8 реальных (то же самое про неканонические пары) |
| Общее число канонических пар нуклеотидов | 20 | 12 | 18 |