EMBL
Знакомство со структурой банка EMBL посредством поисковой системы SRS
a. На странице "EMBl (Release)" можно получить всю необходимую информацию о последнем релизе банка EMBL. Как видно, текущий релиз был введен 18 сентября 2012 и включает в себя 82772678 записей.
b. По ссылке "Data class" находится страница со всеми типами данных, представленными в EMBL (последний столбец - количество записей):
| CON: | Constructed sequence | - |
| EST: | Expressed Sequence Tag | - |
| GRV: | Genome Reviews | - |
| GSS: | Genome Survey Sequence | 34528104 |
| HTC: | High Throughput cDNA sequencing | 491770 |
| HTG: | High Throughput Genome sequencing | 152599 |
| MGA: | Mass Genome Annotation | - |
| PAT: | Patent | - |
| SET: | Project set (EMBL WGS Masters only) | - |
| STD: | Standard | 13920617 |
| STS: | Sequence Tagged Site | 1322570 |
| TSA: | Transcriptome Shotgun Assembly | 7992186 |
| WGS: | Whole Genome Shotgun | - |
Как видно, в SRS не проиндексированы записи классов con, est, grv, mga, set и wgs.
c. Аналогично, на странице "Divisions" можно посмотреть все разделы записей в EMBL:
| ENV: | Пробы окружающей среды | 7762556 |
| FUN: | Грибы | 2402829 |
| HUM: | Человек | 11304977 |
| INV: | Беспозвоночные | 7398240 |
| MAM: | Другие млекопитающие (не вошедшие в другие классы) | 6741732 |
| MUS: | Mus musculus (Домовая мышь) | 5163724 |
| PHG: | Бактериофаги | 8503 |
| PLN: | Растения (+ водоросли) | 20284404 |
| PRO: | Прокариоты | 1639517 |
| ROD: | Грызуны | 1313761 |
| SYN: | Синтетические | 4045013 |
| TGN: | Трансгенные | 285306 |
| UNC: | Неклассифицированные | 8617170 |
| VRL: | Вирусы (не бактериофаги) | 1358516 |
| VRT: | Другие позвоночные (не вошедшие в другие классы) | 4446330 |
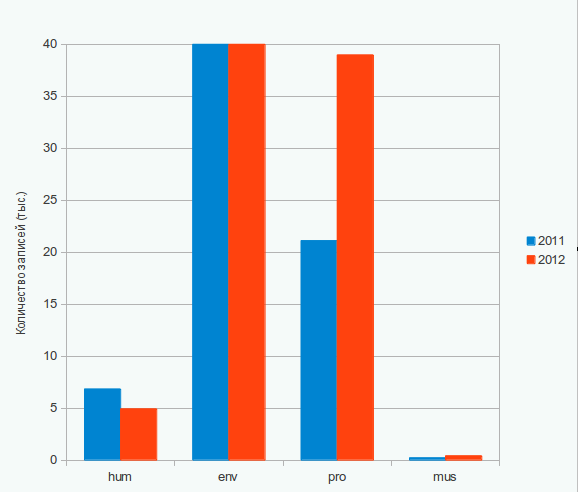
d.* Через Extended Query Form (с отмеченным флажком EMBL (release), конечно) я по очереди нашла все записи, имеющие Data Class "std", Division "hum", "env", "pro" и "mus" и Entry Creation Date от 1 января до 30 марта 2012 и 2011 года. Дата последнего обновления в этом задании, видимо, не учитывается. На приведенных ниже диаграммах видно, что почти для каждого примера в 2012 году было добавлено больше записей. Это логично, потому что процесс секвенирования упрощается, методы совершенствуются, соответственно, в банк заносится все больше и больше геномов. В случае с геномом человека, однако, этот принцип не сохранятся, не очень понятно почему. Может, из-за дат выхода релизов? Подробнее см. ниже. Стоит отметить, что, например, для категории трансгенных организмов такая тенденция не наблюдается (там около 15-и записей за январь-март 2011 и 9 за 2012), но эта категория вообще "необычная". Да и малое число записей не позволяет сделать какие-либо выводы. Графики представляют одни и те же данные, просто сделаны в разном масштабе: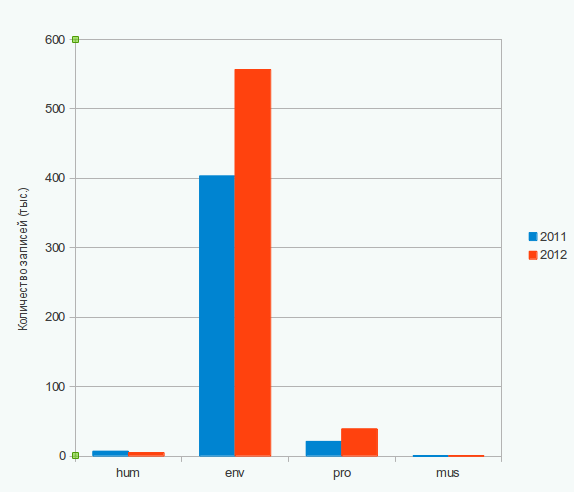
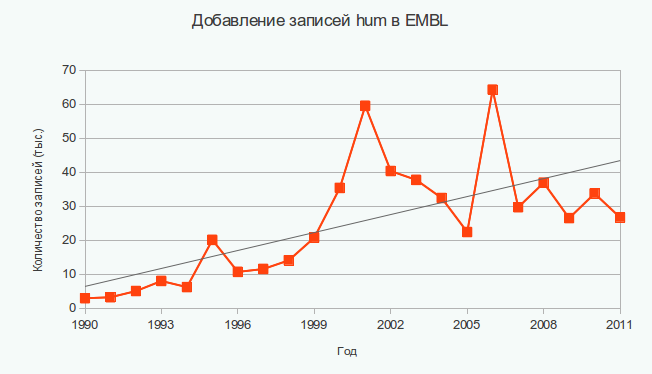
Ради интереса я также построила график появления записей, относящихся к человеку, с 1990 по 2011 год. Видно, что хоть в целом и наблюдается рост (особенно вначале), он крайнее неравномерен, например, есть пики в 1993, 2001 и 2006 годах. Я не знаю, с чем связать это явление, кроме неравномерности выхода релизов, из-за которых в какие-то года добавлялось больше записей, чем в другие. Я брала "разбиение" в датах год, так как при более маленьком это влияние было слишком заметным. Может быть, в 90-х был какой-то прорыв в технологиях и "бум" на секвенирование всего-всего из человека, и рост происходил из-за этого, а ближе к настоящему времени наблюдается некоторая стагнация. Предположений много, какие из них правильные, я не знаю, но график все равно интересный.
Описание гена в записи бенка EMBL
Ген, доставшийся мне - LTB. В файле BA000025 я нашла информацию об этом гене (отдельный файл для удобства). Можно сделать вывод, что:
- направление гена относительно направления, выбранного для записи, прямое;
- кодирующих участков (экзонов) четыре: 361609..361770, 362167..362212, 362396..362467, 362863..363317;
- длина первого экзона - 162 нуклеотида, последнего - 455;
- длина первого интрона - 396 нуклеотидов, последнего - 395.
Нахождение белка по фрагменту гена
Самый длинный кодирующий участок белка LTB - последний, четвертый (362863..363317), его длина составляет 455 нуклеотидов. С помощью соответствующей команды я вырезала в отдельный файл его последовательность:
$ seqret -sask
Reads and writes (returns) sequences
Input (gapped) sequence(s): BA000025.embl
Begin at position [start]: 362863
End at position [end]: 363317
Reverse strand [N]:
output sequence(s) [ba000025.fasta]:
Чтобы найти в Swiss-Prot белок, соответствующий этому гену, на сайте http://blast.ncbi.nlm.nih.gov/ я прошла по ссылке blastx (как объясняет комментарий, эта программа как раз и осуществляет поиск по банкам белков, используя последовательность нуклеотидов), выбрала там в качестве искомой последовательности файл ba000025.fasta - результат вывода команды seqret и установила Database для поиска UniProtKB/Swiss-Prot. Для упрощения работы в Algorithm parameters я уменьшила количество выводимых результатов до минимума (зачем нам много последовательностей, мы ищем одну, самую подходящую) и подняла порог для поиска, чтобы не находились лишние белки. В полученных результатах очевидно, что нужный белок - TNFC_HUMAN. Его идентичность с исходной последовательностью экзона равна 100%, он первый в списке результатов, в общем, в его пользу говорит абсолютно все, даже не пришлось придумывать более эффективные параметры поиска. Стоит отметить, что следующие строчки в результатах занимают аналогичные белки из других организмов.
Если посмотреть запись найденного белка в Swiss-Prot и найти там его последовательность, окажется, что она полностью совпадет с последовательностью из поля "FT /translation=" " embl-файла, что лишний раз свидетельствует, что находка правильная. На самом деле, можно было искать в базе данных белков сразу по этой последовательности уже данных аминокислот, но чтобы уж лишний раз убедиться, что этот ген на самом деле кодирует то, что написано...
Из выравнивания, данного программой BLAST, видно, что данному экзону соответствуют аминокислотные остатки 95-244 найденного белка:
>sp|Q06643.1|TNFC_HUMAN RecName: Full=Lymphotoxin-beta; Short=LT-beta; AltName: Full=Tumor
necrosis factor C; Short=TNF-C; AltName: Full=Tumor necrosis
factor ligand superfamily member 3
Length=244
GENE ID: 4050 LTB | lymphotoxin beta (TNF superfamily, member 3) [Homo sapiens]
(Over 10 PubMed links)
Score = 301 bits (771), Expect = 7e-103, Method: Compositional matrix adjust.
Identities = 150/150 (100%), Positives = 150/150 (100%), Gaps = 0/150 (0%)
Frame = +3
Query 3 APLKGQGLGWETTKEQAFLTSGTQFSDAEGLALPQDGLYYLYCLVGYRGRAPPGGGDPQG 182
APLKGQGLGWETTKEQAFLTSGTQFSDAEGLALPQDGLYYLYCLVGYRGRAPPGGGDPQG
Sbjct 95 APLKGQGLGWETTKEQAFLTSGTQFSDAEGLALPQDGLYYLYCLVGYRGRAPPGGGDPQG 154
Query 183 RSVTLRSSLYRAGGAYGPGTPELLLEGAETVTPVLDPARRQGYGPLWYTSVGFGGLVQLR 362
RSVTLRSSLYRAGGAYGPGTPELLLEGAETVTPVLDPARRQGYGPLWYTSVGFGGLVQLR
Sbjct 155 RSVTLRSSLYRAGGAYGPGTPELLLEGAETVTPVLDPARRQGYGPLWYTSVGFGGLVQLR 214
Query 363 RGERVYVNISHPDMVDFARGKTFFGAVMVG 452
RGERVYVNISHPDMVDFARGKTFFGAVMVG
Sbjct 215 RGERVYVNISHPDMVDFARGKTFFGAVMVG 244Ссылки из записи банка Swiss-Prot на записи банка EMBL
Данный мне белок - YABJ_BACSU. Хочу обратить внимание, что еще в прошлом семестре, как раз в процессе моей работы над белком, он был изучен чуть лучше и его ID поменялся на RIDA_BACSU. AC, разумеется, остался прежним, на то он и AC - P37552.
Сначала я получила .entret файл записью Swiss-Prot о моем белке:
$ entret sw:P37552 -auto
В полученном файле rida_bacsu.entret в поле DR я нашла информацию о соответствующих записях EMBL. Записей всего две:
DR EMBL; D26185; BAA05283.1; -; Genomic_DNA. DR EMBL; AL009126; CAB11824.1; -; Genomic_DNA.
Для того, чтобы заполнить таблицу, конечно, удобнее всего следовать инструкции в задании, то есть зайти на Library Page, воспользоваться поиском и отметить нужные для вывода поля (что я и сделала). Однако для дальнейшей работы мне удобнее иметь полноценные файлы этих записей: D26185.entret и AL009126.entret этих записей (кстати, довольно большие):
entret embl:D26185 -auto entret embl:AL009126 -auto
ID записи |
Тип молекулы |
Класс данных |
Дата внесения в банк |
Описание |
Длина последовательности |
D26185 |
genomic DNA |
STD |
05-FEB-1994 |
Bacillus subtilis gene, 180 kilobase region of replication origin. |
180136 |
AL009126 |
genomic DNA |
STD |
18-JUL-2002 |
Bacillus subtilis subsp. subtilis str. 168 complete genome. |
4215606 |
Как видно, первая запись - последовательность участка генома длиной 180 килобаз, в который попал ген, кодирующий белок rida_bacsu. Причем интересно, что тогда, в 1994 году, еще не знали, что это за белок (/product="unknown") и какую функцию он несет. Белку просто был присвоен свой АС, а изучен он был уже позднее.
Ну а вторая запись представляет собой полный геном Bacillus subtilis, последовательность которого была получена в 2002 году, когда секвенирование вообще значительно упростилось и стало возможным для целых геномов. Туда, закономерно, попал и ген с моим белком. К тому времени он был изучен уже гораздо лучше, и в записи можно найти о нем некоторую информацию:
FT /gene="yabJ" FT /product="putative enzyme resulting in alteration of gene FT expression" FT /function="16.3: Control" FT /function="16.6: Maintain" FT /note="Evidence 3: Function proposed based on presence of FT conserved amino acid motif, structural feature or limited FT homology; PubMedId: 12515541, 14729707, 10557275, 10919400, FT 10368157; Product type pe: putative enzyme"
Однако видно, что тогда он был исследован еще недостаточно и что он пока сохранял имя "YABJ", которое недавно, как уже упоминалось, было заменено на "RIDA".