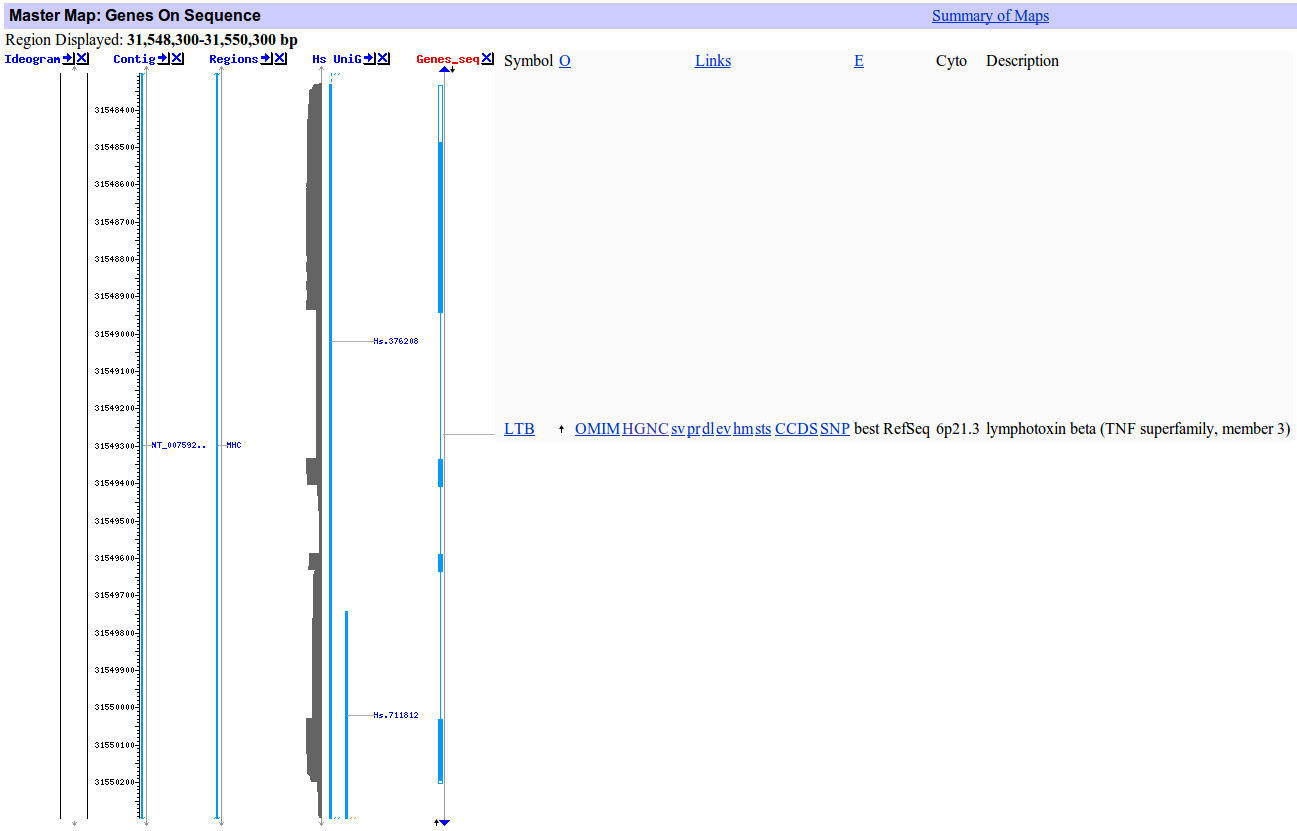
Геномные браузеры
Сначала я провела поиск по самому длинному, последнему, экзону LTB с помощью BLAT. Что мы можем видеть в результатах поиска:
- картинку с местонахождением гена в человеческом геноме (как видно, это шестая хромосома)
- картинку с выравниванием соответствующего участка генома и данной для поиска последовательности (полное совпадение, смотреть неинтересно);
- информацию об этом выравнивании. Здесь мы можем узнать, что этот экзон расположен (еще раз) в шестой хромосоме, координаты 31 548 686 - 31 548 940, на обратной цепи. Искомый экзон и соответствующий участок генома абсолютно идентичны.
Query Chromosome Stats Start End Ori Name Start End Ori Score E-val %ID Length 1 255 - Chr:6 31548686 31548940 + 1309 5.9e-224 100.00 255
Находка только одна.
Чтобы узнать больше, переходим на "ContigView".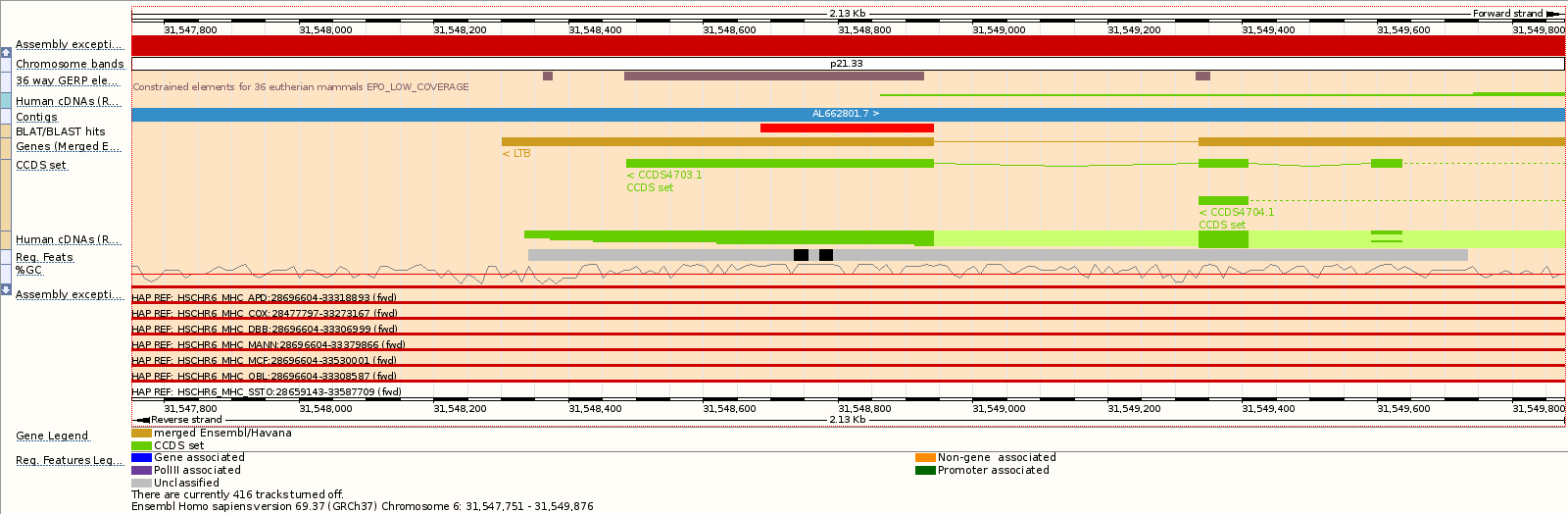
Мы видим соответствующий участок хромосомы. В Region in detail можно подробно посмотреть, какие гены и псевдогены здесь присутствуют. И правда, один из них - искомый LTB, точнее, один из его экзонов. На более крупной картинке мы видим ту же находку BLAT и автоматически аннотированный геном. Возможности ensEMBL позволяют менять масштаб изображения, отображать на ней разные элементы или скрывать их, например, экзоны, подписи и так далее. Можно воспользовать бета-функцией scrolling и "прокрутить" геном, например, до следующего экзона того же гена.
В виде collapsed этого участка прекрасно заметно, что это на самом деле есть часть гена LTB и как точно он расположен. Также здесь мы видим соответствующие CCDS, но о них я хочу рассказать чуть позднее.
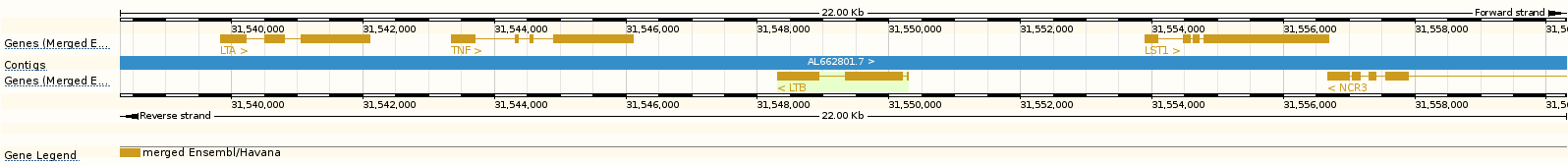
Теперь, кликнув по изображению экзона на "карте" хромосомы, перейдем на страничку гена, ведь нас интересует именно он.
Ген LTB (лимфотоксин бета, TNF superfamily) расположен, как мы уже видели, на обратной цепи, с другими генами не пересекается. "Недалеко" от него на прямой цепи расположен связанный по функции ген LTA.
Мы видим, что у этого гена есть четыре транскрипта, из которых два кодируют белки:
- LTB-001, транскрипт, CCDS4703;
- LTB-002, транскрипт, CCDS4704.
Изображение двух этих транскриптов:
Можно посмотреть более ясную картину сплайсинга всех четырех транскриптов. Тут же видны домены в белках, характерные для семейств.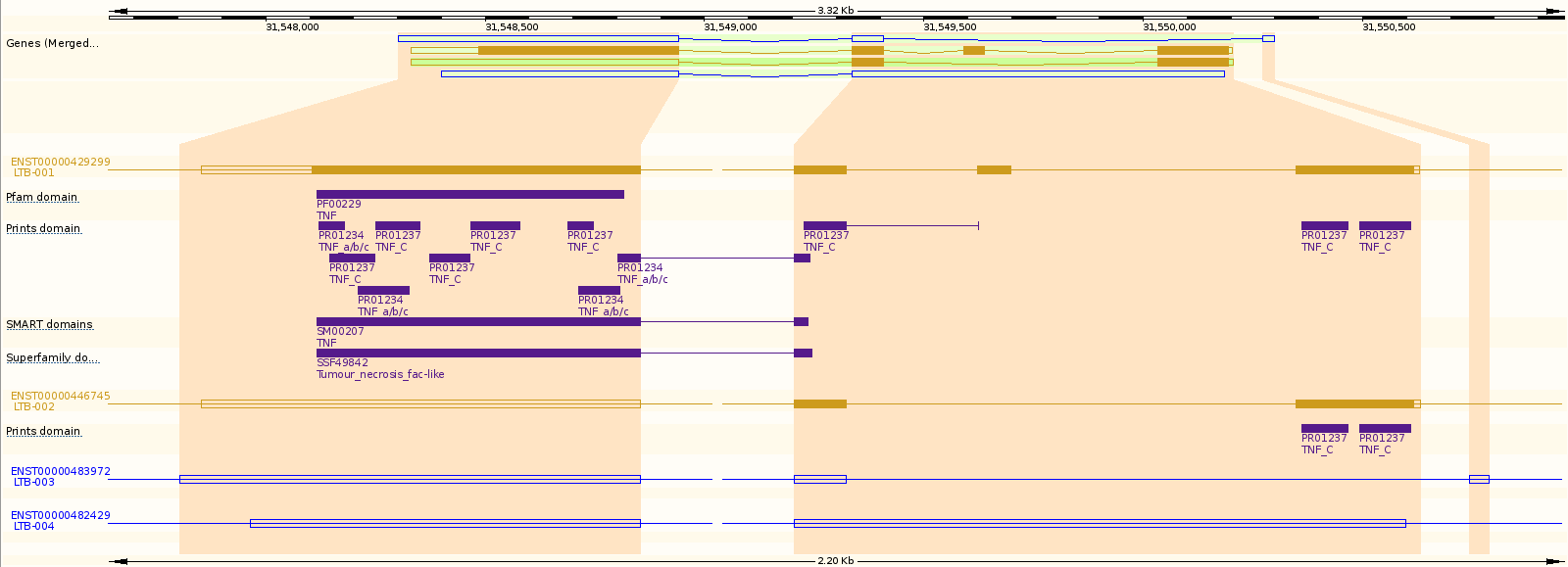
Также есть возможность посмотреть гомологов белка из разных организмов, например, в виде дерева. Как видно, самыми близкими гомологами явлюятся соответствующие белки гориллы и шимпанзе, результат логичный и ожидаемый.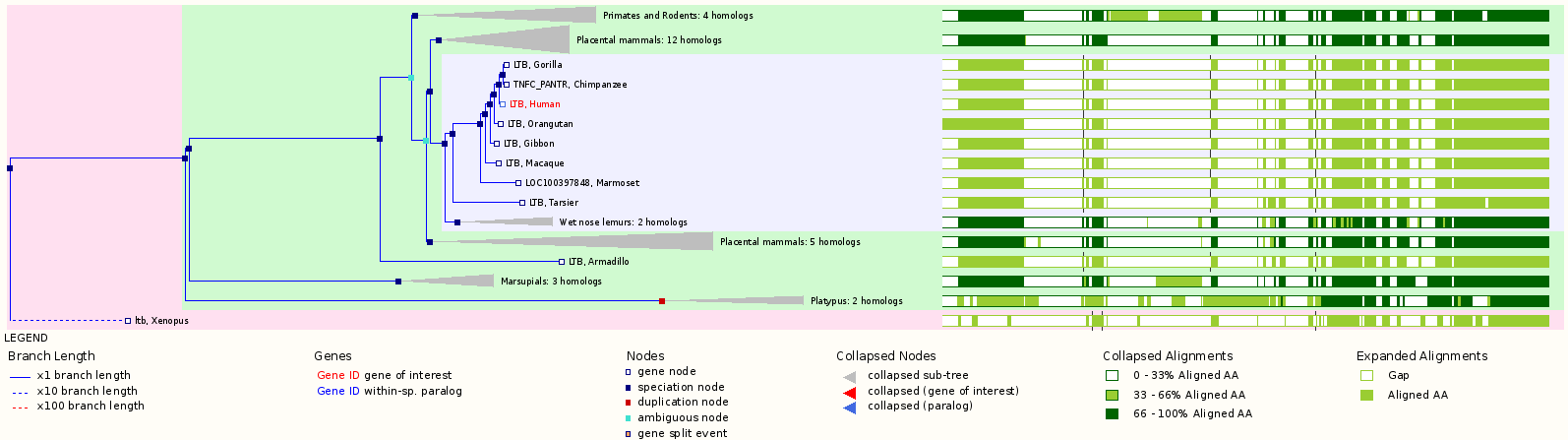
Есть и другие способы попасть на страницу нужного гена, помимо поиска с помощью BLAT по экзону. Во-первых, можно сначала найти нужную запись в EMBL (данная мне находится просто по названию гена), а с ссылки на ней выйти на соответствующу запись enseMBL. Во-вторых, можно поискать сразу в ensEMBL по названию в геноме человека. При этом в случае LTB находится восемь записей генов. Из них один - тот, который нам нужен, а остальные семь - с локацией в "HSCHR6_MHC_MCF". Понятно, что это тот же ген в той же шестой хромосоме, но у них разные записи в EMBL (некурируемая база данных, такое вполне возможно), а название локации странное. Может, потому, что они не входят в "официальную" аннотацию? Тогда они должны иметь альтернативную локацию, чтобы не отображаться при непосредственном просмотре хромосомы.
Vega. Это практически такой же геномный браузер, как ensEMBL, предоставляет те же возможности и даже выглядит так же. О нем рассказывать не очень интересно.
Ucsc. Видны все mRNA и EST, можно отследить альтернативный сплайсинг. Собственно, видимая картина совпадает с описанным выше, повторяться не хочется, поэтому - просто картинка. Вообще сервис известен как genome browser и помогает осуществить поиск по геному и посмотреть возможные варианты транскрипции и сплайсинга. Также есть дополнительные инструменты, например, BLAT и другие.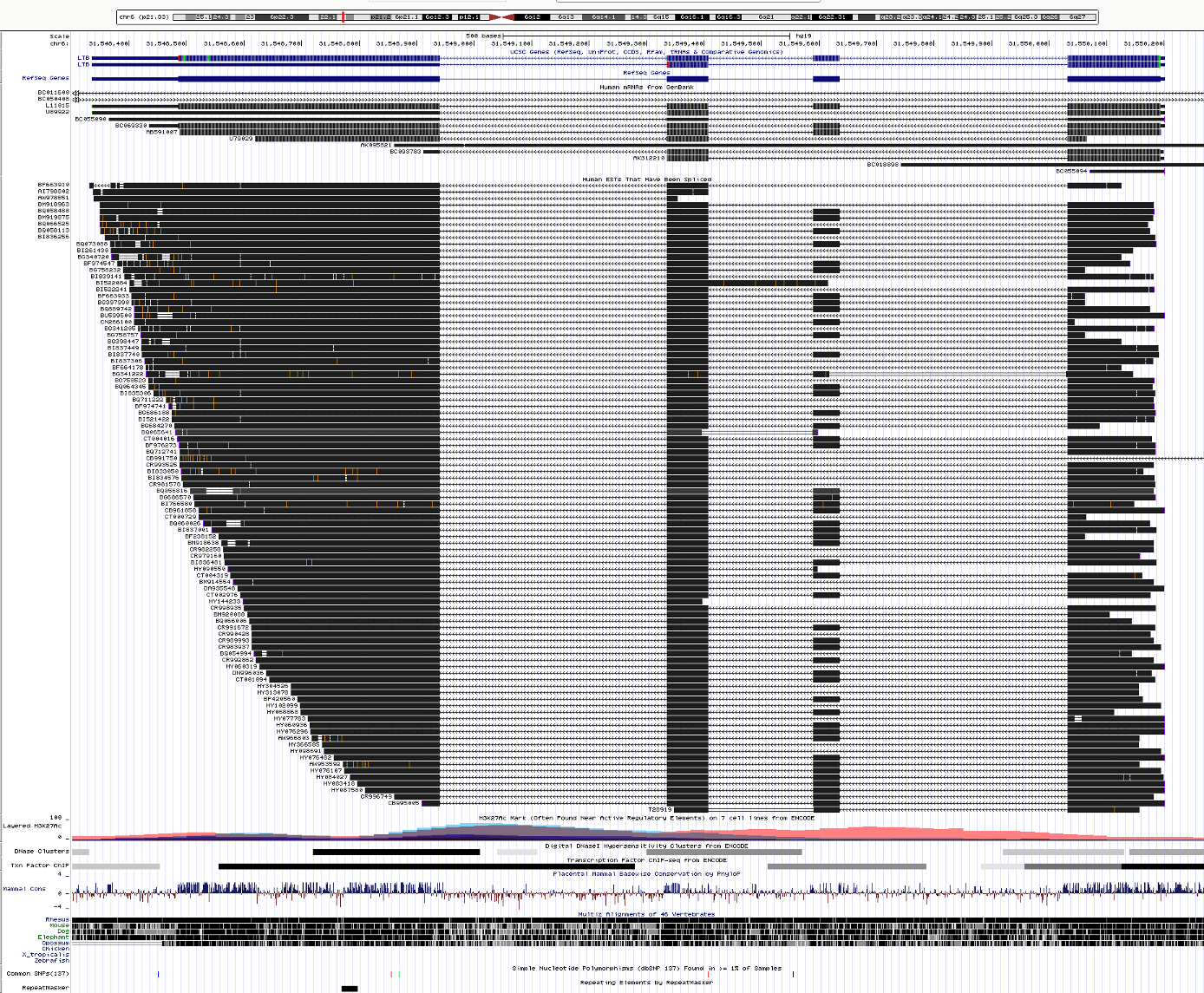
NCBI. Он уже известен нам, но тут, по ссылке в ensEMBL, он показывает карту гена (раньше я не знала, что он имеет такую возможность). Такая карта выглядит не так, как в ensEBML. Во-первых, она вертикальная (лично мне так непривычнее, но удобнее, все-таки html-страницы больше рассчитаны на прокручивание по вертикали, чем по горизонтали). Во-вторых, она может представлять несколько иные данные. Мы можем выбирать те типы данных, которые хотим увидеть. Так же, как и в genome browser, представлены mRNA и EST гена, экзоны и интроны. И, как обычно, есть ссылки на CDS и прочие связанные с геном записи, название самого гена и его описание. Страница самого гена представляет чуть иную информацию, чем ensEMBL. Так как сам характер самого банка данных несколько иной, мы видим информацию об организме (со всей систематикой), откуда был выделен белок, подробное описание самого белка, его статус в RefSeq. Так же, как и в ensEMBL, есть представление соответствующего участка хромосомы с белками, находящимися поблизости, но оно гораздо более лаконично.