Эволюционные домены (2)
Построение филогенетического дерева
В отчете по предыдущему заданию приводились таксоны, для которых характерна одна определенная архитектура; также упоминалось, что, например, в таксоне Fungi вплоть до мелких клад встречаются все три архитектуры. Но этот таксон исключать не хочется, интересно было бы посмотреть, как программы построения деревьев распределят последовательности из этого таксона. Таким образом, были приняты следующие обозначения:
Архитектура:
- 1 - ATP_bind 4
- Ap - Apicomplexa
- Ki - Kinetoplastida
- Ne - Ecdysozoa (Nematoda)
- Ma - Chordata (только Mammalia)
- Ch - Chlorophyta
- 2 - ATP_bind 4 + Ribonuc_L-PSP
- Ot - Chordata (не Mammalia)
- 3 - ATP_bind 4 + Ribonuc_L-PSPx2
- Ar - Ecdysozoa (Arthropoda)
- Oo - Oomycetes
- St - Streptophyta
Fu - Fungi (встречаются все архитектуры). Например, очень много представителей со второй архитектурой из этого таксона.
Таким образом были переименованы все последовательности, выбранные в предыдущем практикуме.
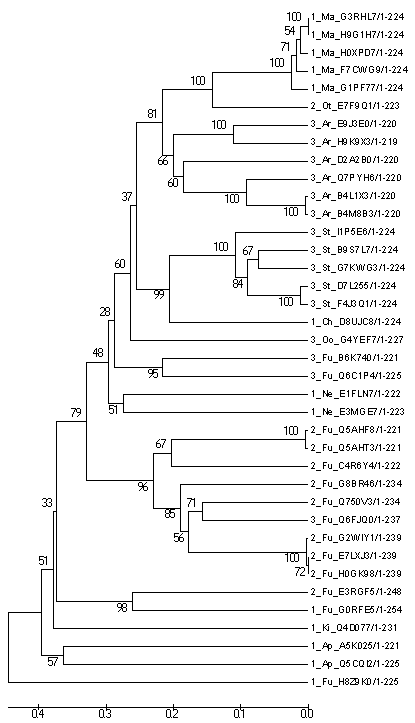
Затем по этим последовательностям было построено филогенетическое дерево. Это было сделано двумя принципиально отличающимися способами : UPGMA и neighbour joining. UPGMA предполагает молекулярные часы, реконструирует длины ветвей и, соответственно, строит укорененные деревья. Neighbour-joining не предполагает молекулярные часы и поэтому не укореняет дерево. Получившиеся деревья:
UPGMA:
Neighbour-joining
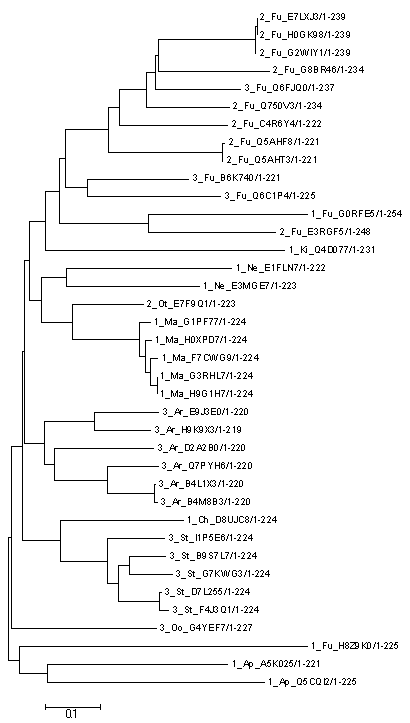
Видно, что в целом деревья похожи, не считая отдельных ветвей. Мне больше похожим на правду показалось второе, построенное методом neighbour-joining, он, например, поместил все грибы (исключая одну последовательность) в одну ветвь. Файл со скобочной формулой - pfam_tree.nwk.
Цветами выделены лучше всего представленные последовательностями таксоны: Chordata (красный), Fungi (синий), Ecdysozoa (зеленый), Viridiplantae (желтый).

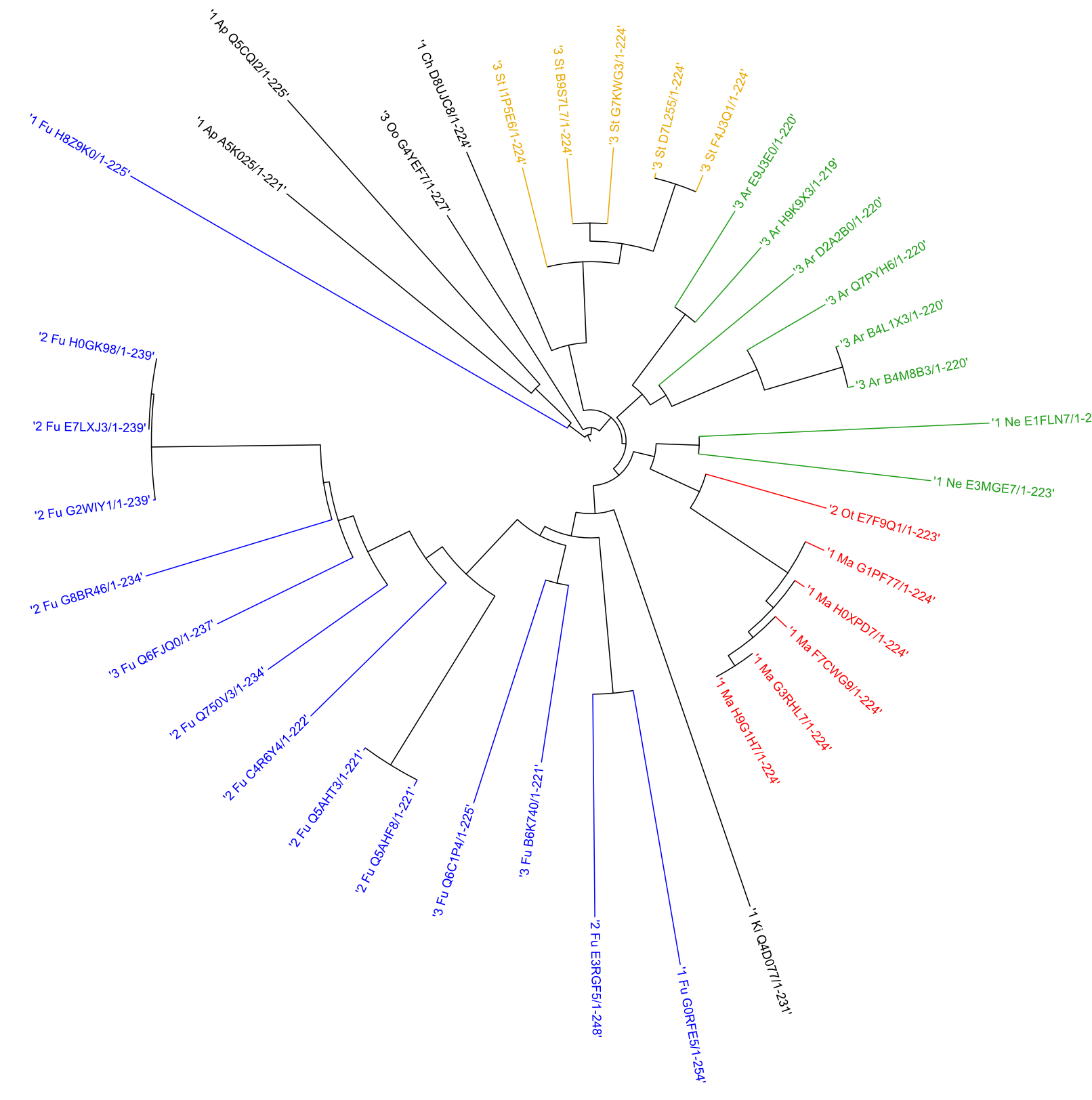
Видно, что одна ветвь грибов отходит отдельно от остальных. Может быть, это связано с тем, что у этой последовательности первая архитектура, а у остальных по большей части представлены вторая или третья (хотя представитель с первой тоже есть, и он входит в одну ветвь с другими). Также нематоды определяются ближе к хордовым, хотя по систематике они ближе к артроподам (ecdysozoa, зеленый цвет).
Определенные тенденции в эволюции домена проследить трудно. Конечно, видно, что, например, у млекопитающих осталась только первая архитектура, то есть у них АТР-связывающий домен не связан с рибонуклеазой. Аналогично у нематод. При этом у других хордовых или линяющих (что было видно еще из прошлого практикума при определении таксономии и наблюдается по дереву) встречаются и архитектуры с рибонуклеазой, то есть в какой-то момент у некоторых групп организмов производится отбор именно такой одиночной архитектуры. C другой стороны, есть и отбор в сторону третьей структуры (АТР-связывающий домен и две рибонуклеазы), например, у артропод и высших растений. У грибов же встречаются абсолютно все архитектуры, причем даже у одних и тех же семейств.