Реконструкция филогенетических деревьев
1.
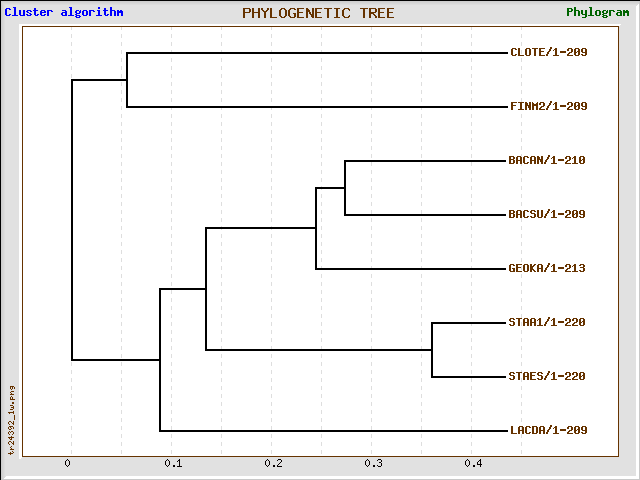
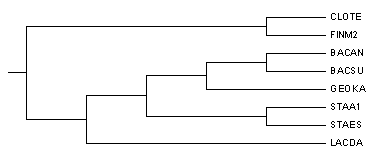
Дерево из предыдущего задания:
Распределение выбранных бактерий по таксонам (общая часть cellular organisms > Bacteria > Firmicutes)*:
| Clostridia | Clostridiales | Clostridiaceae | Clostridium | Clostridium tetani | |
| Clostridiales incertae sedis | Clostridiales Family XI. Incertae Sedis | Finegoldia | Finegoldia magna | ||
| Bacilli | Bacillales | Bacillaceae | Bacillus | Bacillus cereus group | Bacillus anthracis |
| Bacillus subtilis group | Bacillus subtilis | ||||
| Geobacillus | Geobacillus kaustophilus | ||||
| Staphylococcaceae | Staphylococcus | Staphylococcus aureus | |||
| Staphylococcus epidermidis | |||||
| Lactobacillales | Lactobacillaceae | Lactobacillus | Lactobacillus delbrueckii | ||
*таксоны расположены друг другом не всегда в соответствии с систематикой
Как видно, филогенетическое дерево абсолютно соответствует систематикe, что легко проследить по таблице.
2.
Выбран был рибосомный белок L3 (RL3).
Fasta-файл с белком RL3 всех выбранных бактерий. Выравнивание было получено при помощи muscle из web-service Jalview c параметрами по умолчанию.
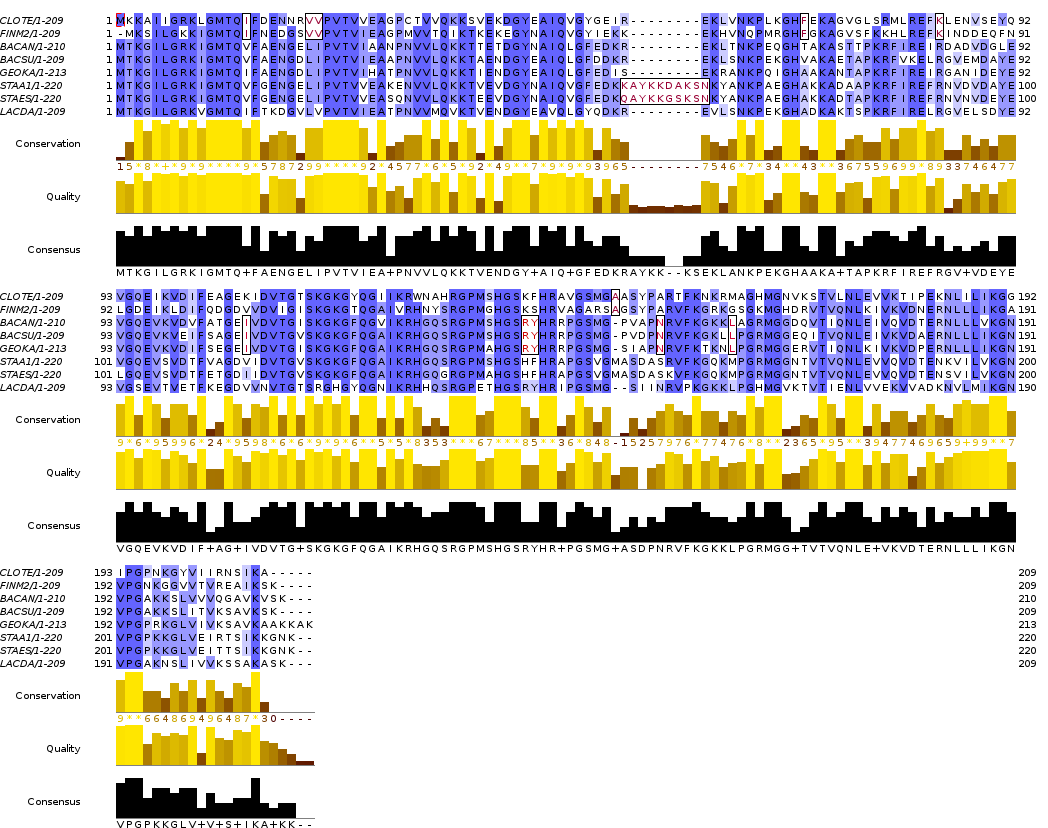
Fasta-файл с выравниванием и jar-файл.
3.
Я попыталась найти диагностические позиции выравнивания. Все упомянутые остатки выделены на рисунках выше.
Замена 22 и 23 остатков у CLOTE и FINM2 на валин, 15 на изолейцин, 69 на фенилаланин, 84 на лизин, 148 на аланин может служить признаком систематической группы Clostridia или Clostridiales. Различающиеся остатки встречаются, как видно, единично, зато совпадают у обоих видов этих таксонов. Эти два вида наиболее "далеки" от остальных.
Сразу бросается в глаза, что остатки 57-66 STAA1 и STAES не выравниваются ни с чем другим и принадлежат белку только этих бактерий, причем между собой эти участки достаточно похожи. Это можно считать признаком Staphylococcaceae или Staphylococcus. Им близок вид LACDA, но по выравниванию этого сразу сказать нельзя, белок из этой бактерии достаточно плохо выравнивается со всеми остальными.
Виды GEOKA, BACAN и BACSU (Bacillali > Bacillales > Bacillaceae) объединяют участки 107 (изолейцин), 138-139 (аргинин и тирозин), 152 (аспарагин) и 160 (лейцин).
4.
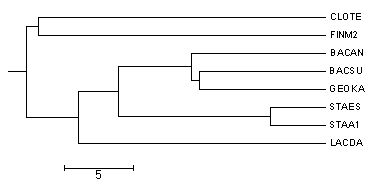 |
 |
 |
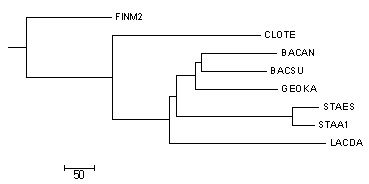 |
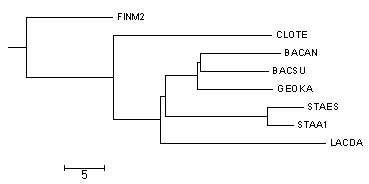
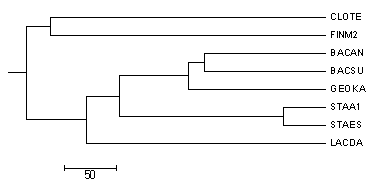
2 и 4 деревья (neighbour join), которые учитывают расстояния, совпадают с правильным (укоренение этот метод производит случайным способом, и если укоренить дерево в другом месте, получится в точности нужная картинка). 3 дерево тоже совпадает с правильным, а в 1 наблюдается различие в ветви ((BACSU,GEOKA),BACAN).
5.
Полученное дерево различается с правильным. LACDA почему-то переместился и нарисовался ближе к BACSU, чем даже BACAN, что странно, ведь ни по логике, ни по выравниванию белка вроде бы так быть не может.
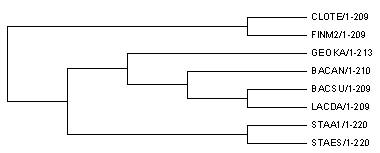
Сохранились ветви {CLOTE,FINM2} vs все остальное и {STAA1,STAES} vs все остальное. Если в правильном дереве есть ветви {BACSU,BACAN}, {BACSU,BACAN,GEOKA} и {BACAN, BACSU, GEOKA, STAA1, STAES} vs {CLOTE,FINM2,LACDA}, то в построенном появилась ветви {BACSU,LACDA}, {BACSU,LACDA,BACAN} и {GEOKA, BACAN, BACSU, LACDA} vs {CLOTE, FINM2, STAA1, STAES}.
6.
К сожалению, не успела поближе ознакомиться с этим сервисом, но сразу бросилось в глаза то, что нельзя прикрепить файл с выравниванием, а надо обязательно вбивать его в текстовое поле (или я просто не нашла?). Хорошо, что на выходе достаточно много информации, а не просто картинка, и во вполне удобном виде. Если надо скопировать формулу или матрицу, то действительно их можно просто взять и скопировать. Да и итоговое дерево совпало с правильным: