Алгоритмы реконструкции деревьев
1. Укоренение в среднюю точку
Деревья, построенные с помощью UPGMA, бессмысленно укоренять в среднюю точку, потому что этот метод не взвешивает длины ветвей. C помощью retree было построено укорененное изображение дерева, построенного с помощью neighbour join using % identity в 4 упражнении предыдущего задания (дерево).
Оригинальное изображение:
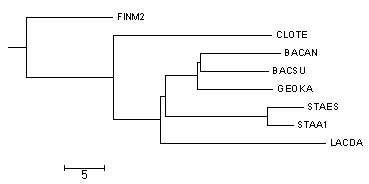
Новое дерево, укорененное в среднюю точку:
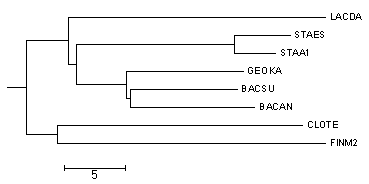
Как видно, укоренение и вообще все дерево полностью совпадает с правильным. Этого следовало ожидать, так как укореняется действительно самая длинная ветвь, а дерево правда было правильным изначально, что мы видели еще в прошлом задании. Картинка правильного дерева:
2. Использование внешней группы
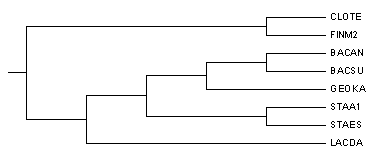
Дерево, построенное с помощью MP, укоренять в среднюю точку бесполезно (Maximum parsimonyб= не реконструирует длины ветвей), поэтому был использован метод внешней группы. Вначале я добавила в файл с невыровненными последовательностями белок RL3 из E.coli и выровняла все с помощью muscle, получив выравнивание. Затем с помощью MEGA я получила дерево этих белков методом максимальной экономии и укоренила его с помощью внешней группы, роль которой выполнял белок из ECOLI. Полученное дерево:
Видно, что дерево построилось как-то совсем иначе, чем предыдущие. FINM2 теперь как будто бы отделяется раньше, чем остальные (укоренение не там, где надо), а не образует нетривиальную ветвь с CLOTE (как правильно); в ветви ((BACAN,GEOKA),BACSU) тоже ошибка.
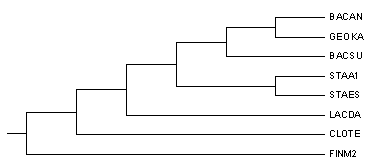
3. Бутстрэп
В качестве метода было выбрано все то же neighbour join. Число реплик для бутстрэпа - 100.
Original tree:
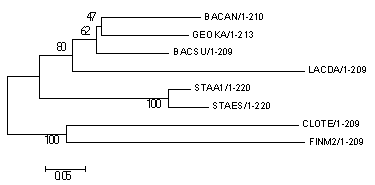
Bootstrap consensus tree:
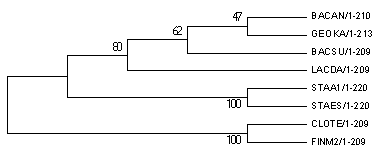
Как видно, деревья одинаковые и оба неправильные. Предположение, что неправильные ветви [(BACAN,GEOKA); ((BACAN,GEOKA),BACSU); (((BACAN,GEOKA),BACSU),LACDA)] имеют меньшую поддержку (47, 62 и 80, соответственно), чем правильные, подтвердилось. Чем ближе к листьям дерева, тем сильнее становится "неправиьлность", а поддержка, соответственно, все меньше.