Задание d1: определение вторичной структуры
Для работы была выбрана структура 1QD9 (RIDA_BACSU, рис. 1), качество которой было проанализировано ранее. В pdb-файле уже размечены элементы вторичной структуры:
HELIX 1 1 ILE A 46 ALA A 64 1 19 HELIX 2 2 GLN A 83 PHE A 95 1 13 HELIX 3 3 ILE B 46 GLY B 65 1 20 HELIX 4 4 GLN B 83 PHE B 95 1 13 HELIX 5 5 ILE C 46 ALA C 64 1 19 HELIX 6 6 GLN C 83 PHE C 95 1 13 SHEET 1 A 6 LYS A 3 VAL A 5 0 SHEET 2 A 6 GLN A 19 VAL A 23 -1 O GLY A 20 N VAL A 5 SHEET 3 A 6 MET A 26 SER A 30 -1 N MET A 26 O VAL A 23 SHEET 4 A 6 LEU A 115 VAL A 124 -1 O VAL A 120 N SER A 29 SHEET 5 A 6 VAL A 72 ALA A 79 -1 N VAL A 72 O ILE A 121 SHEET 6 A 6 ALA A 101 GLU A 106 1 N ALA A 101 O VAL A 72 SHEET 1 B 6 LYS B 3 VAL B 5 0 SHEET 2 B 6 GLN B 19 VAL B 23 -1 O GLY B 20 N VAL B 5 SHEET 3 B 6 MET B 26 SER B 30 -1 N MET B 26 O VAL B 23 SHEET 4 B 6 LEU B 115 VAL B 124 -1 O VAL B 120 N SER B 29 SHEET 5 B 6 VAL B 72 ALA B 79 -1 N VAL B 72 O ILE B 121 SHEET 6 B 6 ALA B 101 GLU B 106 1 N ALA B 101 O VAL B 72 SHEET 1 C 6 LYS C 3 VAL C 5 0 SHEET 2 C 6 GLN C 19 VAL C 23 -1 O GLY C 20 N VAL C 5 SHEET 3 C 6 MET C 26 SER C 30 -1 N MET C 26 O VAL C 23 SHEET 4 C 6 LEU C 115 VAL C 124 -1 O VAL C 120 N SER C 29 SHEET 5 C 6 VAL C 72 ALA C 79 -1 N VAL C 72 O ILE C 121 SHEET 6 C 6 ALA C 101 GLU C 106 1 N ALA C 101 O VAL C 72
В структуре, согласно этой разметке, есть шесть альфа-спиралей (по две на каждую цепь, белок является гомотримером) и три бета-листа (по одному на каждую цепь).
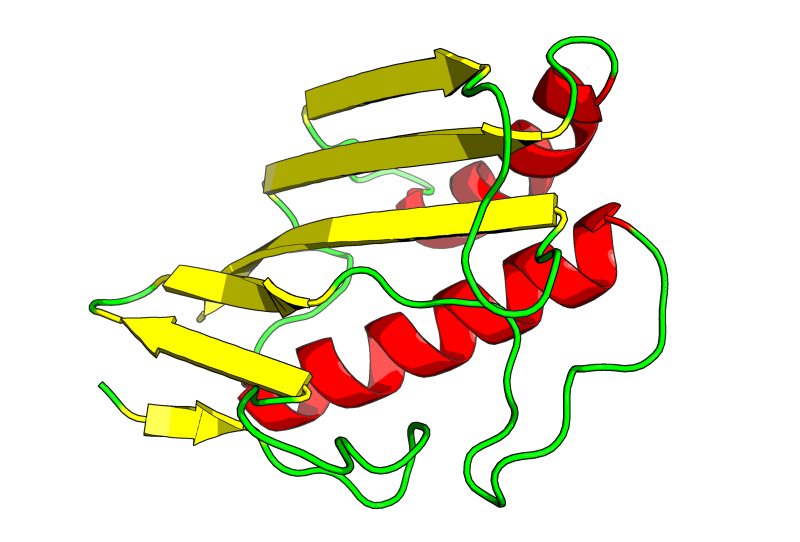
Рис. 1: Структура 1QD9 (цепь А). Красным отмечены альфа-спирали, желтым - бета-лист, зеленым - петли между ними.
1. DSSP
Результат работы алгоритма представлен в файле 1qd9.dssp. В нем мы видим
результат расчета доступной поверхности белка (можно отключить),
разметку по элементам вторичной структуры, образующие их водороднык
связи для всех остатков, значения некоторых дихедралов и т. д.
Всего программой было найдено шесть α-спиралей (по две на каждую цепь). Разметка спиралей немного отличается в различных
цепях (что странно), но в целом они похожи и образованы остатками 45-62 и 84-93 (так определилась спираль для цепи А). Эти значения совпадают с разметкой в pdb-файле с точностью до пары остатков, такой же разброс для предсказания DSSP одной и той же спирали для разных цепей. Таким образом, альфа-спирали определились довольно точно.
Аналогичная ситуация с бета-листом. Он был найден для каждой цепи, принадлежащие
ему остатки тоже были определены достаточно точно (2-5, 20-23, 26-29, 71-78, 101-106, 116-123 для цепи А).
Кроме того, в структуре были найдены искривленные участки (bends), повороты полипептидной цепи. Найденные изолированные бета-тяжи (В) представлены одиночными остатками и не очень понятно, почему они вообще выделены в тяжи.
Более того, было найдено достаточно много нетипичных спиралей 310 (G). В каждой цепи таких было найдено три, все длиной по три остатка (68-70, 81-83, 111-113). Пример такой "спирали" представлен на рис. 2. Мне кажется, что вряд ли это можно назвать спиралью, скорее, это просто поворот цепи. Кроме того, эти участки отличаются достаточно высоким по сравнению с остальной структурой B-фактором, так что утверждать наличие таких нестандартных спиралей нельзя. Видимо, таким образом DSSP определяет бета-повороты, которые по своим параметрам
сходны со спиралями 310.

Рис. 2: "Нетипичная" спираль, определенная DSSP.
2. SheeP
Структура 1QD9 была загружена на веб-сервис для построения карт бета-листов SheeP. Сервис нашел в стректуре всего пять бета-листов. Из них два идентичных, найденных в цепях А и С, очень маленькие (две цепи по три аминокислотных остатка) и, кроме того, по большей части помечены как гэпы. Поэтому вряд ли их можно считать полноценными бета-листами.
Оставшиеся три бета-листа абсолютно идентичны и находятся в трех одинаковых цепях белка. В дальнейшем будут приведены данные для бета-листа в цепи А.
Этот бета-лист состоит из шести достаточно длинных (от 4 аминокислот в бета-листе) бета-тяжей и одного короткого, в две аминокислоты. Карта этого бета-листа приведена на рис. 3.
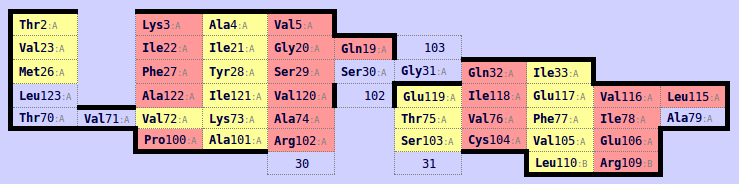
Рис. 3: Карта одного из бета-листов структуры, полученная при помощь SheeP.
По карте видно, что в бета-листе есть гэпы в его середине, обозначающие вставки в тяжах. На рис. 4 показано расположение этого бета-листа во всей структуре. Видно, что одна его сторона обращена в гидрофильную полость между мономерами белка (на рисунке отмечены серым, на карте бета-листа - желтым), а вторая - к гидрофобному ядру мономера (отмечены красным на обоих рисунках). Однако при этом нельзя сказать, что одна группа остатков гидрофильна, а вторая - гидрофобна; в обеих группа остатки с различными свойствами чередуются.
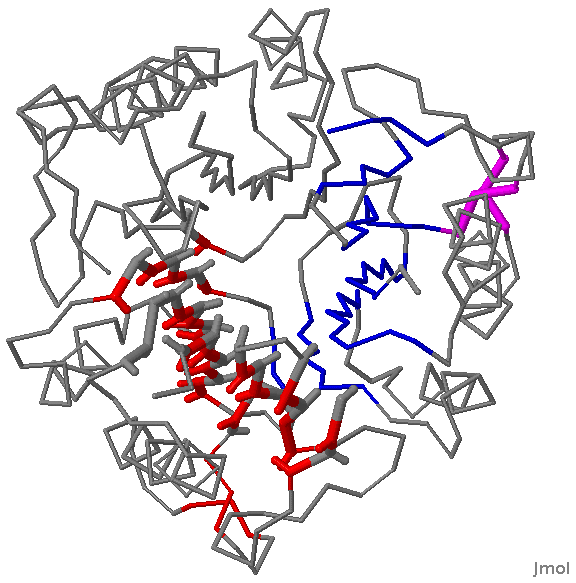
Рис. 4: Структура 1QD9 с показанным бета-листом цепи А (жирные линии). Серым отмечена сторона белка, обращенная в гидрофильную полость между мономерами, красным - в гидрофобное ядро.
На карте бета-листа видно нарушение в периодичности направленности боковых радикалов (Val71-72). На самом деле в этом участке структуры уже вряд ли продолжается бета-тяж; конфигурация цепи больше похожа на петлю. На карте бета-листа этот участок не покрашен в какой-либо цвет (гэпы).
На рис. 5 цветом показан один "хребет" бета-листа, показанный на карте (остатки 5, 20, 29, 120, 74, 102). Как видно, эти остатки действительно расположены в одном хребте и, судя по расстояниям между соответствующими атомами О и N, связаны водородными связями.
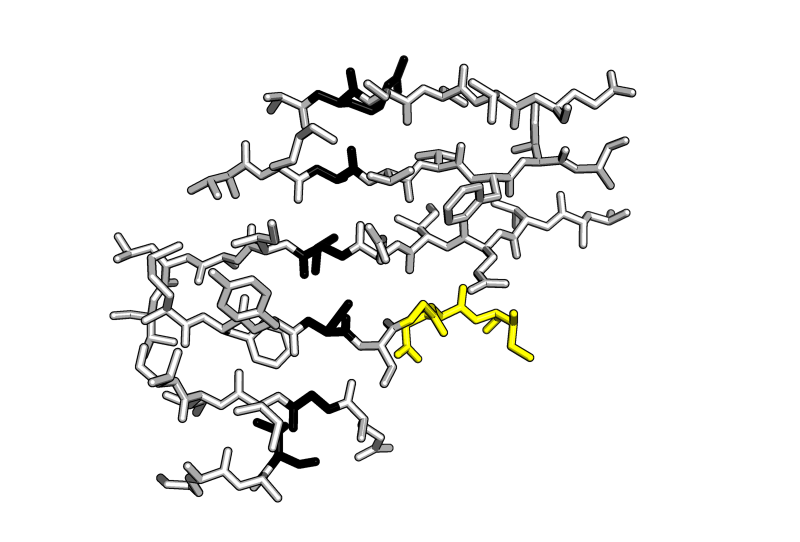
Рис. 5: Бета-лист цепи А структуры 1QD9. Черным отмечен один из "хребтов" бета-листа (пояснение в тексте), желтым - остатки 31-33, причисленные SheeP к бета-тяжу.
Предсказание бета-листа с помощью SheeP несколько отличается от предсказания DSSP и разметки в pdb-файле. В результате SheeP присутствует упомянутый выше маленький бета-тяж 109-110, кроме того, один из тяжей на целых три аминокислоты длиннее (31-33). Они показаны желтым на рис. 5. Трудно сказать, принадлежат ли на самом деле эти остатки бета-тяжу или уже образуют начало петли. Я бы причислила их к тяжу, так как остатки 32 и 33 образуют водородные связи с остаткками 118 и 117 соседнего тяжа. Присутствуют еще различия, но в пределах одной крайней аминокислоты бета-тяжа.