d2. Совмещение структур
1.
С помощью сервиса PDBeFold были найдены структурные гомологи белка RIDA_BACSU (YABJ_BACSU) - структуры 1QD9. Поиск проводился по всем цепям. Было отобрано четыре не слишком близких, но и не слишком далеких гомолога с RMSD от 0.62 до 1.21 ангстрема. Для этих структур было полученно попарное и множественное выравнивания последовательностей по совмещению структур. Также привожу выравнивание элементов вторичной структуры белков:
PDB 1qd9:A s s s H S H S H S PDB 3quw:A s s s H S H S H S PDB 2csl:B s s s H S H S H S PDB 3l7q:I - s s H S H S H S PDB 2uyk:B s s s H S H S H
По выравниванию видно, что вторичная структура белков хорошо накладывается друг на друга.
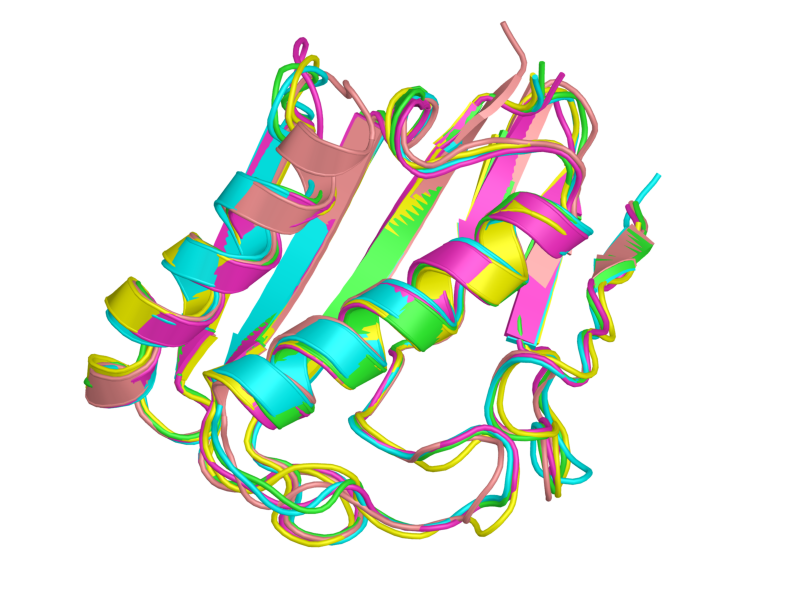
Рис. 1: совмещение структур 1QD9 и ее четырех гомологов.
Из совмещения структур видно, что структуры гомологов действительно похожи между собой. Выделяется только
структура, показанная розовым (2uyk), у которой одна альфа-спираль длиннее, чем у других структур.
C помощью Jalview было построено множественное выравнивание этих последовательностей и сравнено
с выравниванием по структуре. В целом оба выравнивания совпали, значительное различие было только
на одном участке (рис. 2).
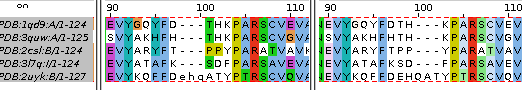
Рис. 2: участок выравниваний по последовательности (слева) и по структуре (справа).
В структурах этот участок соответствует бета-петле между двумя альфа-спиралями. Здесь структура
2uyk отличается от остальных и по структуре, и по последовательности.
В выравнивании по последовательности ее остатки Glu96 и His97 выровнены с остатками других последовательностей,
в другом выравнивании напротив них стоят гэпы (рис. 3).
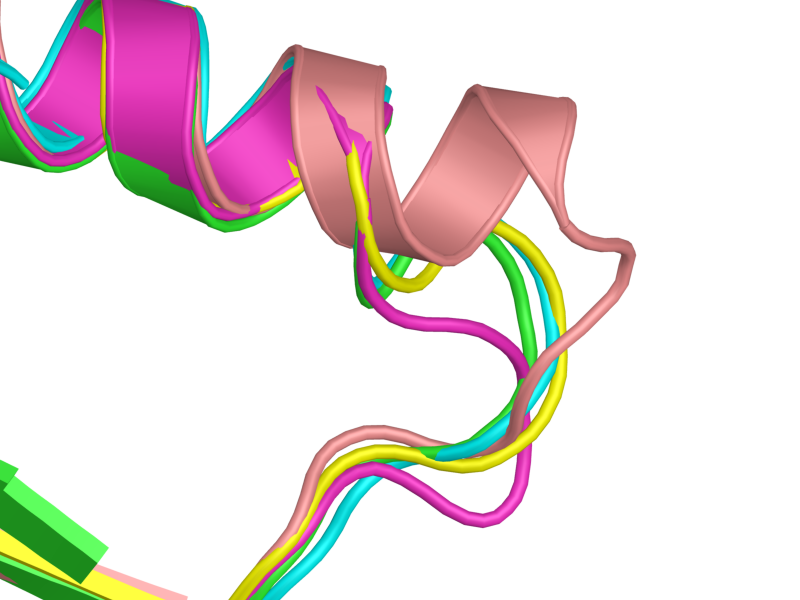
Рис. 3: плохо выровненный участок структур.
Сложно сказать, какое из выравниваний право. Видно, что в последовательности 2uyk есть
вставка из нескольких аминокислотных остатков, которая продлила альфа-спираль и
изменила конформацию петли. Она вряд ли может с чем-то выровниться как по структуре, так
и по последовательности, так что то, что в разных выравниваниях гэпы немного смещены, ничего не значит.
2.
Для работы была выбрана структура 1kgc. Координаты доменов: 118-206 цепи D - альфа-цепь, 119-247 цепи E - бета-цепь. На рис. 4 приведена эта структура
с обозначенными цветом альфа и бета цепями.
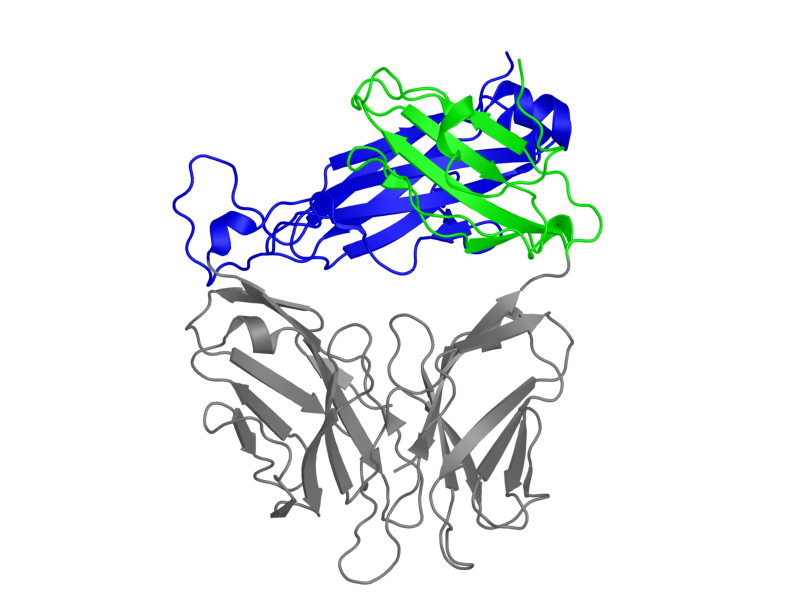
Рис. 4: структура 1kgc. Зеленым показана альфа-цепь,
синим - бета-цепь.
В альфа-цепи был найден всего один бета-лист, в бета-цепи - два. Эти бета-листы похожи и все
три содержат в себе остаток цистеина.
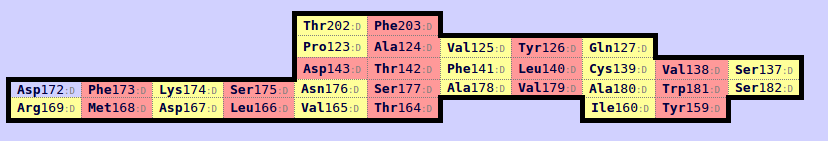
Рис. 5: Карта бета-листа альфа-цепи.

Рис. 6: Карты бета-листов бета-цепи.
Из карт бета-листов и консервативности некоторых остатков (например, Val и Leu около консервативного цистеина 139 в альфа-цепи и 148 в бета-цепи) я предположила, что бета-листу
альфа-цепи соответствует первый лист бета-цепи.
Для выравнивания pair_fit я брала по пять остатков из двух тяжей, один из которых
содержал в себе консервативный цистеин.
pair_fit d2al///137-141/CA, d2be///146-150/CA
RMSD составил 0.226 А - довольно неплохое наложение. По совмещению бета-листов видно,
что они накладываются друг на друга достаточно хорошо. У листа альфа-цепи есть
еще один бета-тяж, который не имеет соответствия в бета-цепи, и центральный тяж намного длиннее,
однако четыре бета-тяжа (два длинных и два коротких) накладываются друг на друга очень хорошо.
Топология этих листов сильно отличается - тяжи соединены не в том порядке, однако сонаправлены.
Соответственно, петли между цепями не накладываются друг на друга. Исключение составляют петли
из одного из коротких тяжей (на рис. 7 сверху) - они практически совпадают.
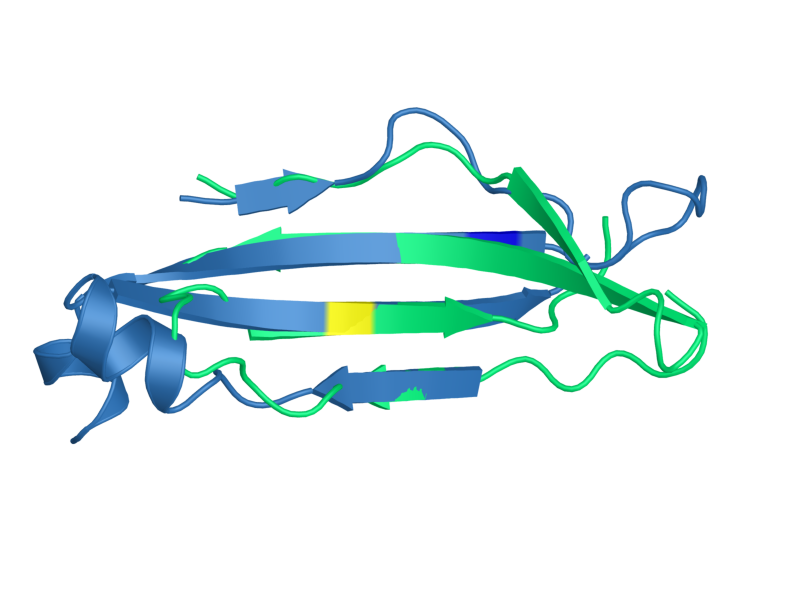
Рис. 7: Совмещение бета-листов альфа- и бета-цепей. Желтым
показан консервативный остаток цистеина.
Файл с совмещением