Интерпретация особенностей РСА
Поиск ошибок в PDB файлах
7GPB
- гликоген фосфорилаза кролика
- 1990 год
- разрешение – 2.9 Å
Общий вид структуры:
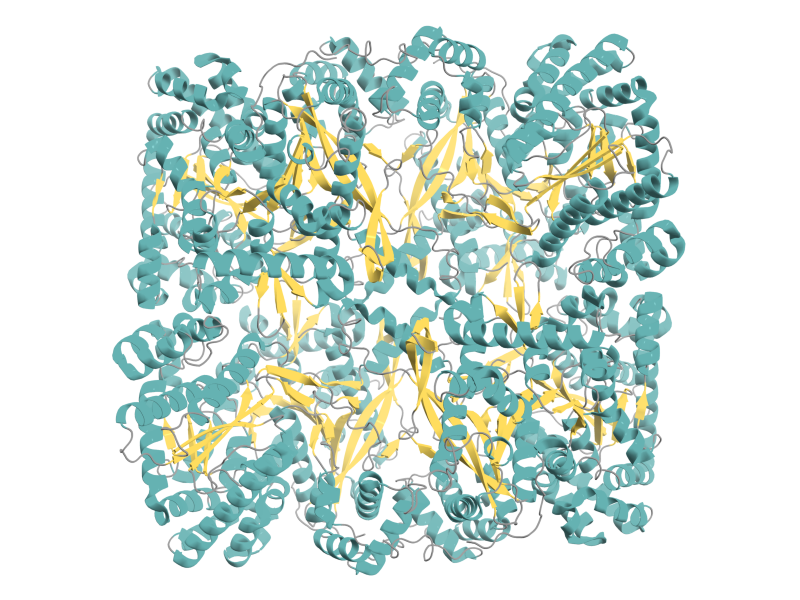
Рис. 1: Общий вид структуры
Белок представляет собой гомотетрамер.
PDB файл выглядит странно: во всех полях, соответствующим информации об эксперименте, не написано ничего (точнее, NULL). Нет ни значения Rfree,
ни данные об условиях эксперимента. В REMARK отмечено лишь о нескольких отсутствующих в структуре атомах и о неправильном расположении сульфата. В протоколе WHAT_IF PDB
много замечаний по RMS между цепями, странных длинах и углах связей, плохом качестве карты Рамачандрана, конформациях главной цепи и еще много о чем.
При осмотре нескольких остатков триптофанов структуры сразу стало понятно, в чем "странность" этого файла. У нормального триптофана ароматические кольца расположены в одной плоскости, что обусловлено единой электронной плотностью этих колец. Это позволяет ему вступать в стекинг-взаимодействия с другими подобными молекулами. На рис. 2 показано, как выглядит триптофан обычно:
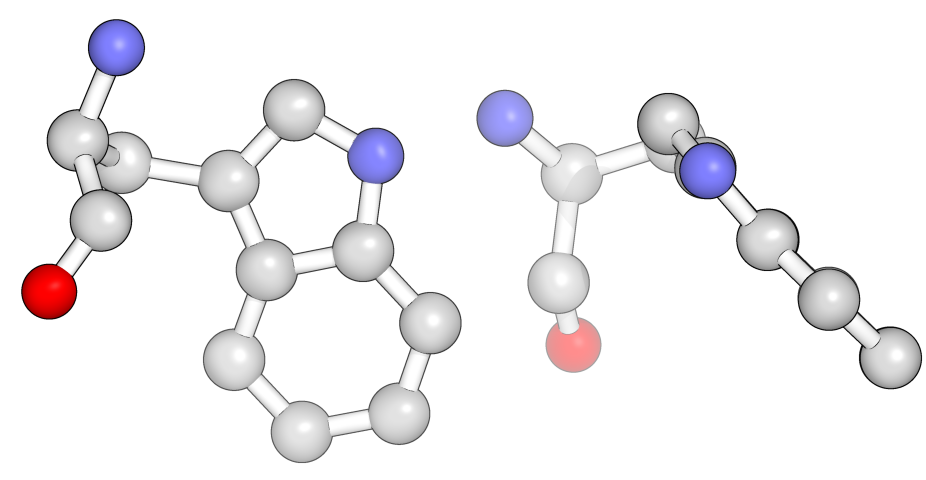
Рис. 2: Вид "нормального" остатка триптофана с двух сторон
В некоторых же остатках триптофана данного белка кольца расположены не в одной плоскости — разница заметна невооруженным взглядом. На рис. 3 и 4 показано два таких остатка (в структуре их еще больше):
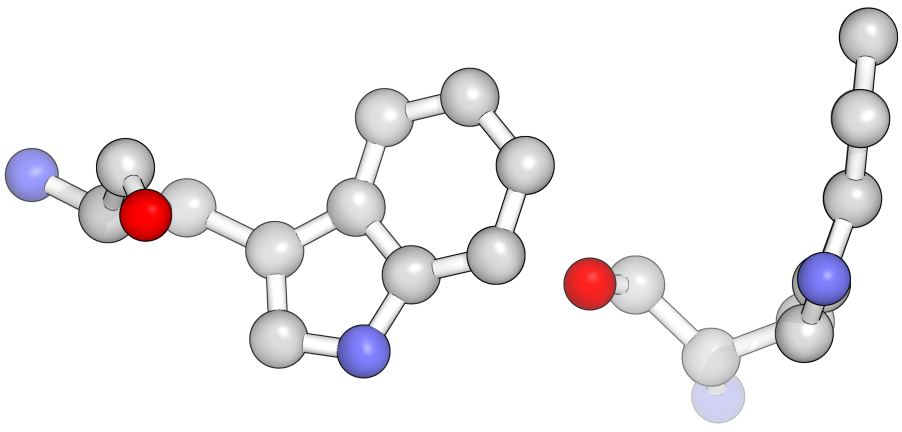
Рис. 3: Остаток Trp67 цепи С структуры
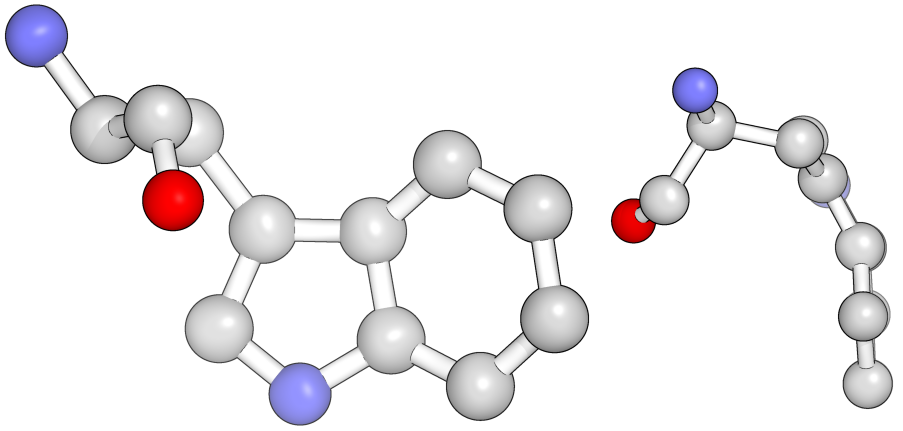
Рис. 4: Остаток Trp67 цепи А структуры
Кроме того, есть совершенно выдающийся случай:
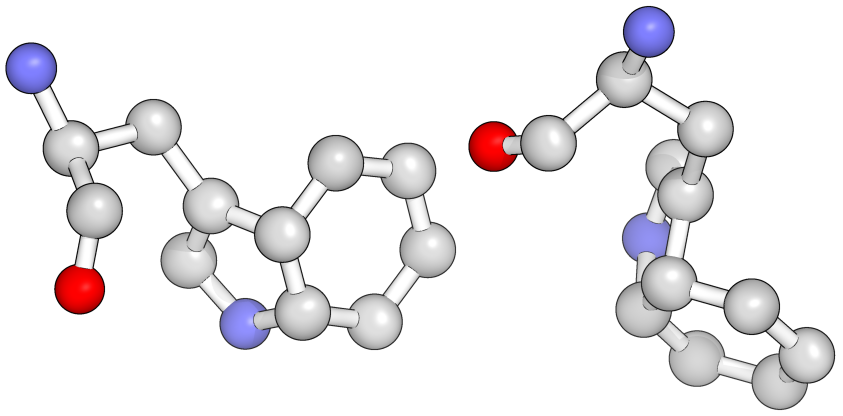
Рис. 5: Остаток Trp67 цепи D структуры
В EPS и PDB нет файла со структурными факторами для этой структуры, так что посмотреть карту электронной плотности и попробовать объяснить происходящее невозможно.
В WHAT_IF протоколе есть свидетельства об этих ошибках структуры, а именно нарушении планарности боковых радикалов и нарушении плоской структуры ароматических колец. Для каждого "плохого" радикала была подсчитана сумма квадратов отклонений от предполагаемой плоскости и приведено в соответствующих таблицах. Как видно по ним, подобные проблемы есть у остатков аспарагина, аспарагиновой кислоты, фенилаланина и глутамата.
Планарность: 2527 TRP ( 67-) D 36.29 58 TRP ( 67-) A 12.99 1170 TRP ( 361-) B 10.86 1129 ASP ( 320-) B 8.53 1704 TRP ( 67-) C 7.28 1606 TRP ( 797-) B 7.07 1300 TRP ( 491-) B 7.06 2820 TRP ( 365-) D 6.17 1196 TRP ( 387-) B 5.98 3252 TRP ( 797-) D 5.96 180 TRP ( 189-) A 5.80 268 ARG ( 277-) A 5.52 1997 TRP ( 365-) C 5.35 2019 TRP ( 387-) C 5.26 1881 TRP ( 244-) C 5.15 1922 GLU ( 290-) C 5.00 1826 TRP ( 189-) C 4.98 881 TRP ( 67-) B 4.96 526 GLU ( 540-) A 4.89 1632 GLU ( 823-) B 4.68 3156 GLU ( 701-) D 4.64 1099 GLU ( 290-) B 4.61 996 TRP ( 182-) B 4.44 88 ASN ( 97-) A 4.37 538 GLU ( 552-) A 4.36 373 TRP ( 387-) A 4.26 477 TRP ( 491-) A 4.05 Ароматика: õ2 TYR ( 820-) C OH 5.10 866 TYR ( 52-) B CB 4.99 2452 TYR ( 820-) C CB 4.75 1071 PHE ( 257-) B CB 4.53 2544 TYR ( 84-) D OH 4.32 1620 PHE ( 811-) B CB 4.28 866 TYR ( 52-) B OH 4.26 865 TYR ( 51-) B OH 4.17 104 TYR ( 113-) A CB 4.14 2512 TYR ( 52-) D OH 4.09 694 PHE ( 708-) A CB 4.06
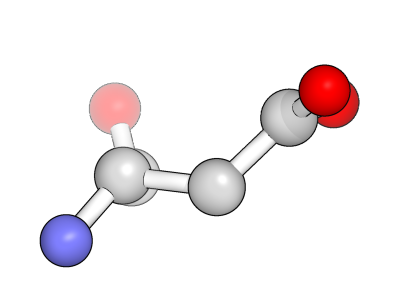
Рис. 6: Остаток Asp320 цепи B
Вывод можно сделать один: это очень плохая структура с множеством недостатков, и верить ей не надо.
5RXN
- рубредоксин
- 1984 год
- 1.2 Å
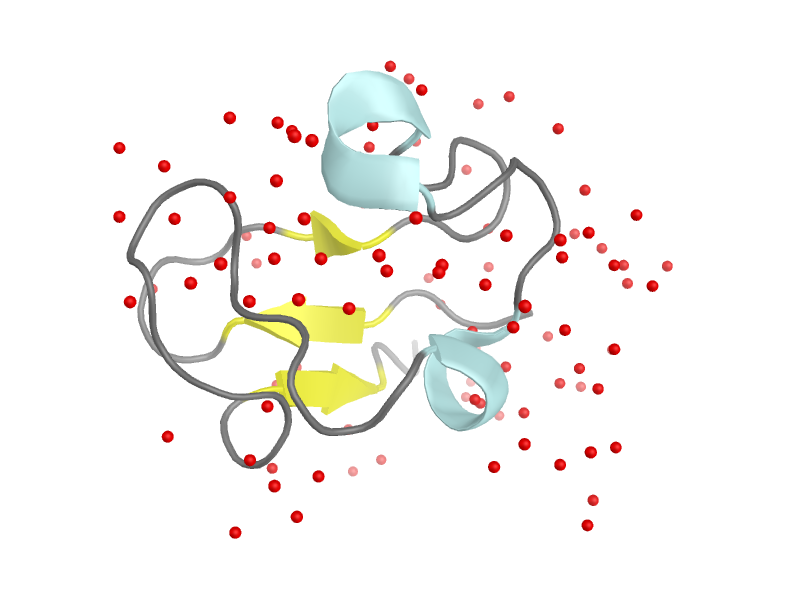
Рис. 7: Общий вид белка
Это структура маленького (54 аминокислотных остатка) белка клостридии с высоким разрешением, настолько, что в структуре есть даже атомы водорода. Но даже в таком маленьком белке есть ошибки. Во всем белке всего три остатка треонина, однако только два из них имеют правильную конфигурацию. У треонина есть два хиральных центра. Один из них - СА, как и у большинства аминокислот, второй - атом СВ. В живой природе встречается только (2S, 3R)-стереоизомер.
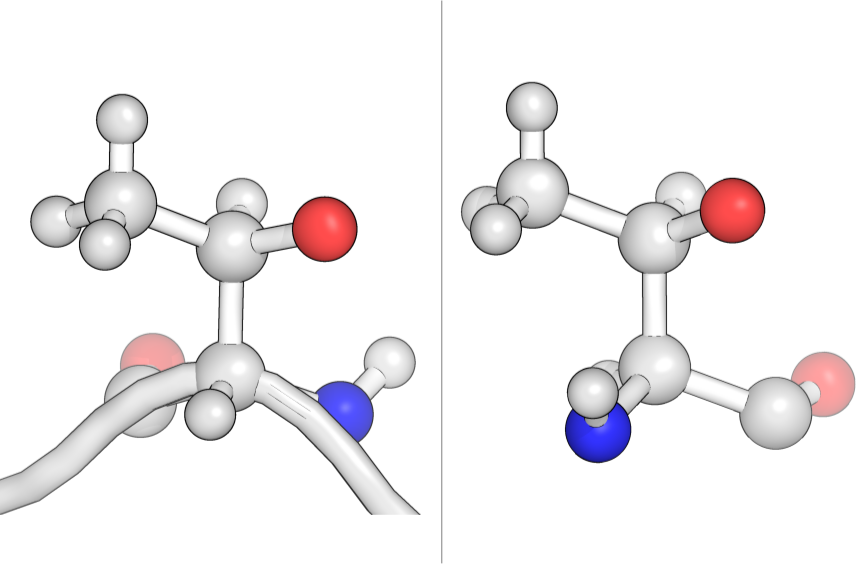
Рис. 8: Thr28 и Thr7 - "нормальные" треонины
Третий же треонин имеет (2S, 3S)-конформацию. Он не мог бы встретиться в живом белке.
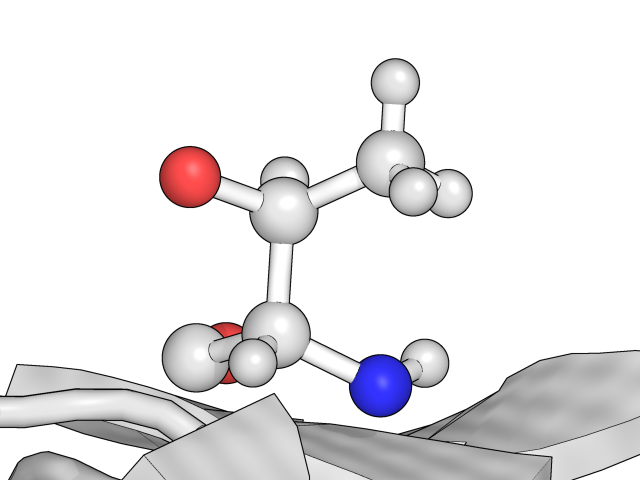
Рис. 9: Thr5
В протоколе WHAT_IF также упомянут этот остаток в связи с тем, что атомы OG и CG "поменялись местами". На рис. 10 показан тот же остаток с наложенной восстановленной картой электронной плотности (уровень подрезки = 1 σ). На самом деле на этой карте совершенно не видны водороды и непонятно, исходя из чего они были восстановлены (на меньших уровнях подрезки их тоже нет, зато есть, само собой, много шума). Если при восстановлении функции электронной плотности пик Сγ оказался вдруг больше Оγ, могла произойти замена этих атомов местами, что и влечет за собой появление неправильного стереоизомера. Кроме того, не знаю, какая в 1984 году была ситуация с автоматизированным анализом конформаций и углов в молекуле, так что эту ошибку можно было пропустить.
| Рис. 10: Thr5 и карта электронной плотности (1 σ) |
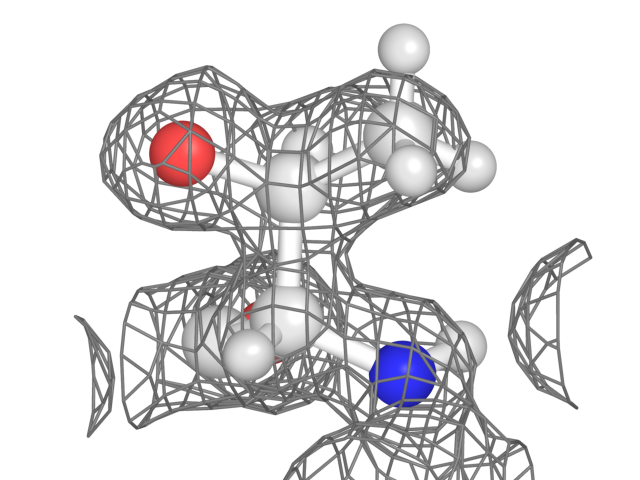 |
Также хиральностью обладает боковой радикал изолейцина, однако, в этой структуре никаких нарушений найдено не было. Наверное, найти в реальной PDB-структуре неправильный изолейцин гораздо труднее, потому что для этого недостаточно просто случайно поменять два атома местами.
1GT0
- транскрипционные факторы, связанные с ДНК
- 2003 год
- 2.60 Å
Структура представляет собой двойную спираль ДНК и два связанных с ней белка.
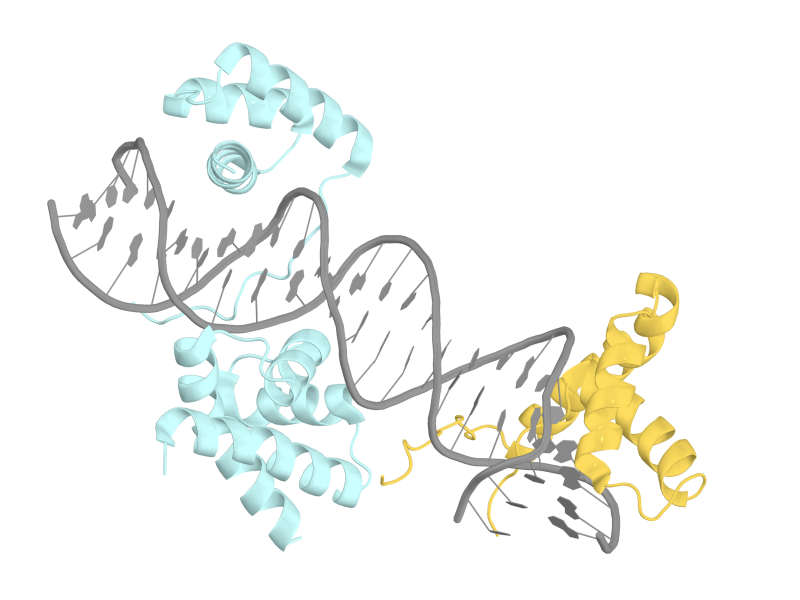
Рис. 10: Общий вид структуры
Участок одного из белков (цепь С) с 78 по 96 остатки отсутствует. При этом "приграничные" остатки имеют очень высокий B-фактор (до 97.7), то есть их расположение тоже не известно точно.
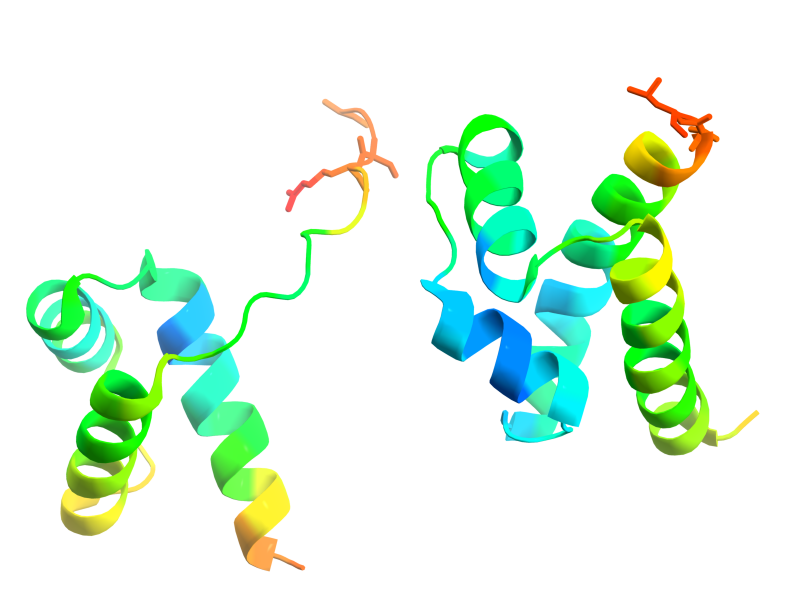
Рис. 11: участок цепи С с отсутствующими остатками; покраска по В-фактору
Наиболее вероятное объяснение отсутствия части цепи таково: остатки с 78 по 96 имели слишком большой В-фактор (больше 100), то есть их расположение в различных ячейках было настолько различным, что дать какую-то представительную картину нельзя. Действительно, этот участок похож на лабильную петлю между двумя альфа-спиралями, ее конформации вправду могут сильно отличаться в разных кристаллографических ячейках. Авторы структуры решили не приводить модель для слишком подвижного участка. Чтобы проверить эту гипотезу, была построена карта электронной плотности для этой области (уровень подрезки = 1 σ).
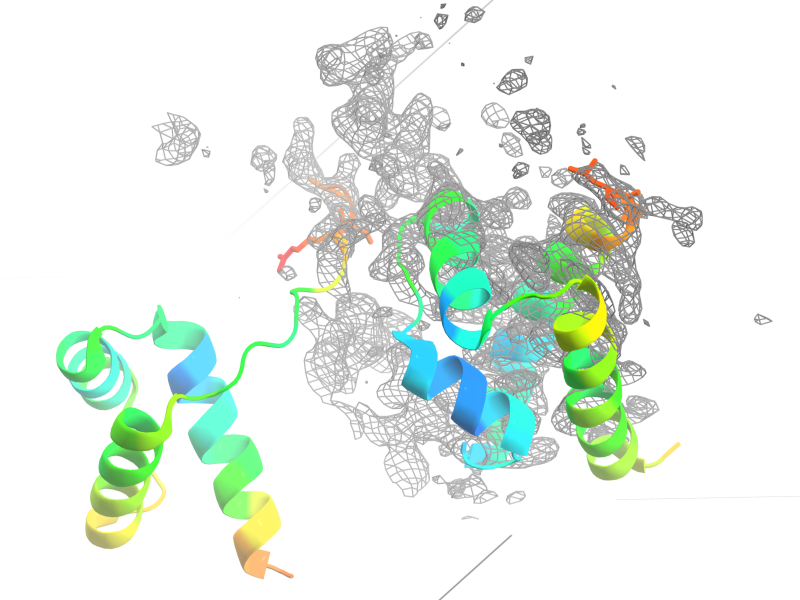
Рис. 12: Участок цепи С с отсутствующими остатками с показанной картой электронной плотности (1 σ), покраска по В-фактору
Если посмотреть на остатки 75-77 и 97-100, видно, что для них электронная плотность тоже не вполне соответствует предсказанной модели. Если общий ход полипептидной цепи еще различим, то длинные боковые остатки сильно размазаны (что соответствует высокому значению В-фактора).
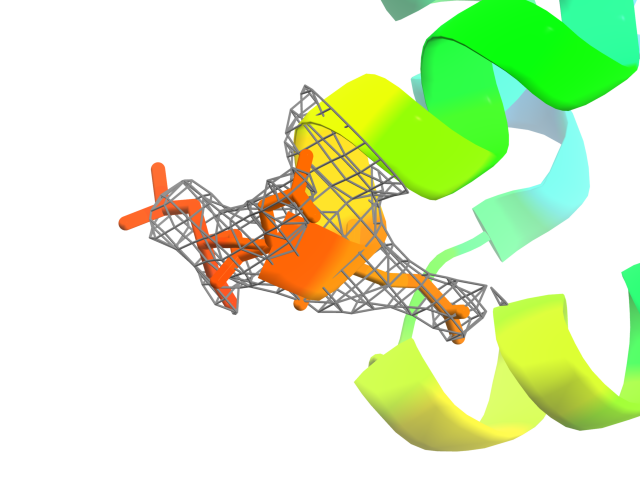
Рис. 13: Карта электронной плотности остатков 75-77
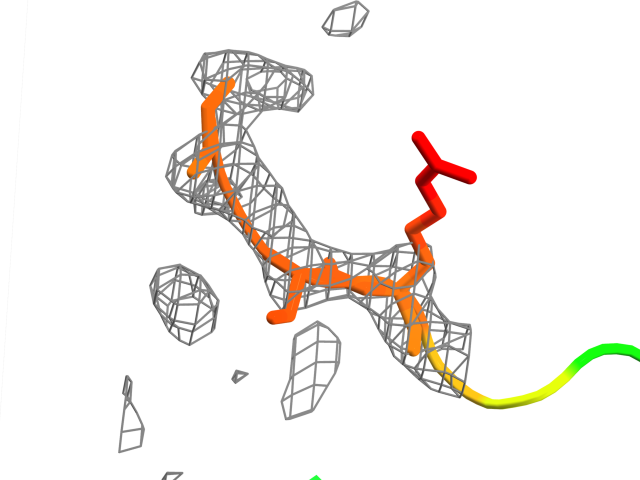
Рис. 14: Карта электронной плотности остатков 97-100
Сравнение значений коэффициента заполнения и температурного фактора в струтурах разного разрешения
Для работы были выбраны структуры 41GS (разрешение - 0.85 Å) и 4RF1 (2.15 Å). Обе структуры представляют собой белки с примерно равным количеством атомов (2787 и 3021, соответственно). Учитывались только атомы белков, без растворителя.
Температурные факторы
Из соответствующих PDB-файлов были получены значения температурных факторов для всех атомов и представлены в виде гистограммы (рис. 15). На рисунке розовым показаны значения для структуры с высоким разрешением (H), голубым - с низким (L). Средние значения показаны пунктиром, медианные видны из графиков. Ширина столбца - 5.
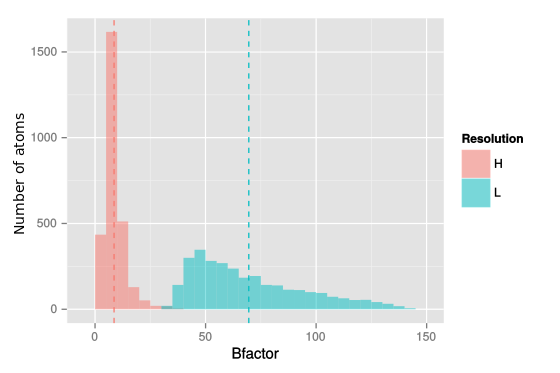
Рис. 15: Гистограмма температурных факторов для структур с атомным и средним разрешением
Эти гистограммы практически не пересекаются. Видно, что для более точной модели температурные факторы значительно меньше, чем для более грубой. Действительно, температурный фактор (он же фактор атомного смещения) показывает, насколько восстановленная электронная плотность широко "размазана" вокруг атома. При высоком разрешении различимы более мелкие детали и вписать атомы в электронную плотность легче, электронная плотность не размывается между атомами. Разумеется, непосредственное значение температурного фактора зависит не только от разрешения, но и от условий и качества эксперимента, от самой структуры (наличия подвижных петель) и так далее. Эти же данные были представлены в виде boxplot, чтобы нагляднее показать различия между температурными факторами атомов структур.
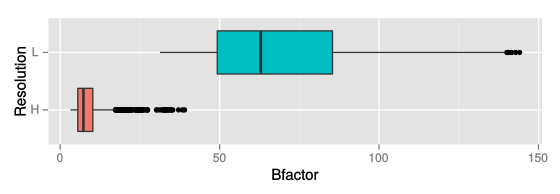
Рис. 16: Boxplot температурных факторов для структур с атомным и средним разрешением
Кроме того, в pdb файле структуры высокого разрешения для каждого атмоа представлен его анизотропный температурный фактор (и других структурах тоже), а в структурах среднего разрешения найти такие сведения можно намного реже.
Коэффициент заполнения
Аналогично были получены коэффициенты заполнения всех атомов структур и построена соответствующая гистограмма (рис. 17).
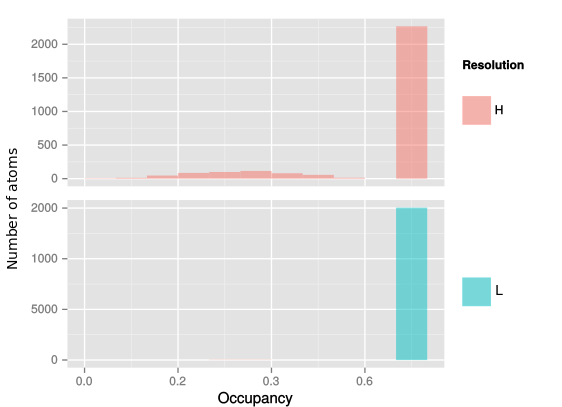
Рис. 17: Гистограммы коэффициентов заполнения для структур с атомным и средним разрешением
Большая часть атомов имеют коэффициент заполнения, равный 1. Однако в структуре с высоким разрешением присутствует некоторое количество атомов и с меньшим коэффициентом заполнения. Аналогчная информация представлена на рис. 18.
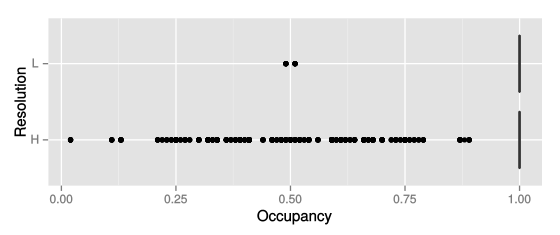
Рис. 18: Boxplot коэффициентов заполнения для структур с атомным и средним разрешением
Однако атомов с коэффициентом заполнения 1 так много, что исключения видны на графиках как выбросы. Кроме того, неясно, зависит ли количество атомов с коэффициентом заполнения меньше единицы от разрешения, или это просто такая структура попалась. Чтобы проверить гипотезу, были проанализированы еще 8 структур (4 с разрешением меньше 1.0 и 4 с разрешением от 2.0 до 2.5) с различным количеством атомов, и проанализирован процент атомов с коэффициентом заполнения меньше единицы (рис. 19).
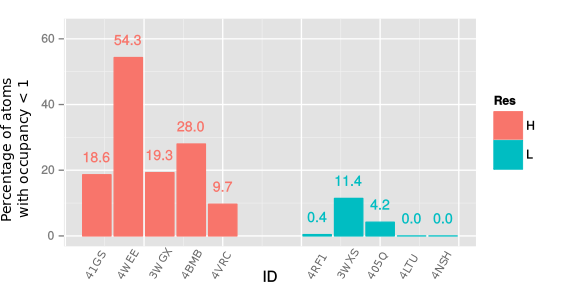
Рис. 19: Процент атомов с низким коэффициентом заполнения для структур с атомным и средним разрешением
В структурах среднего разрешения таких атомов намного меньше, чем в структурых высокого разрешения, в некоторых их нет совсем. Это объясняется тем, что с увеличением точности заметны более мелкие различия в положении атомов, при более низком разрешении же они неразличимы.