Гомологичное моделирование комплекса белка с лигандом
1
В качестве белка для работы был выбран лизоцим тюленя Уэдделла:

С помощью Clustal было построено выравнивание последовательностей выбранного белка и лизоцима форели:
>P1;sp|P11941|LYSC2_ONCMY ------------------KVYDRCELARALKASGMDGYAG NSLPNWVCLSKWESSYNTQATNRNTDG-STDYGIFQINSR YWCDDGRTPGAKNVCGIRCSQLLTDDLTVAIRCAKRVVLD PNGIGAWVAWRLHCQNQDLRSYVAGCGV * >P1;sp|Q659U0|LYSC_LEPWE MKAPLLLGLLLLSVTVQGKVFERCDLARTLKRLGLAGFKG VSLANWMCLAKWESDYNTKATNYNPGSRSTDYGIFQINSR YWCNDGKTPRAVNSCHIPCSDLLKDDITQAVACAKRVVSD PNGIRAWVAWRAHCENQDVSQYVRNCGV *
2..3
Файл с выравниванием был модифицирован для modeller:
В файле со структурой были удалены все молекулы воды, а всем остаткам лиганда присвоен один номер и соответствующие индексы:
4..5
Был написан скрипт, который говорит modeller построить пять моделейвыбранного белка с лигандом, сохраняя взаимное расположение трех пар атомов белка и лиганда, образующих водородные связи. В выравнивании мы отделили белок и лиганд слэшем, то есть сказали, что они должны быть на разных цепях. Поэтому присваиваем остаткам лиганда в скрипте цепь B.
from modeller.automodel import *
class mymodel(automodel):
def special_restraints(self, aln):
rsr = self.restraints
for ids in (('OD2:120:A', 'O6A:149:B'),
('ND2:64:A', 'OLC:149:B'),
('OE2:53:A', 'O6C:149:B')):
atoms = [self.atoms[i] for i in ids]
rsr.add(forms.upper_bound(group=physical.upper_distance,
feature=features.distance(*atoms), mean=3.5, stdev=0.1))
env = environ()
env.io.hetatm = True
a = mymodel(env, alnfile='lysozyme.pir', knowns=('1lmp'), sequence='seq')
a.starting_model = 1
a.ending_model = 5
a.make()
После запуска скрипта мы получили пять моделей белка с лигандом.
- lysc_lepwe.py
- seq.B99990001.pdb
- seq.B99990002.pdb
- seq.B99990003.pdb
- seq.B99990004.pdb
- seq.B99990005.pdb
6
Все модели в участке контакта с лигандом очень похожи друг на друга, поэтому визульно оценить из качество трудно. Редко встречаются различия в расположении остатков непосредственно рядом с лигандом, как, например, аспартата-120 (атом OD2 которого образует водородную связь с O6A лиганда, как было прописано в скрипте):
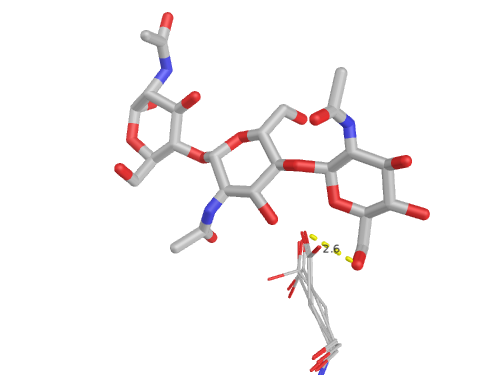
Лиганд и соответствующая водородная связь показаны только один раз, так как взаимодействующие атомы во всех моделях расположены рядом.
7
С помощью WHATIF был проведен общий анализ структур после гомологичного моделирования. По сути, этот анализ сочетает в себе многие другие инструменты, которые можно применить в данном случае, и дает общую оценку. Итоговые результаты представленны в таблице ниже:
| Model 1 | Model 2 | Model 3 | Model 4 | Model 5 | Resolution read from PDB file | -1.000 | -1.000 | -1.000 | -1.000 | -1.000 | 1st generation packing quality | -2.632 | -2.668 | -2.776 | -2.717 | -2.669 | 2nd generation packing quality | -3.037 | -3.031 | -3.297 | -3.039 | -2.806 | Ramachandran plot appearance | -0.232 | -0.087 | -0.404 | -0.265 | -0.279 | chi-1/chi-2 rotamer normality | -1.988 | -2.996 | -1.939 | -2.367 | -2.438 | Backbone conformation | -0.572 | -0.677 | -1.304 | -0.635 | -0.760 | Inside/Outside distribution | 1.068 | 1.034 | 1.045 | 1.035 | 1.059 |
|---|
Согласно этим результатам, все модели получились не особо хорошими, и однозначно выбрать среди них одну я не могу.