Сигналы и мотивы II
Для выполнения задания был выбран вариант 5. В Excel было посчитано количество каждого нуклеотида в i-ой позиции, рассчитана встречаемость для каждого нуклеотида в каждой позиции и далее по формуле IC(b, j) = f(b,j) * log2(f(b,j)/p(b)), где p(b)-базовые частоты встречаемости, было посчитано информационное содержание. Для каждого столбца я вычислила IC(j). Суммарное IC выравнивания составило 11,919. Файл Excel со всеми вычислениями можно скачать здесь, а финальная таблица изображена на рисунке 1.
Также выравнивание было загружено на сайт http://weblogo.berkeley.edu/logo.cgi для построения logo. Результат можно увидеть на рисунке 2.
Задание 2. Проверка PWM для сайтов регуляции разрывной транскрипции sgmRNA.
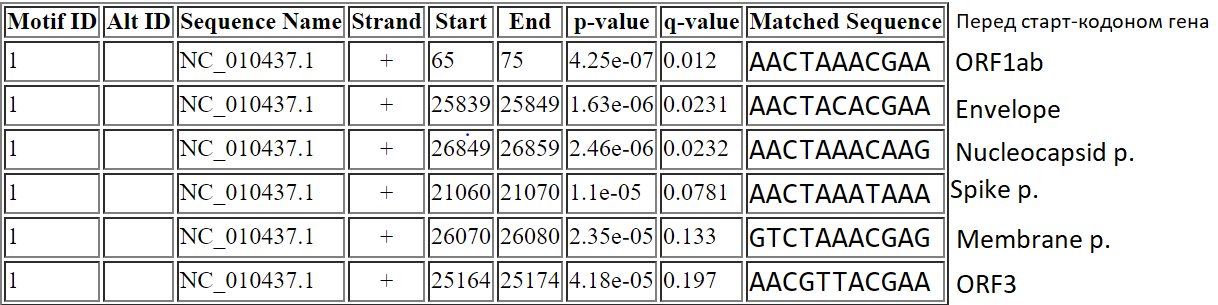
С помощью программф Fimo было получено шесть находок сигнала, заданного с помощью PWM. Результат программы можно увидеть на рисунке 3. Мотив обладает следующими свойствами:
1)Его длина 11 нуклеотидов, но первые шесть нуклеотидов составляют самую консервативную часть. 2)Ровно один сигнал есть в лидерной последовательности, до начала гена ORF1ab полипротеина, как и перед каждым поздним геном (обычно сигнал располагается недалеко от старт-кодона). 3)Нет других сигналов, соответствующих мотиву. 4)P-value этих находок малы(<0.001), поэтому можно утверждать, что они не случайны.
Исходя из всего вышесказанного, можно сделать вывод, что такой мотив близок к идеальному.
Построение LOGO для последовательностей Козак.
Для каждого позднего гена и гена ORF1ab был взят страт-кодон с пятью нуклеотидами до и после него. Как видно по построенному LOGO в этих участках нет сильно консервативных позиций (более менее консервативны 1 и 3 позиция), что может быть связано либо с небольшим количеством генов, кодирующих белки, либо с невысокой консервативностью этого участка. Стоит также отметить, что для последовательностей Козака человека характерен нуклетид в -3 позиции от старт-кодона, что также характерно и для данного коронавируса. Неподалеку от старт-кодонов нескольких поздних генов были найдены участки, содержащие atg, в одной из них на -3 позиции стоял аденин, но на -5 не было цитозина.
Поиск сигнала в близкородственных видах.
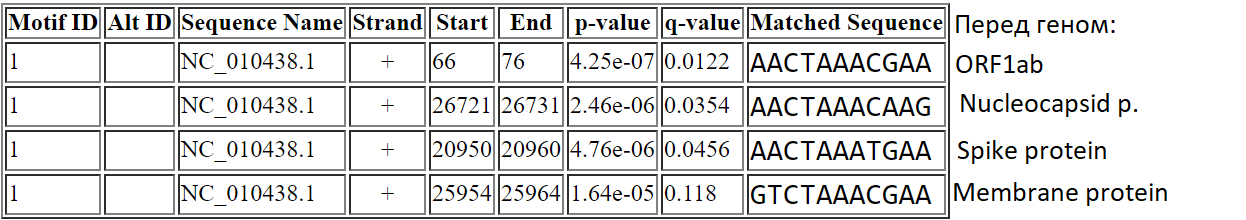
Для определния специфичности сигнала был выбран близкородственный вид Miniopterus pusillus bat coronavirus HKU8-related, который содержит восемь генов, кодирующих белки и вирус из того же рода Porcine epidemic diarrhea virus, содержащий шесть белков. Для каждого из них был проведен поиск сигнала, заданного PWM программой Fimo. Для первого коронавируса программа нашла только четыре сигнала, но все они находятся близко от старт-кодонов определенных генов и первые шесть нуклеотидов почти полностью соответствуют сигналу. Для второго коронавируса находок оказалось только 2. Трудно делать вывод, насколько данный мотив специфичен, но он распространен по крайней мере между близкородственными видами.
Скачать геном Miniopterus pusillus bat coronavirus HKU8-related