BLAST
Поиск гипотетических гомологов изучаемого белка в разных банках
BLAST (сокращение от Basic Local Alignment Search Tool) - программа, осуществляющая поиск участков локального сходства между заданной последовательностью и всеми последовательностями определённого банка данных. Использует последовательности как белковые, так и нуклеотидные, может быть использована для выяснения эволюционных связей между белками, поиска функционально схожих участков в неродственных белках и общей классификации генов в семейства.Поиск BLAST проводился по трём базам данных: Swiss-Prot, PDB (Protein Data Bank) и nr (non-redundunt protein sequences). О первых двух ресурсах упоминалось в предыдущих практикумах, третья же база данных содержит в себе информацию о последовательностях из SwissProt, SwissProt updates, PIR и PDB. Включает большое количество гипотетически существующих последовательностей. При поиске использовалась матрица BLOSUM62, штраф за открытие гэпа составлял 11, а за его продолжение - 2. Варьировались параметры максимального E-value и количества последовательностей в выдаче BLAST.
Таблица 1. Результаты поиска гомологов белка CISY_BACSU с помощью BLAST
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession code | P39119.2 | 2C6X_A | ZP_03590631.1 |
| E-value | 0.0 | 0.0 | 0.0 |
| Вес (в битах) | 807 | 801 | 807 |
| Процент идентичности | 100 | 100 | 100 |
2. Число находок с E-value < 10–10 |
97 | 27 | 6570 |
3. Худшая находка из "удовлетворительных" (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 117 | 27 | 8513 |
| Accession | F4J5S1.1 | 1CSC_A | EKD14678.1 |
| E-value | 0.45 | 2e-12 | 0.99 |
| Вес (в битах) | 36.6 | 70.0 | 40.9 |
| Процент идентичности | 39 | 24 | 23 |
| Процент сходства | 54 | 40 | 45 |
| Длина выравнивания (в а/к остатках) | 125 | 319 | 125 |
| Координаты выравнивания | запрос: 112-232 находка: 1266-1385 |
запрос: 47-341 находка: 94-410 |
запрос: 166-287 находка: 437-561 |
| Число гэпов | 3/51(5%) | 26/319(8%) | 3/125(2%) |
- Во всех трёх запросах первой последовательностью найден рассматриваемый белок, наличествует 3D-структура.
- Число явных гомологов (E-value < 1e-10) сильно различно, но отвечает соотношению PDB < Swiss-Prot < nr, что связано с различиями в объёме баз данных, которые содержат последовательности. NR содержит последовательности, реальное существование соответствующих белков для которых не показано, что увеличивает число найденных последовательностей.
- Во всех поисковых запросах число последовательностей превышает заданное по умолчанию число ответов выдачи.
Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
В этом разделе осуществляется поиск гомологов белка (только по банку Swiss-Prot) в таксонах различного ранга от другого доминиона (Eukaryota) до другого вида того же рода (Bacillus anthracis). Задачей является нахождение приемлемого гомолога (E-value < 0.001) в группе по возможности наиболее удалённой от B. subtilis. Результаты поиска и характеристики находок находятся в таблице 2. 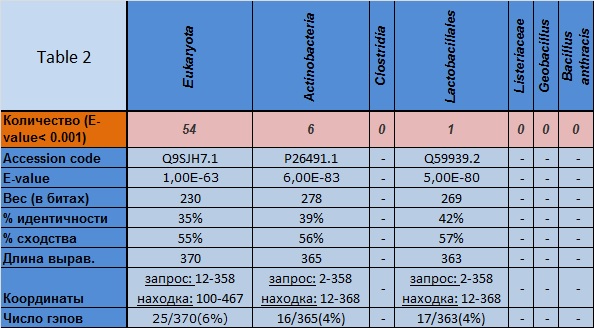 |
Таблица 2. Результаты поиска гомологов в различных группах организмов по банку Swiss-Prot и характеристики находок |
Поиск не дал результатов в четырёх запросах из семи (см. Табл. 2). Три оставшихся запроса выдали записи гомологов, характеристики выравниваний которых указывают на высокое сходство последовательностей с заданной. Кроме того, что неудивительно, количество найденных гомологов уменьшается с уменьшением ранга, а следовательно, и объёма таксона. Наиболее сходным по последовательности гомологом, который принадлежит к удалённому таксону, является пероксомальная цитратсинтаза из Arabidopsis thaliana - растения из семейства крестоцветных, классического объекта многих лабораторных исследований.
BLAST двух последовательностей
В этом разделе BLAST использовался как инструмент для выравнивания последовательности исходного белка и гомолога, найденного среди Lactobacillales (см. раздел 2). Этим гомологом оказалась цитратсинтаза из бактерии Streptococcus mutans. Было проведено выравнивание с порогами E-value 10 и 0.01 (см. рисунок 1). Карта локального сходства и другие характеристики выравнивания не изменялись при разных E-value, приводятся характеристики и карта только для одного случая. 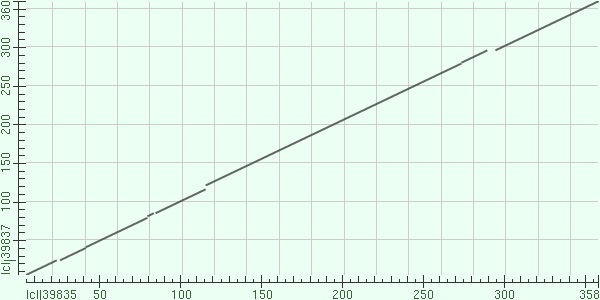 |
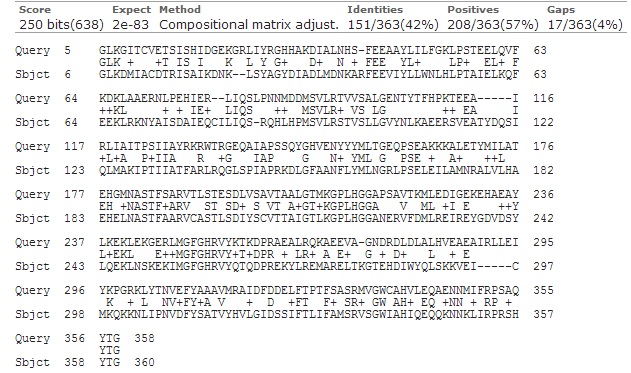 |
| Рис. 1 Карта локального сходства выравниваемых последовательностей. | Рис. 2 Полученное выравнивание белка с его гомологом, характеристики выравнивания. |
Сравнение результатов поиска с использованием различных матриц BLOSUM
В этом разделе мы выясним, как влияет порог кластеризации используемой матрицы на результаты поиска гомологов рассматриваемого белка. Проведём поиск наиболее отдалённого гомолога из Eukaryota с использование матрицы BLOSUM45 и BLOSUM90, затем сравним результаты с уже проведённым поиском с матрицей BLOSUM62.Таблица 3. Сравнение результатов поиска BLAST с использованием различных матриц замен
| Используемая матрица | BLOSUM 62 | BLOSUM 90 | BLOSUM 45 |
| Номер находки в списке описаний | 1 | 1 | 1 |
| Accession | Q9SJH7.1 | Q9SJH7.1 | Q9SJH7.1 |
| E-value | 1E-63 | 5E-61 | 5E-58 |
| Вес (в битах) | 228 | 223 | 197 |
| % идентичности | 35 | 36 | 35 |
| % сходства | 55 | 49 | 56 |
| Длина выравнивания | 370 | 374 | 370 |
| Число гэпов | 25/370(6%) | 33/374(8%) | 25/370(6%) |
В целом, большинство позиций сравнения между тремя матрицами имеют сходные значения, но можно отметить, что из-за замены матрицы происходят изменения в весе выравнивания, что влечёт за собой изменение E-value, identity и similarity. Кроме того, BLAST по умолчанию использует для различных матриц разные штрафы за открытие и продление гэпа, что сказывается на длине выравнивания и количестве пропусков.
Сравнение различных интерфейсов программы BLAST
В этом разделе сравнивается удобство использование программы BLAST, которая доступна не только с сервера NCBI, но и с серверов EMBL-EBI и UniProt. Результаты сравнения представлены в таблице 4.Таблица 4. Сравнение различных интерфейсов для использования BLAST
| Параметры срапвнения | сервер NCBI | сервер EMBL-EBI | сервер Uniprot |
| Простота использования | сложно, много на первый раз непонятных полей | интерфейс проще, введена пошаговая схема, что плюс | интерфейс предельно упрощён |
| Количество и разнообразие БД | БД много, представлены разнообразные по смыслу | БД много, во многих есть выбор подразделов | БД мало, в основном, связанные с Uniprot |
| Количество матриц | 8 | 8 | 5 |
| Точность выбора систематической группы | предельно удобный и точный инструмент выбора | выбор в рамках крупных таксонов в названиях БД | выбор в рамках крупных таксонов в названиях БД |
| Наглядность представления результата | результаты расположены раскрывающимся списком, что неудобно | результаты удобно раcположены по различным смысловым вкладкам | просто ориентироваться, немного различных групп результатов |
Итак, хочется отметить, что сервер Uniprot удобен для неспециализированного использования, даже при незнании глубинных вопросов, связанных с BLAST. Интерфейс крайне "user-friendly", что упрощает и ускоряет работу. Многие опции автоматизированы. Что касается двух оставшихся серверов, то используемые там интерфейсы хороши, если требуется точно настроенный поиск, затрагивающий иногда изменения в самом алгоритме поиска. Сервер EMBL-EBI порадовал большим количеством информации по анализу полученных результатов, в том числе, например, наличием вкладки Functional Predictions, где показаны данные по поиску мотивов, доменов и других характерных для последовательности элементов.
Дата последнего обновления: 17.04.2013
© Dmitry Travin, 2012