PSI-BLAST (Position-Specific Iterated BLAST)
Эта страничка посвящена работе с программой PSI-BLAST (Position-Specific Iterated BLAST) - вариантом BLAST, при котором найденные гомологи можно использовать для следующего раунда поиска путём составления специальных матриц PSSM (Position-Specific Scoring Matrix), что позволяет проводить поиск гомологов более специфично для конкретной группы белков (исходного белка и хорошо с ним сопоставимых и выравниваемых гомологов). Это достигается тем, что при составлении подобного рода матриц учитывается вес аминокислоты в конкретной позиции множественного локального выравнивания. Для новой же последовательности с помощью матрицы рассчитывается степень сходства с уже имеющимися.
Алгоритм работы PSI-BLAST:
Таблица 1. Характеристики результатов PSI-BLAST после каждой итерации
При рассмотрении таблицы 1 заметно, что происходит стабилизация получаемого результата при прохождении нескольких итераций. По результатам поиска выборка гомологов составит для данного белка 11 штук, включая его самого. Белок номер 12 из выдачи PSI-BLAST очевидно негомологичен предыдущим и попал в выборку случайно. Перед каждым новым поиском я не учитывал его в составлении профиля, но при этом он остался в "хороших" результатах. Его негомологичность легко видна, так как. в отличие от других гомологов, принадлежащих различным млекопитающим, этот белок имеет бактериальное происхождение, а кроме того, различие в E-value между ним и предпоследним в списке "хороших" составляет 33 порядка. Такое различие в E-value показывает, что выборка гомологов обособлена достаточно хорошо.
Гомология среди 11-ти представителей полученной выборки подтверждается и выравниванием, построенным с помощью MUSCLE (см. рис. 1).
Все последовательности, найденные при поиске через PSI-BLAST, присутствуют в представленном выравнивании и содержат домен Bclt. Никаких других доменов ни в одной из них не обнаружено, лишь в одной из Callithrix jacchus (обыкновенная игрунка) присутствует домен Bcl2, но в дублированном виде: один домен представлен почти полностью, в то время как от второго осталась только половина.
Таким образом, изучение доменного строения в очередной раз доказывает правомерность объединения найденных белков в группу близких гомологов.
Алгоритм работы PSI-BLAST:
- Обычный (белок-белковый BLAST), основные выбираемые пользователем параметры на этом шаге - ограничение по поиску в определённых таксонах, задание порога E-value и выбор одной из баз данных.
- Получены "хорошие" (выше E-value) и "плохие"(ниже) последовательности.
- Пользователь выбирает по своему усмотрению гомологи, которые будут использованы для составления PSSM для следующего раунда поиска.
- Это продолжается до тех пор, пока не будут появляться в выдаче "хороших" новые результаты, и разрыв в E-value между худшей "хорошей" и лучшей "плохой" находками не увеличится достаточно - то есть происходит стабилизация результатов и обособление группы гомологов.
Формирование семейства гомологов для данной последовательности
Из предложенного списка была выбрана последовательность с ID Q1AHR3, которая кодирует структуру Bcl-2-подобного белка из шимпанзе (Pan troglodytes). В таблице 1 представлены некоторые параметры, характеризующие полученные списки находок PSI-BLAST после каждой из идущих друг за другом итераций.Таблица 1. Характеристики результатов PSI-BLAST после каждой итерации
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 11 | XP_003476407.1 | 2e-14 | ZP_09360138.1 | 6.1 |
| 2 | 12 | NP_302678.1 | 0.005 | YP_005335608.1 | 0.005 |
| 3 | 12 | NP_302678.1 | 8e-04 | XP_003003251.1 | 0.041 |
| 4 | 12 | NP_302678.1 | 0.001 | XP_003003251.1 | 0.043 |
| 5 | 12 | NP_302678.1 | 0.001 | XP_003003251.1 | 0.048 |
При рассмотрении таблицы 1 заметно, что происходит стабилизация получаемого результата при прохождении нескольких итераций. По результатам поиска выборка гомологов составит для данного белка 11 штук, включая его самого. Белок номер 12 из выдачи PSI-BLAST очевидно негомологичен предыдущим и попал в выборку случайно. Перед каждым новым поиском я не учитывал его в составлении профиля, но при этом он остался в "хороших" результатах. Его негомологичность легко видна, так как. в отличие от других гомологов, принадлежащих различным млекопитающим, этот белок имеет бактериальное происхождение, а кроме того, различие в E-value между ним и предпоследним в списке "хороших" составляет 33 порядка. Такое различие в E-value показывает, что выборка гомологов обособлена достаточно хорошо.
Гомология среди 11-ти представителей полученной выборки подтверждается и выравниванием, построенным с помощью MUSCLE (см. рис. 1).
 |
| Рис. 1 Множественное выравнивание полученной выборки гомологов |
Сравните выравнивание семейства домена с полученным выравниванием
Произведём поиск по Pfam для выявления эволюционных доменов, присутствующих в найденной выборке гомологов. Поиск по последовательности исходного белка шимпанзе выявил единственный домен - Bclt (Putative Bcl-2 like protein of testis), я просмотрел выравнивание последовательностей, в которых встречен этот домен - оно представлено на рисунке 2. Всего же таких нашлось 21 в том или ином варианте архитектуры.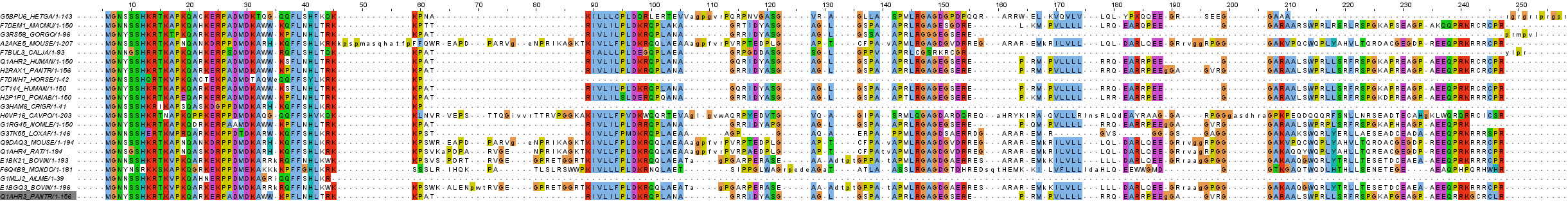 |
| Рис. 2 Множественное выравнивание белков, содержащих домен Bclt (по данным Pfam) |
Все последовательности, найденные при поиске через PSI-BLAST, присутствуют в представленном выравнивании и содержат домен Bclt. Никаких других доменов ни в одной из них не обнаружено, лишь в одной из Callithrix jacchus (обыкновенная игрунка) присутствует домен Bcl2, но в дублированном виде: один домен представлен почти полностью, в то время как от второго осталась только половина.

Таким образом, изучение доменного строения в очередной раз доказывает правомерность объединения найденных белков в группу близких гомологов.
Дата последнего обновления: 27.05.2013
© Dmitry Travin, 2012